Inducibile T7 RNA Polymerase-mediato sistema di espressione multigene, pMGX
Summary
Questo studio descrive metodi per la coespressione mediata da T7 di geni multipli da un singolo plasmide in Escherichia coli usando il sistema plasmid pMGX.
Abstract
La coespressione di molteplici proteine è sempre più essenziale per la biologia sintetica, studiando complessi proteico-proteici e caratterizzando e sfruttando percorsi biosintetici. In questo manoscritto è descritto l'uso di un sistema altamente efficace per la costruzione di operoni sintetici multigene sotto il controllo di una polimerasi RNA T7 indottabile. Questo sistema permette che molti geni siano espressi simultaneamente da un plasmide. In questo caso, un insieme di quattro vettori correlati, pMGX-A, pMGX-hisA, pMGX-K e pMGX-hisK, con il marker selettivo di resistenza a ampicillina o kanamycina (A e K) e posseggono o mancano di una esahistidina N-terminale (Il suo) sono divulgati. Sono forniti protocolli dettagliati per la costruzione di operoni sintetici che utilizzano questo sistema vettoriale, insieme ai dati corrispondenti, che dimostrano che un sistema basato su pMGX contenente cinque geni può essere facilmente costruito e utilizzato per produrre tutte e cinque le proteine codificate in Escherichia coli . Questo sistemaEm e il protocollo consentono ai ricercatori di esprimere in modo frequente moduli multipli complessi e percorsi in E. coli .
Introduction
La coespressione di molteplici proteine è sempre più essenziale, in particolare nelle applicazioni di biologia sintetica, in cui devono essere espressi più moduli funzionali 1 ; Nello studio dei complessi proteico-proteici, in cui espressione e funzione richiedono spesso co-espressione 2 , 3 ; E nel caratterizzare e sfruttare percorsi biosintetici, in cui ogni gene nel percorso deve essere espresso 4 , 5 , 6 , 7 , 8 . Sono stati sviluppati diversi sistemi per la coespressione, in particolare nell'organismo ospitante Escherichia coli , il cavallo da lavoro per l'espressione di proteine ricombinanti di laboratorio 9 . Ad esempio, plasmidi multipli con differenti marcatori selectable possono essere utilizzati per esprimere singole proteine usando una ricchezza di diversi es Vettori di pressione 10 , 11 . I singoli sistemi plasmidi per l'espressione multipla di proteine hanno utilizzato sia promotori multipli per controllare l'espressione di ciascun gene 10 , 12 ; Operoni sintetici, in cui i geni multipli sono codificati su una singola trascrizione 2 , 13 ; O, in alcuni casi, un singolo gene che codifica un polipeptide che viene ultimamente elaborato in modo proteolitico, fornendo le proteine desiderate di interesse 14 .
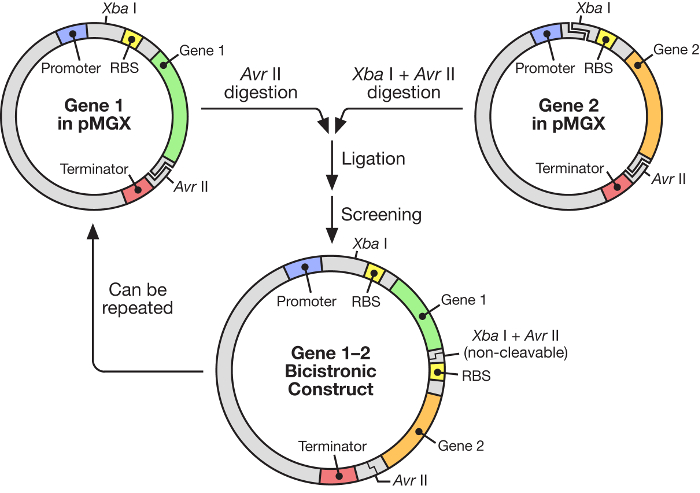
Figura 1: flusso di lavoro pMGX che mostra la costruzione di un vettore policistronico. Il sistema pMGX fornisce una strategia flessibile e facile da usare per la costruzione di operoni sintetici sotto il controllo di un promotore T7 induttivo.E.com/files/ftp_upload/55187/55187fig1large.jpg "target =" _ blank "> Fare clic qui per visualizzare una versione più grande di questa figura.
In questo manoscritto è descritto l'uso di un sistema altamente efficace per la costruzione di operoni sintetici multigene sotto il controllo di una polimerasi RNA T7 indottabile ( figura 1 ). Questo sistema permette che molti geni siano espressi simultaneamente da un plasmide. È basato su un sistema plasmidico, originariamente chiamato pKH22, che è stato utilizzato con successo per una serie di applicazioni diverse 6 , 7 , 8 . Qui, questo set di plasmidi viene ampliato per includere quattro vettori correlati: pMGX-A, un vettore di espressione privo di qualsiasi etichetta C- o N-terminale e con il marker di resistenza dell'ampicillina; PMGX-hisA, un vettore di espressione che codifica un tag di esahistidina N-terminale e con il marker di resistenza dell'ampicillina; PMGX-K, un vettore di espressione lAcking qualsiasi etichetta C- o N-terminale e con il marker di resistenza alla kanamycina; E pMGX-hisK, un vettore di espressione che codifica un tag di hexahistidine N-terminale e con il marker di resistenza alla kanamycina. In questo studio è stato dimostrato il metodo per la generazione di un vettore polistronicico contenente cinque geni usando il sistema pMGX, in particolare pMGX-A, insieme alla produzione di ogni singola proteina in Escherichia coli .
Protocol
Representative Results
Discussion
La coespressione di geni multipli è sempre più essenziale, in particolare per caratterizzare e ricostituire complessi percorsi metabolici multigene 3 , 4 , 5 . Il sistema pMGX rende coespressione multigene in routine E. coli 6 , 7 , 8 e accessibile a diversi ricercatori. In questo studio sono state dimostrate che cinque prot…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questo lavoro è stato sostenuto dal Consiglio di Ricerca delle Scienze Naturali e Ingegneria del Canada.
Materials
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50 x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm X 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box – PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
References
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -. J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998 (2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp Available from: https://www.jove.com/science-education/5074/molecular-cloning (2016)
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp Available from: https://www.jove.com/science-education/5065/the-western-blot (2016)
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).

