4.4:
A Constante de Ligação de Equilíbrio e a Força de Ligação
4.4:
A Constante de Ligação de Equilíbrio e a Força de Ligação
A constante de ligação de equilíbrio (Kb) quantifica a força de uma interação proteína-ligando. Kb pode ser calculado da seguinte forma quando a reação está em equilíbrio:
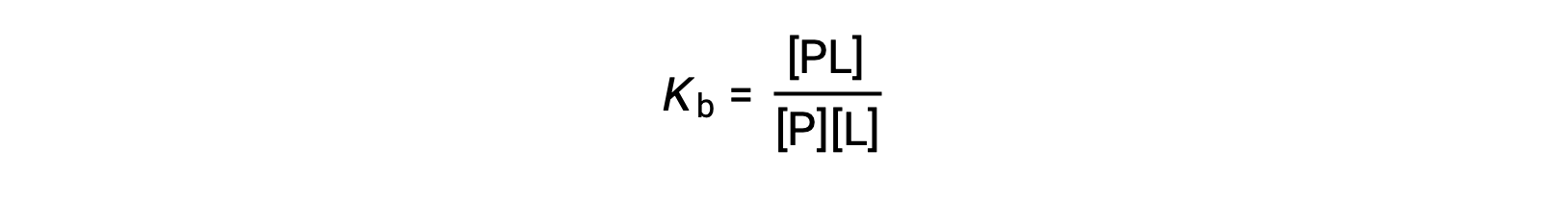
onde P e L são a proteína não ligada e o ligando, respectivamente, e PL é o complexo proteína-ligando.
Como a quantidade de ligando ligado também está relacionada com a taxa de ligação, experiências também podem determinar Kb examinando as taxas de associação (kon) e dissociação (koff) de proteína-ligando usando a seguinte proporção:
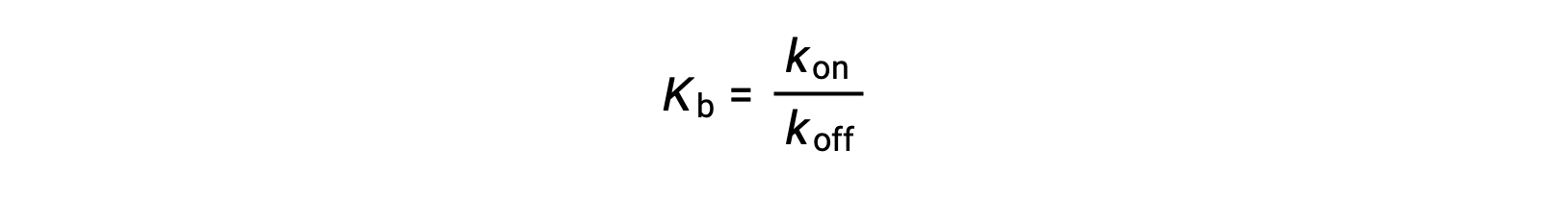
Assim, duas categorias de ensaios de ligação são utilizadas para determinar a constante de ligação de equilíbrio – as que medem as concentrações em equilíbrio e as que medem a cinética de uma reacção. No caso, a reação deve estar em equilíbrio no momento da medição.
O método de determinação das concentrações de equilíbrio depende da sensibilidade desejada e da facilidade de deteção do sinal. Por estas razões, os ensaios espectroscópicos são mais amplamente utilizados. Nestas experiências, a reação produz uma alteração de absorvância de um reagente ou de um produto em um determinado comprimento de onda, detectável por um espectrofotómetro UV-Vis. Alternativamente, o reagente ou produto pode ser marcado com uma sonda fluorescente ou pode conter um fluoróforo intrínseco. Em seguida, o progresso da reação pode ser medido a partir da alteração da fluorescência. Estes ensaios são realizados variando as concentrações de um reagente, enquanto que a restante experiência é mantida constante. Os resultados podem então ser colocados em gráfico e analisados por vários métodos de ajuste de curvas.
Interações entre proteínas e ligandos também são estudadas utilizando uma variedade de técnicas bioquímicas e espectroscópicas. A análise estrutural, utilizando a cristalografia de raios X e a espectroscopia NMR, auxilia na previsão das interações proteína-ligando através de simulações moleculares. Abordagens teóricas e computacionais, como estudos de ligação proteína-ligando, são usadas extensivamente para caracterizar a posição e interações de moléculas pequenas de ligandos, incluindo candidatos a fármacos. O desenvolvimento de fármacos auxiliado por computador é uma alternativa rápida e de baixo custo para acelerar o ritmo da testagem de fármacos convencional de tentativa e erro.
Suggested Reading
- Pollard, Thomas D. "A guide to simple and informative binding assays." Molecular biology of the cell 21, no. 23 (2010): 4061-4067.
- Hulme, Edward C., and Mike A. Trevethick. "Ligand binding assays at equilibrium: validation and interpretation." British journal of pharmacology 161, no. 6 (2010): 1219-1237.
- Medina-Franco, José L., Oscar Méndez-Lucio, and Karina Martinez-Mayorga. "The interplay between molecular modeling and chemoinformatics to characterize protein–ligand and protein–protein interactions landscapes for drug discovery." In Advances in protein chemistry and structural biology, vol. 96, pp. 1-37. Academic Press, 2014.
