Mesurer les propriétés mécaniques des cellules vivantes par microscopie à force atomique
Summary
Cet article démontre un protocole pour caractériser les propriétés mécaniques des cellules vivantes au moyen de microindentation aide d'un microscope à force atomique (AFM).
Abstract
Les propriétés mécaniques des cellules et la matrice extracellulaire (MEC) jouent un rôle important dans de nombreux processus biologiques, y compris la différenciation des cellules souches, la formation de tumeurs, et la cicatrisation des plaies. Les changements de raideur des cellules et ECM sont souvent des signes de changements dans la physiologie ou de maladies dans les tissus cellulaires. Par conséquent, la rigidité de la cellule est un indice pour évaluer l'état de cultures de cellules. Parmi la multitude de méthodes utilisées pour mesurer la rigidité des tissus et cellules, micro-indentation en utilisant un microscope à force atomique (AFM) fournit un moyen de mesurer de façon fiable la rigidité des cellules vivantes. Cette méthode a été largement appliquée pour caractériser la rigidité micro-échelle pour une variété de matériaux allant des surfaces métalliques dans les tissus et les cellules biologiques mous. Le principe de base de cette méthode est de mettre en retrait une cellule avec une pointe AFM de la géométrie sélectionnée et mesurer la force appliquée à partir de la flexion du cantilever AFM. Montage de la courbe force-indentation sur le mode Hertzl pour la géométrie de la pointe correspondante peut donner des mesures quantitatives de la rigidité du matériau. Cet article démontre la procédure pour caractériser la rigidité des cellules vivantes à l'aide de l'AFM. Les étapes clés, y compris le processus de calibration AFM, l'acquisition de force courbe, et l'analyse de données en utilisant une routine MATLAB sont démontrés. Les limites de cette méthode sont également discutés.
Introduction
Propriétés mécaniques, notamment la raideur, des cellules individuelles et leurs matrices extracellulaires environnantes (ECM) sont essentielles pour de nombreux processus biologiques, y compris la croissance cellulaire, la motilité, la division, la différenciation et l'homéostasie tissulaire. 1 Il a été démontré que les cellules rigidité mécanique est principalement déterminée par le cytosquelette, en particulier les réseaux de filaments d'actine et intermédiaires et d'autres protéines associées. 2 Les résultats des essais mécaniques sur des réseaux in vitro des filaments d'actine et intermédiaires suggèrent que les mécanismes cellulaires est largement dépendante de la structure du cytosquelette et la précontrainte dans le cytosquelette. 3-5 Rigidité de cellules vivantes est alors considéré comme un indice pour évaluer la structure du cytosquelette 6, l'activité myosine 7 et de nombreux autres processus cellulaires. Plus important encore, les changements dans les propriétés mécaniques cellulaires sont également souvent considérés comme étroitement associésATED avec diverses maladies telles que la formation de tumeurs et des métastases 8-10 Contrôle de la rigidité mécanique des cellules vivantes peut donc fournir un nouveau moyen de contrôler la physiologie cellulaire;. pour détecter et diagnostiquer les maladies 8;. et d'évaluer l'efficacité des traitements médicamenteux 11 , 12
Plusieurs méthodes, y compris microrheologie de traces de particules, 13-16 cytométrie de torsion magnétique, 17 micropipette aspiration 18,19 et microindentation 20-22 ont été développés pour mesurer l'élasticité des cellules. microrheologie de suivi des particules retrace les vibrations thermiques soit des particules fluorescentes submicroniques injectés dans des cellules ou des marqueurs fiduciaires à l'intérieur du cytosquelette de la cellule. 23 propriétés élastiques et visqueuses de cellules sont calculés à partir des déplacements des particules mesurées en utilisant le théorème de fluctuation-dissipation. 14,23 Cette méthode permet mesures simultanées de locauxpropriétés mécaniques à haute résolution spatiale à différents endroits dans une cellule. Cependant, l'injection de particules fluorescentes dans les cellules peut entraîner des changements dans la fonction cellulaire, la structure du cytosquelette, et donc les mécaniciens cellulaires. Procédé d'aspiration par micropipette applique une pression négative dans la micropipette de diamètre allant de 1 à 5 um d'aspirer un petit morceau de membrane de la cellule dans la pipette. la rigidité de la cellule est calculée à partir de la pression négative appliquée et de la déformation de la membrane cellulaire. 18 Cette méthode, cependant, ne peut pas détecter la distribution hétérogène de la rigidité à travers la cellule. La cytométrie en torsion magnétique (MTC) s'applique champ magnétique pour générer un couple sur des billes paramagnétiques super-attachés à la membrane cellulaire. 17 raideur cellulaire est dérivée de cette méthode à partir de la relation entre le couple appliqué et de la déformation de torsion de la membrane cellulaire. Il est difficile de contrôler l'emplacement des billes magnétiques dans la méthode MTC, et il est également challenging pour caractériser la déformation en torsion avec une grande résolution. Microindentation applique une indentation avec une géométrie bien définie de punch dans la cellule. La force de pénétration et le retrait résultant dans des cellules suivent souvent la prédiction du modèle Hertz. Des modules de Young de cellules peut être calculée à partir des courbes force-indentation en les équipant le modèle Hertz. Cette méthode a été largement appliquée pour tester les propriétés mécaniques des tissus et des cellules en dépit de ses limitations telles que l'incertitude dans la détermination du point de contact, l'applicabilité du modèle Hertz, et le potentiel d'endommager physiquement les cellules. Parmi les nombreux dispositifs pour microindentaion 20, le microscope à force atomique (AFM) est disponible dans le commerce et a été largement appliquée pour caractériser les propriétés mécaniques des cellules et des tissus vivants 21,24-27.
Cet article démontre la procédure d'utilisation d'un asile MFP3D-Bio AFM pour caractériser les mécanismes cellulaires. AFM pas surment fournit une topographie à haute résolution de cellules, mais aussi a été largement appliquée pour caractériser les propriétés mécaniques des cellules des tissus. Le principe de l'AFM indentation est illustrée à la figure 1. Le cantilever AFM se rapproche de la cellule à partir de quelques micromètres ci-dessus; entre en contact avec la cellule; tirets la cellule de sorte que la déviation en porte à faux atteigne une valeur de consigne présélectionnée, et se détache de la cellule. Au cours de ce processus, la déviation du cantilever est enregistrée en fonction de sa position comme indiqué sur la figure 1. Avant de prendre contact avec la cellule, la console se déplace dans le milieu sans aucune déviation apparente. Lorsque l'indentation sur la cellule, les coudes en porte à faux et le signal déviation augmente. Les leviers sont modélisés comme des poutres élastiques de telle sorte que leur déviation est proportionnelle à la force appliquée à la cellule. En réglant la déformation maximale en porte à faux, l'amplitude maximale de la force appliquée à l'échantillon est limitée pour éviter dAmage aux cellules. La portion de la courbe de force du point B au point C de la figure 1, où les tirets de pointe dans la cellule, n'est pas propre au modèle hertz pour extraire la rigidité de la cellule.
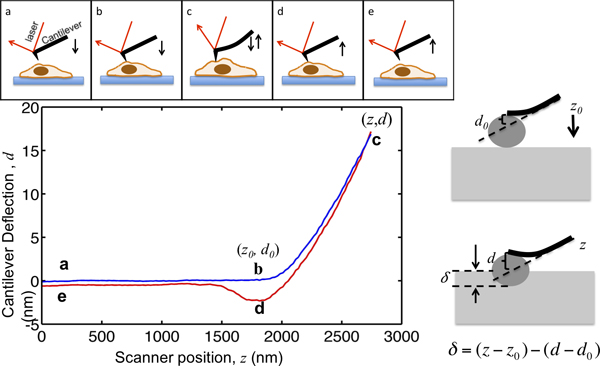
Figure 1. Illustration de l'AFM microindentation et l'interprétation de la courbe de force. Le panneau supérieur montre le mouvement de l'AFM cantilever entraîné par le scanner piezo. La position verticale Z du cantilever et la déviation en porte à faux signal D est enregistrée au cours du processus. Le porte à faux commence à partir du point A, de quelques microns au dessus de la cellule. En se rapprochant de la cellule, l'échantillon indentation δ reste nulle jusqu'à ce qu'il atteigne le point B, où la pointe est en contact avec la cellule. Les coordonnées du point B dans le complot sont des valeurs critiquespour l'analyse de données, notée (z 0, n 0>). De B à C, les tirets en porte à faux dans la cellule jusqu'à ce que la déflection du cantilever atteint un point de consigne, qui est défini comme le rapport entre la force de pénétration maximale ciblée et la constante de ressort cantilever. Une fois le signal de déviation atteint la valeur maximale prédéterminée, le cantilever est alors retiré de la cellule à un point d, où elle souvent être tiré vers le bas en raison de pointe-échantillon adhésion, se détache de la cellule et revient à sa position initiale à e. Le panneau de droite illustre la relation entre le retrait et le z enregistrée et le signal d. Dans la partie inférieure gauche est un tracé d'une courbe représentative de la force, l'indentation maximum d'un porte à faux, dont la constante de ressort est mesurée comme étant de 0.07N / m, est réglé pour être 17 nm de telle sorte que la force maximale indentation appliquée à l'échantillon est de 1,2 nn. Les endroits clés au cours de l'indentation sont marquées.
Protocol
Representative Results
Discussion
La méthode d'indentation AFM a des avantages pour caractériser les propriétés mécaniques des cellules vivantes. Quoique moins sensible que la cytométrie de torsion magnétique et pinces optiques, qui peuvent mesurer des forces sur le plan piconewton 32, l'AFM permet de détecter la force de résistance à partir d'échantillons allant de quelques dizaines de pico-Newton à des centaines de nano-Newton, comparables à aller de force peut être appliqué à des cellules à l'aide d'une…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Les auteurs remercient le Dr Paul Janmey à l'Université de Pennsylvanie pour fournir des lignées cellulaires utilisées dans le présent document. QW reconnaît également JF Byfield et Evan Anderson pour leurs discussions éclairées sur les techniques AFM.
References
- Discher, D. E., Janmey, P., Wang, Y. L. Tissue cells feel and respond to the stiffness of their substrate. Science. 310, 1139-1143 (2005).
- Wagner, O. I., et al. Softness, strength and self-repair in intermediate filament networks. Exp. Cell Res. 313, 2228-2235 (2007).
- Wang, N., et al. Mechanical behavior in living cells consistent with the tensegrity model. Proceedings of the National Academy of Sciences of the United States of America. 98, 7765-7770 (2001).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. American Journal of Physiology. Cell Physiology. 282, 606-616 (2002).
- Kasza, K. E., et al. Filamin A is essential for active cell stiffening but not passive stiffening under external force. Biophysical Journal. 96, 4326-4335 (2009).
- Elson, E. L. Cellular mechanics as an indicator of cytoskeletal structure and function. Annual Review of Biophysics and Biophysical Chemistry. 17, 397-430 (1988).
- Schafer, A., Radmacher, M. Influence of myosin II activity on stiffness of fibroblast cells. Acta Biomaterialia. 1, 273-280 (2005).
- Guck, J., et al. Optical deformability as an inherent cell marker for testing malignant transformation and metastatic competence. Biophysical Journal. 88, 3689-3698 (2005).
- Cross, S. E., Jin, Y. S., Rao, J., Gimzewski, J. K. Nanomechanical analysis of cells from cancer patients. Nature Nanotechnology. 2, 780-783 (2007).
- Plodinec, M., et al. The nanomechanical signature of breast cancer. Nature Nanotechnology. 7, 757-765 (2012).
- Rotsch, C., Radmacher, M. Drug-induced changes of cytoskeletal structure and mechanics in fibroblasts: an atomic force microscopy study. Biophysical Journal. 78 (00), 520-535 (2000).
- Cross, S. E., Jin, Y. S., Lu, Q. Y., Rao, J., Gimzewski, J. K. Green tea extract selectively targets nanomechanics of live metastatic cancer cells. Nanotechnology. 22, 215101 (2011).
- Hoffman, B. D., Crocker, J. C. Cell mechanics: dissecting the physical responses of cells to force. Annual Review of Biomedical Engineering. 11, 259-288 (2009).
- Hoffman, B. D., Massiera, G., Van Citters, K. M., Crocker, J. C. The consensus mechanics of cultured mammalian cells. Proceedings of the National Academy of Sciences of the United States of America. 103, 10259-10264 (2006).
- Lau, A. W., Hoffman, B. D., Davies, A., Crocker, J. C., Lubensky, T. C. Microrheology, stress fluctuations, and active behavior of living cells. Physical Review Letters. 91, 198101 (1981).
- Liu, J., et al. Microrheology probes length scale dependent rheology. Physical Review Letters. 96, 118104 (2006).
- Deng, L., et al. Fast and slow dynamics of the cytoskeleton. Nature Materials. 5, 636-640 (2006).
- Oh, M. J., Kuhr, F., Byfield, F., Levitan, I. Micropipette Aspiration of Substrate-attached Cells to Estimate Cell Stiffness. J. Vis. Exp. (67), e3886 (2012).
- Hochmuth, R. M. Micropipette aspiration of living cells. Journal of Biomechanics. 33, 15-22 (2000).
- Levental, I., et al. A simple indentation device for measuring micrometer-scale tissue stiffness. J. Phys-Condens. Mat. 22, (2010).
- Mahaffy, R. E., Park, S., Gerde, E., Kas, J., Shih, C. K. Quantitative analysis of the viscoelastic properties of thin regions of fibroblasts using atomic force microscopy. Biophysical Journal. 86, 1777-1793 (2004).
- Radmacher, M. Measuring the elastic properties of biological samples with the AFM. IEEE Engineering in Medicine and Biology Magazine: The Quarterly Magazine of the Engineering in Medicine & Biology Society. 16, 47-57 (1997).
- Crocker, J. C., Hoffman, B. D. Multiple-particle tracking and two-point microrheology in cells. Methods in Cell Biology. 83, 141-178 (2007).
- Liu, F., Tschumperlin, D. J. Micro-mechanical characterization of lung tissue using atomic force microscopy. J. Vis. Exp. (54), e2911 (2011).
- Solon, J., Levental, I., Sengupta, K., Georges, P. C., Janmey, P. A. Fibroblast adaptation and stiffness matching to soft elastic substrates. Biophysical Journal. 93, 4453-4461 (2007).
- Wu, H. W., Kuhn, T., Moy, V. T. Mechanical properties of l929 cells measured by atomic force microscopy: Effects of anticytoskeletal drugs and membrane crosslinking. Scanning. 20, 389-397 (1998).
- Radmacher, M., Fritz, M., Kacher, C. M., Cleveland, J. P., Hansma, P. K. Measuring the viscoelastic properties of human platelets with the atomic force microscope. Biophysical Journal. 70, 556-567 (1996).
- Levy, R., Maaloum, M. Measuring the spring constant of atomic force microscope cantilevers: thermal fluctuations and other methods. Nanotechnology. 13, 33-37 (2002).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis–I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129, 430-440 (2007).
- Davis, J. T., Wen, Q., Janmey, P. A., Otteson, D. C., Foster, W. J. Muller cell expression of genes implicated in proliferative vitreoretinopathy is influenced by substrate elastic modulus. Investigative Ophthalmology & Visual Science. 53, 3014-3019 (2012).
- Engler, A. J., Rehfeldt, F., Sen, S., Discher, D. E. Microtissue elasticity: Measurements by atomic force microscopy and its influence on cell differentiation. Method Cell Biol. 83, 521-545 (2007).
- Lele, T. P., et al. Tools to study cell mechanics and mechanotransduction. Methods in Cell Biology. 83 (07), 443-472 (2007).
- A-Hassan, E., et al. Relative microelastic mapping of living cells by atomic force microscopy. Biophysical Journal. 74, 1564-1578 (1998).
- Costa, K. D., Sim, A. J., Yin, F. C. Non-Hertzian approach to analyzing mechanical properties of endothelial cells probed by atomic force microscopy. Journal of Biomechanical Engineering. 128, 176-184 (2006).
- Xiong, Y., Lee, A. C., Suter, D. M., Lee, G. U. Topography and nanomechanics of live neuronal growth cones analyzed by atomic force microscopy. Biophysical Journal. 96, 5060-5072 (2009).
- Byfield, F. J., et al. Absence of filamin A prevents cells from responding to stiffness gradients on gels coated with collagen but not fibronectin. Biophysical Journal. 96, 5095-5102 (2009).
- Cross, S. E., et al. AFM-based analysis of human metastatic cancer cells. Nanotechnology. 19, 384003 (2008).
- Storm, C., Pastore, J. J., MacKintosh, F. C., Lubensky, T. C., Janmey, P. A. Nonlinear elasticity in biological gels. Nature. 435, 191-194 (2005).
- Wen, Q., Janmey, P. A. Polymer physics of the cytoskeleton. Current Opinion in Solid State & Materials Science. 15, 177-182 (2011).
- Dimitriadis, E. K., Horkay, F., Maresca, J., Kachar, B., Chadwick, R. S. Determination of elastic moduli of thin layers of soft material using the atomic force microscope. Biophysical Journal. 82 (02), 2798-2810 (2002).
- Trache, A., Lim, S. M. Live cell response to mechanical stimulation studied by integrated optical and atomic force microscopy. J. Vis. Exp. (44), e2072 (2010).
- Lim, S. M., Kreipe, B. A., Trzeciakowski, J., Dangott, L., Trache, A. Extracellular matrix effect on RhoA signaling modulation in vascular smooth muscle cells. Exp. Cell Res. 316, 2833-2848 (2010).

