Misurare le proprietà meccaniche del Living cellule utilizzando microscopia a forza atomica
Summary
Questo documento illustra un protocollo per caratterizzare le proprietà meccaniche di cellule viventi mediante micropenetrazione utilizzando un microscopio a forza atomica (AFM).
Abstract
Proprietà meccaniche delle cellule e la matrice extracellulare (ECM) svolgono un ruolo importante in molti processi biologici tra cui la differenziazione di cellule staminali, la formazione del tumore, e la guarigione delle ferite. Cambiamenti nella rigidità delle cellule e ECM sono spesso segni di cambiamenti nella fisiologia o malattie nei tessuti delle cellule. Quindi, rigidità cellula è un indice per valutare lo stato di colture cellulari. Tra la moltitudine di metodi applicati per misurare la rigidità di cellule e tessuti, micro-indentazione usando un microscopio a forza atomica (AFM) fornisce un modo per misurare in modo affidabile la rigidità delle cellule viventi. Questo metodo è stato ampiamente applicato per caratterizzare la rigidezza micro-scala per una varietà di materiali, tra cui superfici metalliche a tessuti biologici molli e cellule. Il principio di base di questo metodo è di rientrare una cella con una punta AFM di geometria selezionata e misurare la forza applicata dalla flessione del cantilever AFM. Montaggio della curva forza-indentazione per la modalità di Hertzl per la corrispondente geometria della punta può dare misure quantitative di rigidezza materiale. Questo documento illustra la procedura per caratterizzare la rigidezza delle cellule viventi mediante AFM. Passaggi chiave, tra cui il processo di AFM calibrazione, acquisizione forza-curva, e l'analisi dei dati utilizzando una routine di MATLAB sono dimostrati. Vengono anche discussi limiti di questo metodo.
Introduction
Proprietà meccaniche, in particolare rigidità, di singole cellule e loro matrici extracellulari circostanti (ECM) sono fondamentali per molti processi biologici che comprendono la crescita cellulare, la motilità, divisione, differenziazione e omeostasi tissutale. 1 È stato dimostrato che cellule rigidità meccanica è determinata principalmente dalla il citoscheletro, specialmente le reti di actina e filamenti intermedi e altre proteine ad essi associati. 2 risultati di prove meccaniche su reti in vitro di actina e filamenti intermedi suggeriscono che la meccanica delle cellule dipende in gran parte la struttura del citoscheletro e la pre-stress in il citoscheletro. 3-5 Rigidità di cellule vive è quindi considerato come un indice per valutare la struttura del citoscheletro 6, 7 attività miosina e molti altri processi cellulari. Ancora più importante, i cambiamenti nelle proprietà meccaniche delle cellule sono spesso trovati ad essere strettamente associatiato con diverse condizioni di malattia come la formazione di tumori e metastasi 8-10 Monitoraggio della rigidità meccanica delle cellule viventi può quindi fornire un nuovo modo di monitorare la fisiologia cellulare,. di rilevare e diagnosticare le malattie 8;. ed a valutare l'efficacia dei trattamenti farmacologici 11 , 12
Metodi multipli comprese microrheology particella-tracking, 13-16 torsione magnetica citometria, 17 micropipetta aspirazione 18,19 e micropenetrazione 20-22 sono stati sviluppati per misurare l'elasticità delle cellule. Particella di monitoraggio microrheology ripercorre le vibrazioni termiche di entrambi particelle fluorescenti inferiori al micron iniettati in cellule o marcatori fiduciali all'interno del citoscheletro delle cellule. 23 proprietà elastiche e viscose di cellule sono calcolati gli spostamenti delle particelle misurate usando il teorema di fluttuazione-dissipazione. 14,23 Questo metodo permette di misurazioni simultanee di localeproprietà meccaniche con alta risoluzione spaziale in luoghi diversi in una cella. Tuttavia, l'iniezione di particelle fluorescenti in cellule può portare a cambiamenti nella funzione cellulare, la struttura del citoscheletro, e quindi la meccanica delle cellule. Il metodo di aspirazione micropipetta applica pressione negativa in una micropipetta di diametro compreso tra 1 e 5 um succhiare un piccolo pezzo di membrana cellulare nella pipetta. Cella rigidezza viene calcolata dalla pressione negativa applicata e deformazione della membrana cellulare. 18 Questo metodo, tuttavia, non può rilevare la distribuzione eterogenea della rigidezza attraverso la cella. Citometria torsione magnetica (MTC) applica campo magnetico per generare coppia sulla super-perline paramagnetiche attaccati alla membrana cellulare. 17 Cella rigidezza è derivato in questo metodo dal rapporto tra la coppia applicata e la deformazione torsione della membrana cellulare. È difficile controllare la posizione di perline magnetiche nel metodo MTC, ed è anche challenging per caratterizzare la deformazione torsione con alta risoluzione. Micropenetrazione applica un penetratore con geometrie ben definite a pugni nella cella. La forza di rientro e il rientro risultato cellule seguono spesso la previsione del modello Hertz. Di Young dei moduli delle celle può essere calcolata dalle curve forza indentazione dotandoli al modello Hertz. Questo metodo è stato ampiamente applicato per testare le proprietà meccaniche del tessuto e di cellule dispetto delle sue limitazioni quali l'incertezza nella determinazione contatto puntiforme, applicabilità del modello Hertz, e il potenziale di danneggiare fisicamente le cellule. Tra i molti dispositivi per microindentaion 20, il microscopio a forza atomica (AFM) è disponibile in commercio ed è stato ampiamente applicato per caratterizzare le proprietà meccaniche di cellule e tessuti viventi 21,24-27.
Questo documento dimostra la procedura di utilizzo di un asilo MFP3D-Bio AFM per la caratterizzazione meccanica delle cellule. AFM non suly fornisce topografia ad alta risoluzione di cellule, ma anche è stato ampiamente applicato per caratterizzare le proprietà meccaniche delle cellule dei tessuti. Il principio della AFM indentazione è illustrato in Figura 1. Il cantilever AFM avvicina alla cella da pochi micrometri sopra; fa contatto con la cellula; trattini la cella in modo che la flessione della leva raggiunge un punto di regolazione preselezionata; e tira via dalla cella. Durante questo processo la flessione della leva viene registrata come funzione della sua posizione come mostrato nella Figura 1. Prima di prendere contatto con la cellula, il cantilever muove nel medio senza alcuna deviazione apparente. Quando rientro in cella, le curve a sbalzo e di aumento del segnale di deflessione. I cantilever sono modellati come travi elastica in modo che la loro deflessione è proporzionale alla forza applicata alla cella. Impostando la massima flessione della leva, la grandezza massima della forza applicata al campione viene limitata per evitare dAMAGE alle cellule. La porzione della curva di forza dal punto B al punto C in figura 1, dove i trattini punta nella cellula, viene adattata al modello hertz per estrarre la rigidità cella.
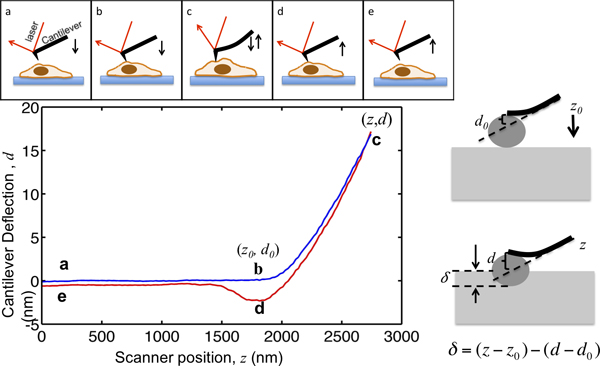
Figura 1. Illustrazione di AFM micropenetrazione e l'interpretazione della curva di forza. Il pannello superiore mostra il movimento del cantilever AFM guidato dallo scanner piezoelettrico. La posizione verticale della mensola z e flessione della leva segnale d viene registrato durante il processo. Il cantilever parte da un punto, di pochi micrometri sopra la cella. Mentre si avvicina la cella, la rientranza δ campione rimane zero fino a raggiungere il punto b, in cui la punta viene a contatto con la cellula. Le coordinate del punto b nel grafico sono valori criticiper l'analisi dei dati, indicato con (z 0, d 0>). Da B a C, i trattini sbalzo nella cella finché flessione della leva raggiunge un punto di regolazione, che è impostato per essere il rapporto tra la forza massima indentazione mirata e la costante elastica cantilever. Una volta che il segnale di deflessione raggiunge il valore massimo prestabilito, il cantilever viene poi ritirato dalla cella a punto d, dove spesso viene tirata verso il basso a causa di punta-campione adesione, stacca dalla cella e ritorna alla sua posizione iniziale a e. Il pannello destro mostra la relazione tra la rientranza e la z registrata e segnale d. In sul pannello di sinistra inferiore è un diagramma di una curva rappresentativa forza, il rientro massimo di un cantilever, la cui costante elastica è misurata essere 0.07N / m, è impostato per essere 17 nm, in modo che la forza massima applicata al rientro campione è 1,2 nN. Le posizioni chiave durante il rientro sono contrassegnati.
Protocol
Representative Results
Discussion
Il metodo di indentazione AFM ha vantaggi per caratterizzare le proprietà meccaniche di cellule viventi. Seppur meno sensibile della citometria a torsione magnetica e pinzette ottiche, in grado di misurare le forze a livello piconewton 32, l'AFM in grado di rilevare la forza di resistenza da campioni che vanno da decine di pico-Newton a centinaia di nano-Newton, paragonabili a spaziare di forza che può essere applicato a cellule usando una micropipetta 19. Questo intervallo di forza adatta al…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Gli autori ringraziano il Dott. Paolo Janmey presso l'Università della Pennsylvania per la fornitura di linee cellulari utilizzate in questo documento. QW anche riconoscere JF Byfield e Evan Anderson per le loro discussioni penetranti sulle tecniche di AFM.
References
- Discher, D. E., Janmey, P., Wang, Y. L. Tissue cells feel and respond to the stiffness of their substrate. Science. 310, 1139-1143 (2005).
- Wagner, O. I., et al. Softness, strength and self-repair in intermediate filament networks. Exp. Cell Res. 313, 2228-2235 (2007).
- Wang, N., et al. Mechanical behavior in living cells consistent with the tensegrity model. Proceedings of the National Academy of Sciences of the United States of America. 98, 7765-7770 (2001).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. American Journal of Physiology. Cell Physiology. 282, 606-616 (2002).
- Kasza, K. E., et al. Filamin A is essential for active cell stiffening but not passive stiffening under external force. Biophysical Journal. 96, 4326-4335 (2009).
- Elson, E. L. Cellular mechanics as an indicator of cytoskeletal structure and function. Annual Review of Biophysics and Biophysical Chemistry. 17, 397-430 (1988).
- Schafer, A., Radmacher, M. Influence of myosin II activity on stiffness of fibroblast cells. Acta Biomaterialia. 1, 273-280 (2005).
- Guck, J., et al. Optical deformability as an inherent cell marker for testing malignant transformation and metastatic competence. Biophysical Journal. 88, 3689-3698 (2005).
- Cross, S. E., Jin, Y. S., Rao, J., Gimzewski, J. K. Nanomechanical analysis of cells from cancer patients. Nature Nanotechnology. 2, 780-783 (2007).
- Plodinec, M., et al. The nanomechanical signature of breast cancer. Nature Nanotechnology. 7, 757-765 (2012).
- Rotsch, C., Radmacher, M. Drug-induced changes of cytoskeletal structure and mechanics in fibroblasts: an atomic force microscopy study. Biophysical Journal. 78 (00), 520-535 (2000).
- Cross, S. E., Jin, Y. S., Lu, Q. Y., Rao, J., Gimzewski, J. K. Green tea extract selectively targets nanomechanics of live metastatic cancer cells. Nanotechnology. 22, 215101 (2011).
- Hoffman, B. D., Crocker, J. C. Cell mechanics: dissecting the physical responses of cells to force. Annual Review of Biomedical Engineering. 11, 259-288 (2009).
- Hoffman, B. D., Massiera, G., Van Citters, K. M., Crocker, J. C. The consensus mechanics of cultured mammalian cells. Proceedings of the National Academy of Sciences of the United States of America. 103, 10259-10264 (2006).
- Lau, A. W., Hoffman, B. D., Davies, A., Crocker, J. C., Lubensky, T. C. Microrheology, stress fluctuations, and active behavior of living cells. Physical Review Letters. 91, 198101 (1981).
- Liu, J., et al. Microrheology probes length scale dependent rheology. Physical Review Letters. 96, 118104 (2006).
- Deng, L., et al. Fast and slow dynamics of the cytoskeleton. Nature Materials. 5, 636-640 (2006).
- Oh, M. J., Kuhr, F., Byfield, F., Levitan, I. Micropipette Aspiration of Substrate-attached Cells to Estimate Cell Stiffness. J. Vis. Exp. (67), e3886 (2012).
- Hochmuth, R. M. Micropipette aspiration of living cells. Journal of Biomechanics. 33, 15-22 (2000).
- Levental, I., et al. A simple indentation device for measuring micrometer-scale tissue stiffness. J. Phys-Condens. Mat. 22, (2010).
- Mahaffy, R. E., Park, S., Gerde, E., Kas, J., Shih, C. K. Quantitative analysis of the viscoelastic properties of thin regions of fibroblasts using atomic force microscopy. Biophysical Journal. 86, 1777-1793 (2004).
- Radmacher, M. Measuring the elastic properties of biological samples with the AFM. IEEE Engineering in Medicine and Biology Magazine: The Quarterly Magazine of the Engineering in Medicine & Biology Society. 16, 47-57 (1997).
- Crocker, J. C., Hoffman, B. D. Multiple-particle tracking and two-point microrheology in cells. Methods in Cell Biology. 83, 141-178 (2007).
- Liu, F., Tschumperlin, D. J. Micro-mechanical characterization of lung tissue using atomic force microscopy. J. Vis. Exp. (54), e2911 (2011).
- Solon, J., Levental, I., Sengupta, K., Georges, P. C., Janmey, P. A. Fibroblast adaptation and stiffness matching to soft elastic substrates. Biophysical Journal. 93, 4453-4461 (2007).
- Wu, H. W., Kuhn, T., Moy, V. T. Mechanical properties of l929 cells measured by atomic force microscopy: Effects of anticytoskeletal drugs and membrane crosslinking. Scanning. 20, 389-397 (1998).
- Radmacher, M., Fritz, M., Kacher, C. M., Cleveland, J. P., Hansma, P. K. Measuring the viscoelastic properties of human platelets with the atomic force microscope. Biophysical Journal. 70, 556-567 (1996).
- Levy, R., Maaloum, M. Measuring the spring constant of atomic force microscope cantilevers: thermal fluctuations and other methods. Nanotechnology. 13, 33-37 (2002).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis–I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129, 430-440 (2007).
- Davis, J. T., Wen, Q., Janmey, P. A., Otteson, D. C., Foster, W. J. Muller cell expression of genes implicated in proliferative vitreoretinopathy is influenced by substrate elastic modulus. Investigative Ophthalmology & Visual Science. 53, 3014-3019 (2012).
- Engler, A. J., Rehfeldt, F., Sen, S., Discher, D. E. Microtissue elasticity: Measurements by atomic force microscopy and its influence on cell differentiation. Method Cell Biol. 83, 521-545 (2007).
- Lele, T. P., et al. Tools to study cell mechanics and mechanotransduction. Methods in Cell Biology. 83 (07), 443-472 (2007).
- A-Hassan, E., et al. Relative microelastic mapping of living cells by atomic force microscopy. Biophysical Journal. 74, 1564-1578 (1998).
- Costa, K. D., Sim, A. J., Yin, F. C. Non-Hertzian approach to analyzing mechanical properties of endothelial cells probed by atomic force microscopy. Journal of Biomechanical Engineering. 128, 176-184 (2006).
- Xiong, Y., Lee, A. C., Suter, D. M., Lee, G. U. Topography and nanomechanics of live neuronal growth cones analyzed by atomic force microscopy. Biophysical Journal. 96, 5060-5072 (2009).
- Byfield, F. J., et al. Absence of filamin A prevents cells from responding to stiffness gradients on gels coated with collagen but not fibronectin. Biophysical Journal. 96, 5095-5102 (2009).
- Cross, S. E., et al. AFM-based analysis of human metastatic cancer cells. Nanotechnology. 19, 384003 (2008).
- Storm, C., Pastore, J. J., MacKintosh, F. C., Lubensky, T. C., Janmey, P. A. Nonlinear elasticity in biological gels. Nature. 435, 191-194 (2005).
- Wen, Q., Janmey, P. A. Polymer physics of the cytoskeleton. Current Opinion in Solid State & Materials Science. 15, 177-182 (2011).
- Dimitriadis, E. K., Horkay, F., Maresca, J., Kachar, B., Chadwick, R. S. Determination of elastic moduli of thin layers of soft material using the atomic force microscope. Biophysical Journal. 82 (02), 2798-2810 (2002).
- Trache, A., Lim, S. M. Live cell response to mechanical stimulation studied by integrated optical and atomic force microscopy. J. Vis. Exp. (44), e2072 (2010).
- Lim, S. M., Kreipe, B. A., Trzeciakowski, J., Dangott, L., Trache, A. Extracellular matrix effect on RhoA signaling modulation in vascular smooth muscle cells. Exp. Cell Res. 316, 2833-2848 (2010).

