Medir as propriedades mecânicas das células vivas Usando Microscopia de Força Atômica
Summary
Este documento revela um protocolo para caracterizar as propriedades mecânicas de células vivas por meio de microindentação utilizando um microscópio de força atómica (AFM).
Abstract
As propriedades mecânicas das células e da matriz extracelular (ECM) desempenham papéis importantes em muitos processos biológicos, incluindo a diferenciação de células estaminais, a formação de tumores e cicatrização de feridas. Alterações na rigidez de células e de ECM são frequentemente sinais de alterações na fisiologia da célula ou doenças em tecidos. Assim, a rigidez da célula é um índice para avaliar a qualidade de culturas de células. Entre a multiplicidade de métodos aplicados para medir a rigidez das células e tecidos, micro-recuo usando um Microscópio de Força Atômica (AFM) fornece uma maneira de medir de forma confiável a rigidez das células vivas. Este método tem sido amplamente utilizado para caracterizar a rigidez em micro-escala de uma variedade de materiais que vão desde as superfícies metálicas para os tecidos moles e as células biológicas. O princípio básico deste método é recuar um celular com uma ponta do AFM da geometria selecionada e medir a força aplicada a partir da flexão do cantilever AFM. Montagem da curva força-recuo para o modo de Hertzl para a geometria da ponta correspondente pode dar medições quantitativas de rigidez do material. Este documento revela o procedimento para caracterizar a rigidez de células vivas utilizando AFM. Etapas-chave, incluindo o processo de AFM calibração, aquisição de força curva, e análise de dados usando uma rotina MATLAB são demonstrados. As limitações deste método também são discutidos.
Introduction
As propriedades mecânicas, em especial a rigidez, de células individuais e as suas áreas de matrizes extracelulares (ECM) são críticos para muitos processos biológicos incluindo o crescimento celular, motilidade, divisão, diferenciação e na homeostase de tecido. Uma Foi demonstrado que a rigidez mecânica da célula é determinada principalmente pela do citoesqueleto, em particular as redes de filamentos de actina e intermédios e outras proteínas associadas a eles. 2 Resultados dos testes mecânicos nas redes em in vitro de filamentos de actina e intermediários sugerem que os mecanismos celulares é em grande parte dependente da estrutura do citoesqueleto e a pré-tensão no o citoesqueleto. 3-5 Rigidez de células vivas é então considerado como um índice para avaliar a estrutura do citoesqueleto 6, 7 e actividade miosina muitos outros processos celulares. Mais importante ainda, as alterações nas propriedades mecânicas das células são também frequentemente encontrado para ser estreitamente associadaciado a várias condições de doença, tais como a formação de tumores e metástases 8-10 Controlo da rigidez mecânica de células vivas podem, portanto, proporcionar uma nova forma de controlar a fisiologia da célula,. de detectar e diagnosticar doenças 8,. e para avaliar a eficácia dos tratamentos com medicamentos 11 , 12
Vários métodos, incluindo microrheology partícula de rastreamento, 13-16 citometria de torção magnética, 17 micropipeta aspiração microindentação 18,19 e 20-22 foram desenvolvidos para medir a elasticidade das células. Partícula de rastreamento microrheology traça as vibrações térmicas, tanto de partículas submicrométricas fluorescentes injectados em células ou marcadores fiduciais dentro do citoesqueleto celular. 23 propriedades elásticas e viscosas de células são calculados a partir dos deslocamentos de partículas medidos usando o teorema de dissipação de flutuação. 14,23 Este método permite medições simultâneas de locaispropriedades mecânicas com alta resolução espacial em diferentes lugares em uma célula. No entanto, a injeção de partículas fluorescentes em células pode levar a alterações na função celular, a estrutura do citoesqueleto, e, portanto, a mecânica da célula. O método aplica-se a aspiração de pressão negativa micropipeta em uma micropipeta de diâmetro variando entre 1 e 5 um para chupar um pequeno pedaço de membrana celular para a pipeta. Rigidez das células é calculada a partir da pressão negativa aplicada e deformação da membrana celular. 18 Este método, no entanto, não é possível detectar a distribuição heterogénea da rigidez em toda a célula. Citometria de torção magnética (MTC) se aplica o campo magnético para gerar binário em grânulos super-paramagnéticas ligadas à membrana celular. Celular rigidez 17 é derivado no presente método a partir da relação entre o binário aplicado e a deformação de torção da membrana celular. É difícil controlar a localização das esferas magnéticas no método MTC, e é também challenging a caracterizar a deformação de torção, com alta resolução. Microindentação aplica um penetrador com uma geometria bem definida, para perfurar na célula. A força de recuo e o recuo, resultando em células seguem frequentemente a previsão do modelo Hertz. Módulos de Young de células pode ser calculada a partir das curvas de força de recuo, equipando-os com o modelo Hertz. Este método tem sido amplamente utilizado para testar as propriedades mecânicas do tecido, e as células, apesar de as suas limitações, tais como a incerteza na determinação do ponto de contacto, a aplicabilidade do modelo de Hertz, e o potencial para danificar fisicamente as células. Entre os muitos dispositivos para microindentaion 20, o Microscópio de Força Atômica (AFM) está disponível comercialmente e tem sido amplamente aplicada para caracterizar as propriedades mecânicas das células vivas e tecidos 21,24-27.
Este trabalho demonstra o procedimento de usar um Asylum MFP3D-Bio AFM para caracterizar mecânica celulares. AFM não emly fornece topografia de alta resolução das células, mas também tem sido amplamente aplicada para caracterizar as propriedades mecânicas das células dos tecidos. O princípio da AFM recuo é ilustrada na Figura 1. O braço de suporte de AFM se aproxima da pilha de alguns micrómetros acima; faz contacto com a célula, travessões, a célula de modo que a deformação do cantilever atinge um ponto de ajuste pré-seleccionado, e puxa para fora da célula. Durante este processo, a deformação do cantilever é registada como uma função da sua posição, como mostrado na Figura 1. Antes de fazer o contacto com a célula, o cantilever se move no meio sem qualquer deflexão aparente. Quando recuo sobre a célula, as curvas em cantilever e os aumentos de sinal de deflexão. As vigas são modeladas como vigas elásticas, de modo que a sua deformação é proporcional à força aplicada à célula. Ao definir a deflexão máxima cantiléver, a magnitude da força máxima aplicada à amostra é limitado para evitar dAmage às células. A parte da curva de força a partir do ponto b ao ponto C na Figura 1, onde os travessões da ponta para dentro da célula, está apto para o modelo hertz para extrair a rigidez da célula.
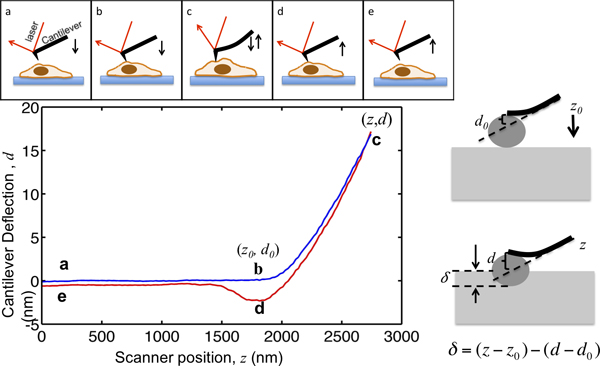
Figura 1. Ilustração da AFM microindentação e interpretação da curva de força. O painel superior mostra o movimento de AFM cantilever impulsionado pelo scanner piezo. A localização vertical do braço de suporte e o braço de suporte z deflexão d sinal é registado durante o processo. O braço de suporte começa a partir de um ponto, alguns micrómetros acima da célula. Ao aproximarem-se da célula, o recuo δ amostra permanece zero até atingir o ponto b, onde a ponta entra em contacto com a célula. As coordenadas do ponto b no gráfico são os valores críticospara a análise de dados, indicado por (z 0, d 0>). De b para c, os travessões cantilever na célula até que a deflexão do cantilever atinge um ponto de ajuste, que é definido como sendo a relação entre a força de recuo máximo alvo ea constante da mola cantilever. Uma vez que o sinal de deflexão atinge o valor máximo pré-definido, o cantilever é então retirado a partir da célula até ao ponto d, em que, muitas vezes, ser puxado para baixo devido à adesão de ponta-amostra, destaca a partir da célula e retorna à sua posição inicial em E. O painel da direita mostra a relação entre a reentrância e o sinal gravado e z d. Em no painel inferior esquerdo é um gráfico de uma curva representativa de força, o recuo máximo de um braço de suporte, das quais a constante de mola é medido para ser 0.07N / m, é ajustado para ser de 17 nm, de modo que a máxima força de recuo aplicada ao amostra é de 1,2 nN. Os locais-chave durante o recuo são marcadas.
Protocol
Representative Results
Discussion
O método de recuo AFM tem vantagens para caracterizar as propriedades mecânicas de células vivas. Embora menos sensível do que a citometria de torção magnética e pinças ópticas, que pode medir forças no nível piconewton 32, o AFM pode detectar a força de resistência de amostras variando de dezenas de pico-Newton para centenas de nano-Newton, comparáveis a gama de força que pode ser aplicado a células utilizando uma micropipeta 19. Esta gama de força se adapta às necessidades…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Os autores agradecem ao Dr. Paul Janmey na Universidade da Pensilvânia, para a prestação de linhas celulares utilizadas neste trabalho. QW também reconhecer JF Byfield e Evan Anderson para suas discussões criteriosas sobre técnicas de AFM.
References
- Discher, D. E., Janmey, P., Wang, Y. L. Tissue cells feel and respond to the stiffness of their substrate. Science. 310, 1139-1143 (2005).
- Wagner, O. I., et al. Softness, strength and self-repair in intermediate filament networks. Exp. Cell Res. 313, 2228-2235 (2007).
- Wang, N., et al. Mechanical behavior in living cells consistent with the tensegrity model. Proceedings of the National Academy of Sciences of the United States of America. 98, 7765-7770 (2001).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. American Journal of Physiology. Cell Physiology. 282, 606-616 (2002).
- Kasza, K. E., et al. Filamin A is essential for active cell stiffening but not passive stiffening under external force. Biophysical Journal. 96, 4326-4335 (2009).
- Elson, E. L. Cellular mechanics as an indicator of cytoskeletal structure and function. Annual Review of Biophysics and Biophysical Chemistry. 17, 397-430 (1988).
- Schafer, A., Radmacher, M. Influence of myosin II activity on stiffness of fibroblast cells. Acta Biomaterialia. 1, 273-280 (2005).
- Guck, J., et al. Optical deformability as an inherent cell marker for testing malignant transformation and metastatic competence. Biophysical Journal. 88, 3689-3698 (2005).
- Cross, S. E., Jin, Y. S., Rao, J., Gimzewski, J. K. Nanomechanical analysis of cells from cancer patients. Nature Nanotechnology. 2, 780-783 (2007).
- Plodinec, M., et al. The nanomechanical signature of breast cancer. Nature Nanotechnology. 7, 757-765 (2012).
- Rotsch, C., Radmacher, M. Drug-induced changes of cytoskeletal structure and mechanics in fibroblasts: an atomic force microscopy study. Biophysical Journal. 78 (00), 520-535 (2000).
- Cross, S. E., Jin, Y. S., Lu, Q. Y., Rao, J., Gimzewski, J. K. Green tea extract selectively targets nanomechanics of live metastatic cancer cells. Nanotechnology. 22, 215101 (2011).
- Hoffman, B. D., Crocker, J. C. Cell mechanics: dissecting the physical responses of cells to force. Annual Review of Biomedical Engineering. 11, 259-288 (2009).
- Hoffman, B. D., Massiera, G., Van Citters, K. M., Crocker, J. C. The consensus mechanics of cultured mammalian cells. Proceedings of the National Academy of Sciences of the United States of America. 103, 10259-10264 (2006).
- Lau, A. W., Hoffman, B. D., Davies, A., Crocker, J. C., Lubensky, T. C. Microrheology, stress fluctuations, and active behavior of living cells. Physical Review Letters. 91, 198101 (1981).
- Liu, J., et al. Microrheology probes length scale dependent rheology. Physical Review Letters. 96, 118104 (2006).
- Deng, L., et al. Fast and slow dynamics of the cytoskeleton. Nature Materials. 5, 636-640 (2006).
- Oh, M. J., Kuhr, F., Byfield, F., Levitan, I. Micropipette Aspiration of Substrate-attached Cells to Estimate Cell Stiffness. J. Vis. Exp. (67), e3886 (2012).
- Hochmuth, R. M. Micropipette aspiration of living cells. Journal of Biomechanics. 33, 15-22 (2000).
- Levental, I., et al. A simple indentation device for measuring micrometer-scale tissue stiffness. J. Phys-Condens. Mat. 22, (2010).
- Mahaffy, R. E., Park, S., Gerde, E., Kas, J., Shih, C. K. Quantitative analysis of the viscoelastic properties of thin regions of fibroblasts using atomic force microscopy. Biophysical Journal. 86, 1777-1793 (2004).
- Radmacher, M. Measuring the elastic properties of biological samples with the AFM. IEEE Engineering in Medicine and Biology Magazine: The Quarterly Magazine of the Engineering in Medicine & Biology Society. 16, 47-57 (1997).
- Crocker, J. C., Hoffman, B. D. Multiple-particle tracking and two-point microrheology in cells. Methods in Cell Biology. 83, 141-178 (2007).
- Liu, F., Tschumperlin, D. J. Micro-mechanical characterization of lung tissue using atomic force microscopy. J. Vis. Exp. (54), e2911 (2011).
- Solon, J., Levental, I., Sengupta, K., Georges, P. C., Janmey, P. A. Fibroblast adaptation and stiffness matching to soft elastic substrates. Biophysical Journal. 93, 4453-4461 (2007).
- Wu, H. W., Kuhn, T., Moy, V. T. Mechanical properties of l929 cells measured by atomic force microscopy: Effects of anticytoskeletal drugs and membrane crosslinking. Scanning. 20, 389-397 (1998).
- Radmacher, M., Fritz, M., Kacher, C. M., Cleveland, J. P., Hansma, P. K. Measuring the viscoelastic properties of human platelets with the atomic force microscope. Biophysical Journal. 70, 556-567 (1996).
- Levy, R., Maaloum, M. Measuring the spring constant of atomic force microscope cantilevers: thermal fluctuations and other methods. Nanotechnology. 13, 33-37 (2002).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis–I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129, 430-440 (2007).
- Davis, J. T., Wen, Q., Janmey, P. A., Otteson, D. C., Foster, W. J. Muller cell expression of genes implicated in proliferative vitreoretinopathy is influenced by substrate elastic modulus. Investigative Ophthalmology & Visual Science. 53, 3014-3019 (2012).
- Engler, A. J., Rehfeldt, F., Sen, S., Discher, D. E. Microtissue elasticity: Measurements by atomic force microscopy and its influence on cell differentiation. Method Cell Biol. 83, 521-545 (2007).
- Lele, T. P., et al. Tools to study cell mechanics and mechanotransduction. Methods in Cell Biology. 83 (07), 443-472 (2007).
- A-Hassan, E., et al. Relative microelastic mapping of living cells by atomic force microscopy. Biophysical Journal. 74, 1564-1578 (1998).
- Costa, K. D., Sim, A. J., Yin, F. C. Non-Hertzian approach to analyzing mechanical properties of endothelial cells probed by atomic force microscopy. Journal of Biomechanical Engineering. 128, 176-184 (2006).
- Xiong, Y., Lee, A. C., Suter, D. M., Lee, G. U. Topography and nanomechanics of live neuronal growth cones analyzed by atomic force microscopy. Biophysical Journal. 96, 5060-5072 (2009).
- Byfield, F. J., et al. Absence of filamin A prevents cells from responding to stiffness gradients on gels coated with collagen but not fibronectin. Biophysical Journal. 96, 5095-5102 (2009).
- Cross, S. E., et al. AFM-based analysis of human metastatic cancer cells. Nanotechnology. 19, 384003 (2008).
- Storm, C., Pastore, J. J., MacKintosh, F. C., Lubensky, T. C., Janmey, P. A. Nonlinear elasticity in biological gels. Nature. 435, 191-194 (2005).
- Wen, Q., Janmey, P. A. Polymer physics of the cytoskeleton. Current Opinion in Solid State & Materials Science. 15, 177-182 (2011).
- Dimitriadis, E. K., Horkay, F., Maresca, J., Kachar, B., Chadwick, R. S. Determination of elastic moduli of thin layers of soft material using the atomic force microscope. Biophysical Journal. 82 (02), 2798-2810 (2002).
- Trache, A., Lim, S. M. Live cell response to mechanical stimulation studied by integrated optical and atomic force microscopy. J. Vis. Exp. (44), e2072 (2010).
- Lim, S. M., Kreipe, B. A., Trzeciakowski, J., Dangott, L., Trache, A. Extracellular matrix effect on RhoA signaling modulation in vascular smooth muscle cells. Exp. Cell Res. 316, 2833-2848 (2010).

