Medición de las propiedades mecánicas de las células vivas mediante microscopía de fuerza atómica
Summary
Este documento demuestra un protocolo para caracterizar las propiedades mecánicas de las células vivas por medio de microindentación utilizando un Microscopio de Fuerza Atómica (AFM).
Abstract
Las propiedades mecánicas de las células y la matriz extracelular (ECM) desempeñan papeles importantes en muchos procesos biológicos, incluyendo la diferenciación de células madre, la formación del tumor, y la cicatrización de heridas. Los cambios en la rigidez de las células y ECM son a menudo signos de cambios en la fisiología celular o enfermedades en los tejidos. Por lo tanto, la rigidez de células es un índice para evaluar el estado de los cultivos celulares. Entre la multitud de métodos aplicados para medir la rigidez de las células y tejidos, micro-indentación usando un Microscopio de Fuerza Atómica (AFM) proporciona una manera de medir de forma fiable la rigidez de las células vivas. Este método ha sido aplicado ampliamente para caracterizar la rigidez de micro-escala para una variedad de materiales que van desde superficies metálicas a los tejidos biológicos blandos y células. El principio básico de este método es para sangrar una célula con una punta de AFM de la geometría seleccionada y medir la fuerza aplicada a partir de la flexión del cantilever de AFM. Montaje de la curva fuerza-sangría para el modo de Hertzl para la geometría de la punta correspondiente puede dar mediciones cuantitativas de la rigidez del material. En este trabajo se demuestra el procedimiento para caracterizar la rigidez de las células vivas utilizando AFM. Se demuestran los pasos clave, incluyendo el proceso de calibración de AFM, la adquisición de la fuerza de la curva, y el análisis de datos mediante una rutina de MATLAB. También se discuten las limitaciones de este método.
Introduction
Las propiedades mecánicas, especialmente la rigidez, de células individuales y sus matrices extracelulares que rodean (ECM) son fundamentales para muchos procesos biológicos, incluyendo el crecimiento celular, la motilidad, la división, diferenciación, y la homeostasis del tejido. 1 Se ha demostrado que la rigidez mecánica de células está determinada principalmente por el citoesqueleto, especialmente las redes de actina y filamentos intermedios y otras proteínas asociadas con ellos. 2 Los resultados de ensayos mecánicos sobre en redes in vitro de actina y filamentos intermedios sugieren que la mecánica de células es dependiente en gran medida de la estructura del citoesqueleto y la pre-tensión en el citoesqueleto. 3-5 Rigidez de células vivas se considera entonces como un índice para evaluar la estructura del citoesqueleto 6, la actividad de la miosina 7 y muchos otros procesos celulares. Más importante aún, los cambios en las propiedades mecánicas de células también se encuentran a menudo para ser estrechamente asociadosocasionada por la diversas condiciones de enfermedad, tales como la formación de tumores y la metástasis 8-10 Control de la rigidez mecánica de las células vivas por lo tanto, puede proporcionar una nueva manera de controlar la fisiología celular;. para detectar y diagnosticar enfermedades 8;., y para evaluar la eficacia de los tratamientos farmacológicos 11 , 12
Múltiples métodos incluyendo microrheology partícula de seguimiento, 13-16 magnética citometría de torsión, 17 micropipeta aspiración 18,19 y microindentación 20-22 se han desarrollado para medir la elasticidad de las células. Partículas microrheology seguimiento rastrea las vibraciones térmicas de cualquiera de las partículas fluorescentes submicrométricos inyectados en células o marcadores de referencia dentro del citoesqueleto celular. 23 propiedades elásticas y viscosas de células se calculan a partir de los desplazamientos de partículas medidos utilizando el teorema de fluctuación-disipación. 14,23 Este método permite mediciones simultáneas de localespropiedades mecánicas con alta resolución espacial en distintos lugares de una célula. Sin embargo, la inyección de partículas fluorescentes en las células puede conducir a cambios en la función celular, la estructura del citoesqueleto, y por lo tanto la mecánica celular. El método de aspiración con micropipeta se aplica presión negativa en una micropipeta de diámetro varía de 1 a 5 micras para aspirar una pequeña pieza de membrana celular en la pipeta. Rigidez celular se calcula a partir de la presión negativa aplicada y la deformación de la membrana celular. 18 Este método, sin embargo, no puede detectar la distribución heterogénea de la rigidez a través de la célula. Citometría de torsión magnética (MTC) se aplica el campo magnético para generar par de torsión sobre perlas de súper paramagnéticas unidas a la membrana celular. 17 rigidez célula se deriva en este método a partir de la relación entre el par aplicado y la deformación de torsión de la membrana celular. Es difícil controlar la ubicación de perlas magnéticas en el método de MTC, y también es challenging para caracterizar la deformación de torsión con alta resolución. Microindentación se aplica un penetrador con una geometría bien definida para perforar en la célula. La fuerza de penetración y la indentación resultante en las células a menudo siguen la predicción del modelo de Hertz. Módulos de Young de las células se puede calcular a partir de las curvas de fuerza-indentación ajustándolas al modelo de Hertz. Este método ha sido ampliamente aplicado para probar las propiedades mecánicas de los tejidos y las células a pesar de sus limitaciones, tales como la incertidumbre en la determinación del punto de contacto, la aplicabilidad del modelo de Hertz, y el potencial para dañar físicamente las células. Entre los muchos dispositivos para microindentaion 20, el Microscopio de Fuerza Atómica (AFM) está disponible comercialmente y se ha aplicado ampliamente para caracterizar las propiedades mecánicas de las células y tejidos vivos 21,24-27.
En este trabajo se demuestra el procedimiento de utilizar un asilo MFP3D-Bio AFM para caracterizar la mecánica celular. AFM no enLY proporciona topografía de alta resolución de las células, pero también se ha aplicado ampliamente para caracterizar las propiedades mecánicas de las células del tejido. El principio de AFM indentación se ilustra en la Figura 1. El cantilever de AFM se aproxima a la célula desde unos pocos micrómetros más arriba; hace contacto con la célula; guiones la célula de modo que la deflexión voladizo alcanza un punto de consigna preseleccionado, y se aleja de la célula. Durante este proceso, la deflexión en voladizo se registra como una función de su ubicación, como se muestra en la Figura 1. Antes de hacer contacto con la célula, el voladizo se mueve en el medio sin ninguna desviación aparente. Cuando sangría en la célula, las curvas en voladizo y se incrementa la señal de deflexión. Los voladizos se modelan como vigas elásticas para que su desviación es proporcional a la fuerza aplicada a la célula. Al establecer la deflexión máxima en voladizo, la máxima magnitud de la fuerza aplicada a la muestra es limitada para evitar dNingún daño a las células. La porción de la curva de fuerza desde el punto B al punto C en la Figura 1, donde los guiones punta en la célula, se adaptan al modelo de hercios para extraer la rigidez celular.
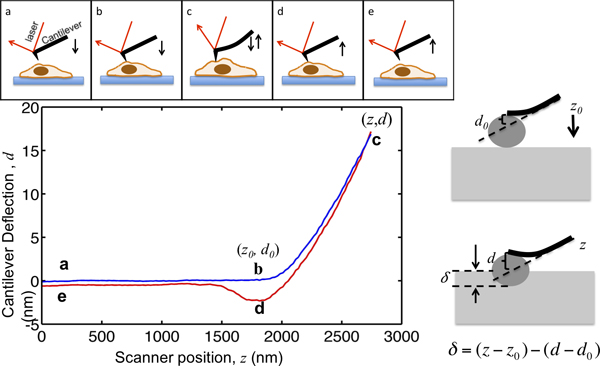
Figura 1. Ilustración de la AFM microindentación e interpretación de la curva de fuerza. El panel superior muestra el movimiento de la AFM voladizo accionada por el escáner piezoeléctrico. La ubicación vertical del voladizo z y la deflexión en voladizo señal d se registra durante el proceso. El voladizo se inicia desde el punto a, unos pocos micrómetros por encima de la celda. Al acercarse a la célula, la muestra δ indentación permanece en cero hasta que se alcanza el punto B, donde la punta entra en contacto con la célula. Las coordenadas del punto b en el diagrama son valores críticospara el análisis de datos, que se denota por (z 0, d 0>). De B a C, los guiones en voladizo en la célula hasta que la deflexión voladizo llega a un punto de ajuste, que se ajusta para que sea la relación entre la fuerza de penetración máxima al final del voladizo y la constante del resorte. Una vez que la señal de deflexión alcanza el valor máximo prefijado, el voladizo se retira entonces de la célula al punto d, donde a menudo ser tirado hacia abajo debido a la adhesión de la punta-de la muestra, se separa de la célula y vuelve a su ubicación inicial en el correo. El panel de la derecha ilustra la relación entre la indentación y la z grabada y la señal d. En en el panel inferior izquierdo es un gráfico de una curva representativa fuerza, la indentación máximo de un voladizo, de que la constante del resorte se mide a ser 0.07N / m, se ajusta para que sea 17 nm de manera que la fuerza máxima aplicada a la sangría muestra es 1,2 nN. Los puntos clave durante el sangrado se marcan.
Protocol
Representative Results
Discussion
El método de indentación AFM tiene ventajas para caracterizar las propiedades mecánicas de las células vivas. Aunque menos sensible que la citometría de torsión magnético y pinzas ópticas, que puede medir fuerzas en el nivel picoNewton 32, el AFM puede detectar la fuerza de resistencia de las muestras que van desde decenas de pico-Newton a cientos de nano-Newton, comparable a la gama de la fuerza que se puede aplicar a las células utilizando una micropipeta 19. Este rango de la fuerza se a…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Los autores agradecen al Dr. Paul Janmey en la Universidad de Pennsylvania para el suministro de las líneas celulares utilizadas en este trabajo. QW también reconoce JF Byfield y Evan Anderson por sus discusiones interesantes sobre las técnicas de AFM.
References
- Discher, D. E., Janmey, P., Wang, Y. L. Tissue cells feel and respond to the stiffness of their substrate. Science. 310, 1139-1143 (2005).
- Wagner, O. I., et al. Softness, strength and self-repair in intermediate filament networks. Exp. Cell Res. 313, 2228-2235 (2007).
- Wang, N., et al. Mechanical behavior in living cells consistent with the tensegrity model. Proceedings of the National Academy of Sciences of the United States of America. 98, 7765-7770 (2001).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. American Journal of Physiology. Cell Physiology. 282, 606-616 (2002).
- Kasza, K. E., et al. Filamin A is essential for active cell stiffening but not passive stiffening under external force. Biophysical Journal. 96, 4326-4335 (2009).
- Elson, E. L. Cellular mechanics as an indicator of cytoskeletal structure and function. Annual Review of Biophysics and Biophysical Chemistry. 17, 397-430 (1988).
- Schafer, A., Radmacher, M. Influence of myosin II activity on stiffness of fibroblast cells. Acta Biomaterialia. 1, 273-280 (2005).
- Guck, J., et al. Optical deformability as an inherent cell marker for testing malignant transformation and metastatic competence. Biophysical Journal. 88, 3689-3698 (2005).
- Cross, S. E., Jin, Y. S., Rao, J., Gimzewski, J. K. Nanomechanical analysis of cells from cancer patients. Nature Nanotechnology. 2, 780-783 (2007).
- Plodinec, M., et al. The nanomechanical signature of breast cancer. Nature Nanotechnology. 7, 757-765 (2012).
- Rotsch, C., Radmacher, M. Drug-induced changes of cytoskeletal structure and mechanics in fibroblasts: an atomic force microscopy study. Biophysical Journal. 78 (00), 520-535 (2000).
- Cross, S. E., Jin, Y. S., Lu, Q. Y., Rao, J., Gimzewski, J. K. Green tea extract selectively targets nanomechanics of live metastatic cancer cells. Nanotechnology. 22, 215101 (2011).
- Hoffman, B. D., Crocker, J. C. Cell mechanics: dissecting the physical responses of cells to force. Annual Review of Biomedical Engineering. 11, 259-288 (2009).
- Hoffman, B. D., Massiera, G., Van Citters, K. M., Crocker, J. C. The consensus mechanics of cultured mammalian cells. Proceedings of the National Academy of Sciences of the United States of America. 103, 10259-10264 (2006).
- Lau, A. W., Hoffman, B. D., Davies, A., Crocker, J. C., Lubensky, T. C. Microrheology, stress fluctuations, and active behavior of living cells. Physical Review Letters. 91, 198101 (1981).
- Liu, J., et al. Microrheology probes length scale dependent rheology. Physical Review Letters. 96, 118104 (2006).
- Deng, L., et al. Fast and slow dynamics of the cytoskeleton. Nature Materials. 5, 636-640 (2006).
- Oh, M. J., Kuhr, F., Byfield, F., Levitan, I. Micropipette Aspiration of Substrate-attached Cells to Estimate Cell Stiffness. J. Vis. Exp. (67), e3886 (2012).
- Hochmuth, R. M. Micropipette aspiration of living cells. Journal of Biomechanics. 33, 15-22 (2000).
- Levental, I., et al. A simple indentation device for measuring micrometer-scale tissue stiffness. J. Phys-Condens. Mat. 22, (2010).
- Mahaffy, R. E., Park, S., Gerde, E., Kas, J., Shih, C. K. Quantitative analysis of the viscoelastic properties of thin regions of fibroblasts using atomic force microscopy. Biophysical Journal. 86, 1777-1793 (2004).
- Radmacher, M. Measuring the elastic properties of biological samples with the AFM. IEEE Engineering in Medicine and Biology Magazine: The Quarterly Magazine of the Engineering in Medicine & Biology Society. 16, 47-57 (1997).
- Crocker, J. C., Hoffman, B. D. Multiple-particle tracking and two-point microrheology in cells. Methods in Cell Biology. 83, 141-178 (2007).
- Liu, F., Tschumperlin, D. J. Micro-mechanical characterization of lung tissue using atomic force microscopy. J. Vis. Exp. (54), e2911 (2011).
- Solon, J., Levental, I., Sengupta, K., Georges, P. C., Janmey, P. A. Fibroblast adaptation and stiffness matching to soft elastic substrates. Biophysical Journal. 93, 4453-4461 (2007).
- Wu, H. W., Kuhn, T., Moy, V. T. Mechanical properties of l929 cells measured by atomic force microscopy: Effects of anticytoskeletal drugs and membrane crosslinking. Scanning. 20, 389-397 (1998).
- Radmacher, M., Fritz, M., Kacher, C. M., Cleveland, J. P., Hansma, P. K. Measuring the viscoelastic properties of human platelets with the atomic force microscope. Biophysical Journal. 70, 556-567 (1996).
- Levy, R., Maaloum, M. Measuring the spring constant of atomic force microscope cantilevers: thermal fluctuations and other methods. Nanotechnology. 13, 33-37 (2002).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis–I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129, 430-440 (2007).
- Davis, J. T., Wen, Q., Janmey, P. A., Otteson, D. C., Foster, W. J. Muller cell expression of genes implicated in proliferative vitreoretinopathy is influenced by substrate elastic modulus. Investigative Ophthalmology & Visual Science. 53, 3014-3019 (2012).
- Engler, A. J., Rehfeldt, F., Sen, S., Discher, D. E. Microtissue elasticity: Measurements by atomic force microscopy and its influence on cell differentiation. Method Cell Biol. 83, 521-545 (2007).
- Lele, T. P., et al. Tools to study cell mechanics and mechanotransduction. Methods in Cell Biology. 83 (07), 443-472 (2007).
- A-Hassan, E., et al. Relative microelastic mapping of living cells by atomic force microscopy. Biophysical Journal. 74, 1564-1578 (1998).
- Costa, K. D., Sim, A. J., Yin, F. C. Non-Hertzian approach to analyzing mechanical properties of endothelial cells probed by atomic force microscopy. Journal of Biomechanical Engineering. 128, 176-184 (2006).
- Xiong, Y., Lee, A. C., Suter, D. M., Lee, G. U. Topography and nanomechanics of live neuronal growth cones analyzed by atomic force microscopy. Biophysical Journal. 96, 5060-5072 (2009).
- Byfield, F. J., et al. Absence of filamin A prevents cells from responding to stiffness gradients on gels coated with collagen but not fibronectin. Biophysical Journal. 96, 5095-5102 (2009).
- Cross, S. E., et al. AFM-based analysis of human metastatic cancer cells. Nanotechnology. 19, 384003 (2008).
- Storm, C., Pastore, J. J., MacKintosh, F. C., Lubensky, T. C., Janmey, P. A. Nonlinear elasticity in biological gels. Nature. 435, 191-194 (2005).
- Wen, Q., Janmey, P. A. Polymer physics of the cytoskeleton. Current Opinion in Solid State & Materials Science. 15, 177-182 (2011).
- Dimitriadis, E. K., Horkay, F., Maresca, J., Kachar, B., Chadwick, R. S. Determination of elastic moduli of thin layers of soft material using the atomic force microscope. Biophysical Journal. 82 (02), 2798-2810 (2002).
- Trache, A., Lim, S. M. Live cell response to mechanical stimulation studied by integrated optical and atomic force microscopy. J. Vis. Exp. (44), e2072 (2010).
- Lim, S. M., Kreipe, B. A., Trzeciakowski, J., Dangott, L., Trache, A. Extracellular matrix effect on RhoA signaling modulation in vascular smooth muscle cells. Exp. Cell Res. 316, 2833-2848 (2010).

