नैदानिक नमूनों से आरएनए वायरस की निष्पक्ष गहरी अनुक्रमण
Summary
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Abstract
यहाँ हम एक अगली पीढ़ी आरएनए अनुक्रमण प्रोटोकॉल सक्षम बनाता है कि नए सिरे से विधानसभाओं और नैदानिक और जैविक स्रोतों से एकत्र वायरल जीनोम की इंट्रा-मेजबान संस्करण कॉल रूपरेखा। विधि निष्पक्ष और सार्वभौमिक है; यह सीडीएनए संश्लेषण के लिए यादृच्छिक प्राइमरों का उपयोग करता है और वायरल अनुक्रम सामग्री का कोई पूर्व ज्ञान की आवश्यकता है। सहित पाली (आरए) वाहक और ribosomal शाही सेना – – वायरल शाही सेना नमूना से पुस्तकालय निर्माण से पहले, चयनात्मक RNase एच-आधारित पाचन अवांछित आरएनए चूस लेना करने के लिए इस्तेमाल किया जाता है। चयनात्मक कमी दोनों डेटा की गुणवत्ता और अद्वितीय की संख्या वायरल शाही सेना अनुक्रमण पुस्तकालयों में पढ़ता में सुधार। इसके अलावा, एक Transposase-आधारित 'tagmentation' कदम प्रोटोकॉल में इस्तेमाल के रूप में यह समग्र पुस्तकालय निर्माण के समय को कम कर देता है। प्रोटोकॉल के तेजी से गहरी अनुक्रमण के लिए सक्षम है और 600 से अधिक लासा इबोला वायरस के नमूने सहित दोनों के रक्त और ऊतकों को अलग-और मोटे तौर पर अन्य माइक्रोबियल जीनोमिक्स के अध्ययन के लिए लागू है से संग्रह।
Introduction
नैदानिक स्रोतों से वायरस की अगली पीढ़ी के अनुक्रमण पारेषण और संक्रमण के महामारी विज्ञान, साथ ही मदद समर्थन उपन्यास निदान, टीका और चिकित्सीय विकास को सूचित कर सकते हैं। सीडीएनए यादृच्छिक प्राइमरों का उपयोग संश्लेषण का पता लगाने और अलग-अलग, सह-संक्रमण से जीनोम या यहां तक कि उपन्यास वायरस 1,2 के विधानसभा अनुमति दी गई है। अन्य निष्पक्ष तरीकों के साथ के रूप में, अवांछित दूषित पदार्थों को कई अनुक्रमण पढ़ता है और नकारात्मक अनुक्रमण परिणामों को प्रभावित कब्जा करना था। होस्ट और पाली (आरए) वाहक आरएनए कई मौजूदा वायरल नमूना संग्रह में मौजूद दूषित पदार्थों को कर रहे हैं।
प्रोटोकॉल गहरी अनुक्रमण आरएनए वायरस निष्पक्ष कुल शाही सेना-सेक के आधार पर जीनोम के एक कुशल और लागत प्रभावी तरीके से वर्णन करता है। विधि अवांछित मेजबान ribosomal और वाहक आरएनए को हटाने के लिए एक RNase एच चयनात्मक कमी चरण 3 का इस्तेमाल करता है। चयनात्मक कमी वायरल सामग्री (चित्रा 1) के लिए समृद्ध करती और अनुक्रमण डेटा के समग्र गुणवत्ता में सुधार(चित्रा 2) नैदानिक नमूनों से। इसके अलावा, tagmentation प्रोटोकॉल के रूप में यह काफी पुस्तकालय निर्माण के समय को कम कर देता लिए आवेदन किया है। इन तरीकों में तेजी से इबोला और लासा वायरस जीनोम 2,4,5 के बड़े डेटासेट उत्पन्न करने के लिए इस्तेमाल किया गया है और आरएनए वायरस की एक विस्तृत श्रृंखला का अध्ययन करने के लिए इस्तेमाल किया जा सकता है। अन्त में, दृष्टिकोण मानव नमूनों तक सीमित नहीं है; चयनात्मक कमी की उपयोगिता लासा संक्रमित मूषक और गैर मानव प्राइमेट रोग मॉडल 5,6 से एकत्र ऊतकों के नमूनों पर प्रदर्शन किया गया।
चित्रा 1. कुल शाही सेना सामग्री लासा वायरस चयनात्मक कमी का उपयोग कर सामग्री के संवर्धन को दर्शाता है। नौ विभिन्न नैदानिक वियोजन से समग्र सामग्री (आरएनए इनपुट) और अद्वितीय लासा वायरस (LASV) के संवर्धन पढ़ता (लाइब्रेरी सामग्री) rRNA कमी पर शुरू। यह आंकड़ा 6 से संशोधित किया गया है <em>। यह आंकड़ा का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
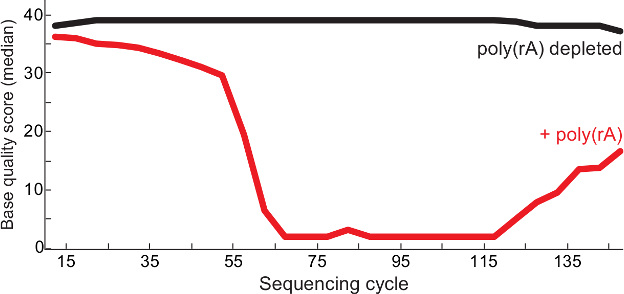
चित्रा 2. उच्च गुणवत्ता अनुक्रमण के बाद कैरियर आरएनए कमी। पाली की अनुक्रमण चक्र (आरए) प्रति माध्य आधार गुणों लासा वायरस पुस्तकालयों (लाल) और QC रिपोर्ट 13 से नियंत्रण (कोई वाहक पुस्तकालय में मनाया, काला) -contaminated। दोनों 1 पढ़ें और पढ़ें बनती अंत के 2 पुस्तकालय बेम फ़ाइल में विलय कर रहे हैं और गुणवत्ता स्कोर के प्रत्येक आधार पर दिखाए जाते हैं पढ़ता है। यह आंकड़ा 6 से संशोधित किया गया है। यह आंकड़ा का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
वायरल शाही सेना seq प्रोटोकॉल विवरण निकाली से सीधे पुस्तकालयों का निर्माणआरएनए नैदानिक और जैविक नमूने से एकत्र की। व्यक्तिगत सुरक्षा सुनिश्चित करने के लिए, सभी वायरल सीरम, प्लाज्मा और ऊतकों के नमूनों आरएनए निकासी करने से पहले उचित buffers में निष्क्रिय किया जाना चाहिए। कुछ निष्क्रियता और निकासी किट में, वाहक पाली (आरए) आरएनए भी शामिल है; इस प्रारंभिक RNase एच चयनात्मक कमी चरण के दौरान हटा दिया जाएगा। पूरी तरह ठीक होने के आधार पर, वाहक आरएनए की उम्मीद है और एकाग्रता 100 एनजी / μl है। प्रोटोकॉल में, 110 एनजी / μl oligo डीटी आरएनए (1.1X वाहक एकाग्रता) की कमी के लिए प्रयोग किया जाता है। अगर पाली (आरए) वाहक नमूने में मौजूद नहीं है, तो oligo (डीटी) कमी करने से पहले नहीं जोड़ा जाना चाहिए।
निम्नलिखित प्रोटोकॉल (250 μl मात्रा तक) पीसीआर थाली प्रारूप में 24 प्रतिक्रियाओं के लिए बनाया गया है। इस प्रोटोकॉल के पुराने संस्करण Matranga, एट अल में बताया गया था। 6।
Protocol
Representative Results
Discussion
उल्लिखित दृष्टिकोण मजबूत, सार्वभौमिक, तेजी अनुक्रमण सक्षम बनाता है और 2014 के प्रकोप 2,4 दौरान इबोला वायरस क्रमबद्ध करने के लिए इस्तेमाल किया गया था। चयनात्मक कमी और tagmentation पुस्तकालय निर्माण के साथ सीडीएनए संश्लेषण युग्मन, समग्र प्रक्रिया में समय पिछले एडाप्टर बंधाव तरीकों से ~ 2 दिन की कमी थी। अभी हाल ही में इस प्रोटोकॉल बड़ी सफलता 15,16 के साथ अंतरराष्ट्रीय सहयोगियों और अन्य लोगों द्वारा नियोजित किया गया था और स्थानीय जीनोमिक्स आधारित अनुसंधान अध्ययन और निदान 17 समर्थन करने के लिए पश्चिम अफ्रीका में प्रयोगशालाओं के लिए तैनात किया जाएगा।
यहां वर्णित प्रोटोकॉल यादृच्छिक प्राइमरों का उपयोग करता वायरल शाही सेना seq पुस्तकालयों के लिए सीडीएनए तैयार करने के लिए। पिछले वायरल शाही सेना seq दृष्टिकोण के विपरीत, यह कोई एक विशिष्ट वायरस या क्लेड के लिए अनुक्रम डेटा या विस्तृत और समय लेने वाली प्रथम डिजाइन की एक प्राथमिकताओं ज्ञान की आवश्यकता है। विधि किसी भी वायरल शाही सेना के नमूने के लिए लागू किया जा सकता है। उदाहरण के लिए, यह दोनों इबोला से वायरल सामग्री उत्पन्न करने के लिए इस्तेमाल किया गया थाऔर लासा नमूने 6। प्रोटोकॉल भी मेजबान transcriptomic, metagenomic और रोगज़नक़ खोज अनुक्रमण परियोजनाओं 1 के लिए इस्तेमाल किया जा सकता है।
प्रोटोकॉल की एक महत्वपूर्ण कदम लक्षित है RNase एच पाचन, एक उच्च throughput, वायरल के नमूनों से अवांछित वाहक और मेजबान आरएनए को दूर करने के लिए कम लागत विधि। प्रोटोकॉल के चुनिंदा कमी कदम कई घटकों का उपयोग करता है और कौशल और सटीकता की आवश्यकता है। अतिरिक्त समय और देखभाल प्रारंभिक सेटअप के दौरान लिया जाना चाहिए।
के रूप में सबसे नैदानिक सीरम और प्लाज्मा नमूनों अक्सर बहुत कम न्यूक्लिक एसिड सामग्री है, प्रदूषण और नमूना नुकसान आम हैं। इन मुद्दों से बचने के लिए, विशेष ध्यान जब इस प्रोटोकॉल का उपयोग किया जाना चाहिए। सबसे पहले, आरएनए अत्यधिक गिरावट की संभावना है; इसलिए सभी क्षेत्रों को स्वच्छ और न्युक्लिअसिज़ से मुक्त होना चाहिए। दूसरा, इस प्रोटोकॉल में उपयोग के लिए उपयुक्त नमूनों की पहचान करने के लिए, दोनों मेजबान शाही सेना और वायरस के लिए QRT- पीसीआर assays मात्रा का ठहराव 5,6 के लिए इस्तेमाल किया जाना चाहिए </sup>। इनपुट की तुलना प्रोटोकॉल से अनुक्रमण परिणाम, अनुक्रमण सफलता के साथ मात्रा में जब (यानी, पूर्ण वायरल विधानसभा के लिए पर्याप्त डेटा की पीढ़ी) के नमूने है कि निहित कम से कम 100 पीजी कुल शाही सेना और वायरस के 1,000 प्रतियों के साथ सहसंबद्ध। तीसरा, न्यूक्लिक एसिड की पर्यावरण सूत्रों के जोखिम से बचा जाना चाहिए। यहाँ उल्लिखित प्रोटोकॉल सुरक्षा सावधानियों के लिए एक जैव सुरक्षा कैबिनेट में और पर्यावरण दूषित पदार्थों को सीमित करने के लिए किया जाता है। इसके अलावा, हमारे समूह और दूसरों को देखा है वाणिज्यिक एंजाइमों कम इनपुट नमूने में बैक्टीरिया 6,18 न्यूक्लिक एसिड को दूषित करने का एक और स्रोत हो सकता है। एक साफ कार्यक्षेत्र (जैसे, पीसीआर हुड, जैव सुरक्षा कैबिनेट) और नकारात्मक नियंत्रण का प्रयोग करें (जैसे, पानी या बफर) को कम करने में मदद करेगा और प्रदूषण को ट्रैक, क्रमशः। साथ नमूने के लिए <कुल शाही सेना के 100 पीजी, केवल पाली (आरए) वाहक शाही सेना, नहीं rRNA, उच्च गुणवत्ता अनुक्रमण परिणाम सुनिश्चित करने के लिए है, जबकि माल के नुकसान को सीमित करने से समाप्त किया जाना चाहिए। के लिए बहुतकम इनपुट के नमूने, सीडीएनए प्रवर्धन विधियों, अधिक उपयुक्त 19 हो सकता है, हालांकि पाली (आरए) वाहक सीडीएनए संश्लेषण से पहले हटा दिया जाना चाहिए।
मेजबान rRNA की कमी अनुक्रमण पुस्तकालयों में वायरल सामग्री के लिए समृद्ध करती और सीरम या प्लाज्मा, और मूषक और गैर मानव प्राइमेट 5,6 से ऊतकों के कई प्रकार सहित विभिन्न नमूना संग्रह करने के लिए लागू है। गैर मानव जीवों में, संरेखित पढ़ता 28S rRNA के लिए कमी के बाद बने रहे, सुझाव 28S rRNA मानव और अन्य प्रजातियों 6,20 के बीच कम संरक्षित है। गैर मानव वियोजन के साथ इस पद्धति का उपयोग करते हैं, यह विशिष्ट मेजबान 3,21 की विविधतावादी rRNA दृश्यों के लिए पूरक डीएनए ओलिगोस के साथ पूरक करने के लिए आवश्यक हो सकता है।
चूंकि प्रोटोकॉल निष्पक्ष है, वायरल केवल कुल पुस्तकालय सामग्री का एक छोटा सा अंश का प्रतिनिधित्व कर सकते पढ़ता है। हालांकि rRNA मेजबान शाही सेना के सबसे प्रचुर मात्रा में प्रजातियों और केवल rRNA का एक छोटा सा प्रतिशत पढ़ता है (0, 1%) पाए जाते हैं चयनात्मक कमी के बाद, अन्य सभी मेजबान आरएनए (जैसे, mRNA) की कमी के बाद बनी रहती है और कई अनुक्रमण के लिए नमूना से पढ़ता खाते सकता होगा। इसलिए "oversampling" (यानी, oversequencing) व्यक्तिगत पुस्तकालयों वायरल विधानसभा और संस्करण कॉल के लिए पर्याप्त कवरेज करने के क्रम में आवश्यक है। हमारे अध्ययन के लिए, हम अनुक्रम करने का प्रयास ~ 20 मिलियन वायरल जीनोमिक और संबद्ध वेरिएंट के विश्लेषण के लिए पर्याप्त गहराई के साथ ही metagenomic सामग्री 2,5 राशि के लिए नमूना प्रति पढ़ता है। metagenomic और रोगज़नक़ खोज पढ़ाई के लिए, यह ध्यान दें कि दूषित मेजबान डीएनए DNase पाचन द्वारा हटा दिया जाता है महत्वपूर्ण है। इसलिए वायरस और अन्य रोगाणुओं कि डीएनए जीनोम शामिल प्रक्रिया के दौरान खो दिया जा सकता है, तथापि आरएनए मध्यवर्ती अभी भी अनुक्रम किया जा सकता है।
Divulgations
The authors have nothing to disclose.
Acknowledgements
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
Materials
| 96-Well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µL, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5M NaCl | Ambion | AM9760G | |
| 1M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
References
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631 (2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882 (2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519 (2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- . Kapa Biosystems Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015)
- . Illumina Technologies Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf (2015)
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3 (2012).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, 713 (2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515 (2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19 (2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13 (2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).

