Klinik Örneklerden RNA Virüs tarafsız Derin Sıralama
Summary
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Abstract
Burada de novo meclisleri ve klinik ve biyolojik kaynaklardan toplanan viral genom içi konak varyant aramaları sağlayan yeni nesil RNA sıralama protokol özetlemektedir. yöntem tarafsız ve evrensel olduğu; Bu cDNA sentezi için, rastgele primerler kullanarak ve viral sekans içeriğine hiçbir ön bilgi gerektirir. dahil olmak üzere, poli (rA) taşıyıcı ve ribozomal RNA – – Viral RNA numunesinden kütüphane yapımı önce seçici RNase H tabanlı sindirim istenmeyen RNA tüketmek için kullanılır. Seçici tükenmesi veri kalitesini ve benzersiz sayısının viral RNA dizileme kütüphanelerde okur hem geliştirir. genel kütüphane oluşturma süresini azaltır Üstelik, bir transposaz dayalı 'tagmentation' aşamalı protokol kullanılır. protokol hızlı derin sıralama sağladı 600'ün üzerinde Lassa ve Ebola virüsü örnekleri dahil kan ve doku hem izolatları-ve diğer mikrobik genomik çalışmalar geniş çapta uygulanabilir tahsilatlar.
Introduction
Klinik kaynaklardan virüslerin Gelecek nesil dizileme iletimi ve enfeksiyonların epidemiyolojisi, yanı sıra teşhis yardım desteği romanı, aşı ve tedavi gelişimini bilgilendirebilir. rastgele primerler kullanılarak cDNA sentez farklı eş bulaşmaya genomları ya da yeni bir virüs 1,2 algılama ve montaj sağladı. diğer tarafsız yöntemlerinde olduğu gibi, istenmeyen kirletici birçok sıralama okur ve olumsuz sıralama sonuçlarını etkileyebilir işgal. Sunucu ve poli (rA) taşıyıcı RNA birçok mevcut viral örnek koleksiyonlarında mevcut kirletici maddelerdir.
protokol tarafsız toplam RNA seq dayalı derin sıralama RNA virüsüdür genom verimli ve maliyet-etkin bir şekilde açıklamaktadır. Yöntem istenmeyen konak ribozomal ve taşıyıcı RNA kaldırmak için bir RNase H seçici tükenmesi adımı 3 kullanır. Zayıflatılması viral içeriğine (Şekil 1) için zenginleştirir ve dizi verileri kalitesini artırırKlinik örneklerden (Şekil 2). Ayrıca, tagmentation önemli ölçüde kütüphane oluşturma süresini azaltır protokol uygulanır. Bu yöntemler, hızlı bir şekilde Ebola ve Lassa virüsü genomlarının 2,4,5 büyük veri kümelerini üretmek için kullanılmış ve RNA virüslerinin geniş bir çalışma için kullanılabilir. Son olarak, bu yaklaşım, insan örnekleri ile sınırlı değildir; zayıflatılması yardımcı Lassa enfekte kemirgen ve insan olmayan primat hastalık modellerinde 5,6 toplanan doku örnekleri üzerinde gösterilmiştir.
Şekil 1. Toplam RNA İçerik Zenginleştirme Seçici tükenmesi kullanarak Lassa Virüsü İçerik yansıtır. Dokuz farklı klinik izole edilen rRNA tükenmesi üzerine genel içerik (RNA girişi) ve benzersiz Lassa virüsü (LASV) zenginleşmesine okur (Kütüphane içerik) başlatılıyor. Bu Şekil 6, modifiye edilmiş <em>. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
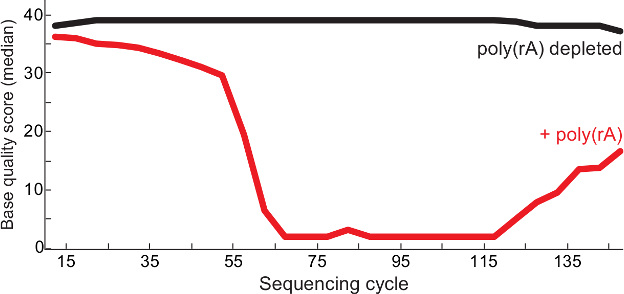
Şekil 2. Yüksek Kalitede Dizi Taşıyıcı RNA tükenmesi. Poli sıralama döngüsü (rA) başına medyan baz nitelikleri Lassa virüsü kitaplıkları (kırmızı) ve QC raporu 13 den kontrol (siyah kütüphanesinde gözlenen hiçbir taşıyıcı) Kontamine sonra. Hem 1 okumak ve eşleştirilmiş sonu 2 kitaplık BAM dosyasında birleştirilir okur ve kalite puanları her üssünde gösterilmiştir okuyun. Bu rakam 6'dan modifiye edilmiştir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
çıkarılan doğrudan kütüphanelerin viral RNA seq protokol detayları inşaatRNA klinik ve biyolojik örneklerden topladı. kişisel güvenliğini sağlamak için, tüm viral serum, plazma ve doku örnekleri RNA ekstraksiyonu öncesinde uygun tamponlar içinde inaktive edilmelidir. Bazı inaktivasyonu ve çıkarma kitleri, taşıyıcı poli (rA) RNA dahildir; Bu ilk RNase H zayıflatılması aşaması esnasında çıkarılır. tam iyileşme göre taşıyıcı RNA beklenen konsantrasyonu, 100 ng / ml. Protokolde, 110 ng / | il oligo dT RNA (1.1x taşıyıcı yoğunluğu) azalması için kullanılmaktadır. poli (rA), taşıyıcı numunede mevcut değilse, o zaman, oligo (dT) azalması önce ilave edilmemelidir.
Aşağıdaki protokol (250 ul hacim kadar) PCR levha formatında 24 reaksiyonlar için tasarlanmıştır. Bu protokolün bir önceki sürümü Matranga, vd bildirildi. 6..
Protocol
Representative Results
Discussion
Özetlenen yaklaşım sağlam, evrensel, hızlı sıralama sağlar ve 2014 mihrak 2,4 sırasında Ebola virüsü sıralanması için kullanılmıştır. tagmentation kütüphane yapımı seçici tükenmesi ve cDNA sentezi birleşerek, genel işlem süresi bir önceki bağdaştırıcı ligasyon yöntemleri ~ 2 gün kadar azaltılmıştır. Daha yakın zamanlarda, bu protokol büyük bir başarı 15,16 ile uluslararası işbirlikçileri ve diğerleri tarafından istihdam edildi ve yerel genomik tabanlı araştırmalar ve teşhis 17 desteklemek için Batı Afrika'da laboratuarlara görevlendirilecek.
Burada açıklanan protokol Viral RNA'ya DİZİ kütüphaneleri için cDNA hazırlamak için rastgele primerler kullanılır. Önceki Viral RNA'ya DİZİ yaklaşımlardan farklı olarak, hiçbir özel bir virüs ya da dalının için sekans verileri veya karmaşık ve zaman alıcı bir primer tasarımı hakkında önsel bir bilgi gerektirmektedir. yöntem, herhangi bir viral RNA numunesinin tatbik edilebilir. Örneğin, her iki Ebola viral içeriği oluşturmak için kullanılanve Lassa örnekleri 6. Protokolü ayrıca ana transcriptomic, metagenomic ve patojen bulma dizi projelerine 1 için de kullanılabilir.
protokol kritik bir safha, Rnaz H sindirim, yüksek verimli viral örnekleri istenmeyen bir taşıyıcı ve konakçı RNA çıkarmak için düşük maliyetli bir yöntemdir hedeflenmektedir. protokol seçici tükenmesi adımı birçok bileşenleri kullanır ve beceri ve doğruluk gerektirir. Ekstra zaman ve bakım ilk kurulum sırasında alınmalıdır.
çoğu klinik serum ve plazma numuneleri genellikle çok az nükleik asit malzeme var gibi, kirlenme ve örnek kaybı yaygındır. Bu protokolü kullanırken bu sorunları önlemek için, özel bakım alınmalıdır. İlk olarak, RNA ayrışma son derece duyarlı olduğu; Bu nedenle tüm alanları temiz ve nükleazlar arındırılmış olmalıdır. İkinci olarak, bu protokolde kullanılmak için müsait örnekleri belirlemek için, her iki ana RNA ve virüs için QRT-PCR miktarının 5,6 kullanılmalıdır </sus>. Giriş karşılaştıran protokolden sıralama sonuçları, sıralama başarı ile miktarlar zaman (yani, tam viral montaj için yeterli veri nesil) en az 100 pg toplam RNA ve virüsün 1,000 kopya içeren örnekleri ile ilişkili. Nükleik asitlerin, çevresel kaynaklardan Üçüncü olarak, maruz kaçınılmalıdır. Burada özetlenen protokol güvenlik önlemleri için bir biyogüvenlik kabini ve çevre kirleticileri sınırlamak için yapılır. Ayrıca, bizim grup ve diğerleri ticari enzimler düşük girdi örneklerinde 6,18 bakteriyel nükleik asitlerin kirletici başka bir kaynak olabileceğini fark etmiş. Temiz bir çalışma alanı (örneğin PCR kaput, biyogüvenlik kabini) ve negatif kontrollerin kullanımı (örneğin, su veya tampon), sırasıyla hafifletmek ve kirlenmeyi izlemenize yardımcı olacaktır. numuneler için <toplam RNA 100 ug, sadece poli (rA), taşıyıcı RNA değil, rRNA, malzeme kaybını sınırlarken, yüksek kaliteli dizileme sonuçları elde etmek için tükenmiş edilmelidir. çokpoli (rA), taşıyıcı cDNA sentezi önce çıkarılmalıdır, ancak düşük giriş örnekleri, cDNA amplifikasyon yöntemleri, 19 daha uygun olabilir.
Ev sahibi rRNA tükenmesi dizileme kütüphaneleri viral içeriğinden zenginleştiren ve serum veya plazma, kemirgenler ve insan olmayan primatlar 5,6 dokuların çok tip dahil olmak üzere farklı Numune alım için de geçerlidir. 28S rRNA tükenmesi sonra kalan insan olmayan organizmalar, rRNA, insan ve diğer türler 6,20 arasında daha az korunmuş bir 28S düşündürmektedir hizalama okunur. Insan olmayan izolatlar, bu yöntem kullanılarak, belirli bir ana bilgisayar 3,21 ıraksak rRNA dizilerine tamamlayıcı DNA oligo ile tamamlamak için gerekli olabilir.
Protokol tarafsız olduğu viral toplam kütüphanesi içeriğinin sadece küçük bir bölümünü temsil edebilir okur. rRNA en bol konak RNA türlerinin ve sadece rRNA küçük bir yüzdesini okur (olmasına rağmen0% 1), seçici yok olmasından sonra, tüm diğer konakçı RNA (örneğin mRNA) yok olmasından sonra kalır ve bir çok dizi örnek okur için hesap bulunur. Bu nedenle "örnekleme" (yani, oversequencing), ayrı ayrı kütüphaneler, viral düzeneği ve varyant aramalar için yeterli kapsama alanı elde etmek için gereklidir. Bizim çalışmaları için, biz dizisine ~ 20 milyon viral genomik ve ilişkili varyantların analizi için yeterli derinlik yanı sıra metagenomic içeriğin 2,5 olması numune başına okur girişimi. metagenomic ve patojen bulma çalışmaları için, konakçı DNA kirletici DNase sindirilerek kaldırılır dikkat etmek önemlidir. Bu nedenle DNA genomları ihtiva virüs ve diğer patojenleri sürecinde kaybolmuş olabilir, ancak RNA ara yine sıralı olabilir.
Divulgations
The authors have nothing to disclose.
Acknowledgements
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
Materials
| 96-Well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µL, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5M NaCl | Ambion | AM9760G | |
| 1M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
References
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631 (2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882 (2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519 (2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- . Kapa Biosystems Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015)
- . Illumina Technologies Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf (2015)
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3 (2012).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, 713 (2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515 (2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19 (2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13 (2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).

