Imitando la funzione di proteine di segnalazione: Verso artificiale trasduzione del segnale Therapy
Summary
We present guidelines for developing synthetic ‘chemical transducers’ that can induce communication between naturally unrelated proteins. In addition, detailed protocols are presented for synthesizing and testing a specific ‘transducer’ that enables a growth factor to activate a detoxifying enzyme and consequently, to regulate the cleavage of an anticancer prodrug.
Abstract
Signal transduction pathways, which control the response of cells to various environmental signals, are mediated by the function of signaling proteins that interact with each other and activate one other with high specificity. Synthetic agents that mimic the function of these proteins might therefore be used to generate unnatural signal transduction steps and consequently, alter the cell’s function. We present guidelines for designing ‘chemical transducers’ that can induce artificial communication between native proteins. In addition, we present detailed protocols for synthesizing and testing a specific ‘transducer’, which can induce communication between two unrelated proteins: platelet-derived growth-factor (PDGF) and glutathione-S-transferase (GST). The way by which this unnatural PDGF-GST communication could be used to control the cleavage of an anticancer prodrug is also presented, indicating the potential for using such systems in ‘artificial signal transduction therapy’. This work is intended to facilitate developing additional ‘transducers’ of this class, which may be used to mediate intracellular protein-protein communication and consequently, to induce artificial cell signaling pathways.
Introduction
Vie di trasduzione del segnale giocano un ruolo significativo praticamente in ogni processo cellulare e permettono alla cellula di rispondere rapidamente a segnali ambientali. 1 Questi percorsi sono spesso innescati dal legame di una molecola di segnalazione ad un recettore extracellulare, che si traduce in attivazione di enzimi intracellulari. Amplificazione e propagazione di questo segnale all'interno della cellula è mediata dalla funzione di segnalazione proteine che formano una rete di interazioni proteina-proteina in cui gli enzimi sono reversibilmente attivati con elevata specificità. A causa disregolazione di queste reti porta spesso allo sviluppo del cancro, c'è stato molto interesse a stabilire 'trasduzione del segnale terapia del cancro', 2 in cui i farmaci sono progettati per distruggere vie di segnalazione maligni. Abbiamo recentemente proposto un approccio alternativo per segnalare terapia trasduzione che si basa sulla capacità dei farmaci di generare trasduzione del segnale innaturali. <sup> 3 In particolare, riteniamo che progettando agenti sintetici che mimano la funzione di proteine di segnalazione, sarebbe possibile modulare la funzione della cellula indirettamente. Ad esempio, queste reti artificiali possono consentire proteine biomarcatori per attivare gli enzimi che solcano profarmaci. In alternativa, questi mimetici proteina di segnalazione potrebbero essere in grado di attivare vie di segnalazione cellulare innaturali, con conseguente effetti terapeutici.
Per dimostrare la fattibilità di questo approccio, abbiamo recentemente creato una sintetica 'trasduttore chimico' 4, che consente il fattore di crescita derivato dalle piastrine (PDGF) per attivare la scissione di un profarmaco antitumorale attivando glutatione-s-transferasi (GST), che è non il suo partner naturale vincolante. La struttura di questo 'trasduttore' costituito da un anti-PDGF aptamer DNA che viene modificato con un inibitore bivalente per GST. Quindi, questo agente sintetico appartiene ad una famiglia di molecole con i siti di legame perdiverse proteine, 5-7 come induttori chimici di dimerizzazione (CID) 8-10 e anche al gruppo di proteine-leganti basato su coniugati molecola oligonucleotidi sintetici. 11-21
I principi generali alla base della progettazione di tali sistemi sono descritti nel presente documento e protocolli dettagliati per sintetizzare e testare la funzione di questo 'trasduttore' con saggi enzimatici convenzionali sono forniti. Questo lavoro è destinato a facilitare lo sviluppo di "trasduttori" aggiuntivi di questa classe, che può essere usato per mediare comunicazione proteina-proteina intracellulare e conseguentemente, per indurre vie di segnalazione delle cellule artificiali.
La figura 1 descrive schematicamente i principi di funzionamento di sintesi 'trasduttori chimici' che possono mediare la comunicazione proteina-proteina innaturali. In questa illustrazione, un 'trasduttore chimica', che integra leganti sintetici per proteins I e II (leganti I e II), consente proteina II per innescare l'attività catalitica della proteina I, che non è il suo partner naturale vincolante. In assenza di proteine II, il trasduttore lega il sito catalitico dell'enzima (proteina I) e inibisce la sua attività (Figura 1, stato ii). Il legame del 'trasduttore' alle proteine II, tuttavia, promuove interazioni tra legante I e la superficie della proteina II (Figura 1, stato iii), che riduce l'affinità verso proteine I. Di conseguenza, la concentrazione efficace del ' libera trasduttore 'nella soluzione viene ridotta, il che porta alla dissociazione del trasduttore-proteina complesso I e riattivazione della proteina I (Figura 1, stato iv). Nel loro insieme, questi passi in evidenza tre principi fondamentali alla base della progettazione di "trasduttori 'efficienti: (1) un' sensore 'dovrebbe avere un legante specifico per ciascuno dei bersagli proteici, (2) l'interazione between legante II e proteine II dovrebbe essere più forte l'interazione tra binder I e proteine I, e (3) binder devo essere in grado di interagire con la superficie della proteina II. Quest'ultimo principio non richiede necessariamente che legante io solo avrebbe un alta affinità e selettività verso proteine II. Invece, si basa su nostri recenti studi che hanno dimostrato che portare una molecola sintetica in prossimità di una proteina è in grado di favorire interazioni tra questa molecola e la superficie della proteina. 19,22,23
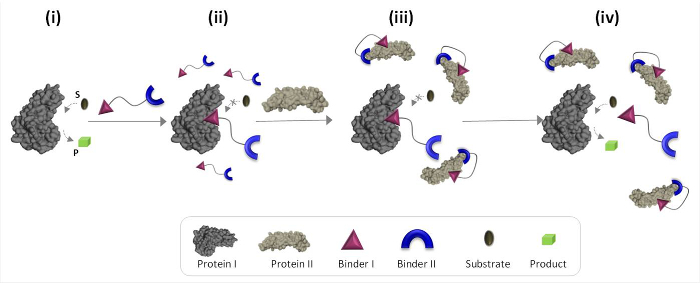
Figura 1:. Funzionamento principi di 'trasduttori chimici "Quando si aggiunge il' trasduttore chimica 'per una proteina attiva I (stato i), che si lega al suo sito attivo attraverso legante I e inibisce la sua attività (stato ii). In presenza di proteine II, tuttavia, la non legato 't chimicaransducer 'interagisce con proteine II tramite legante II, che promuove le interazioni tra legante I e la superficie della proteina II. Questo legante indotta I-II proteina interazione riduce la concentrazione effettiva di legante io, che porta alla dissociazione del 'transducer'-proteina complesso I e alle proteine I riattivazione (stato iv). Clicca qui per vedere una versione più grande di questa figura .
Protocol
Representative Results
Discussion
We presented a method for designing and testing of a ‘chemical transducer’ that can induce artificial communication between two naturally unrelated proteins, GST and PDGF, without modifying the native proteins. The unnatural GST-PDGF communications could be detected in real time by using enzymatic assays that follow the changes in the activity of GST in the presence of the ‘chemical transducer’ and increasing the concentrations of PDGF. In addition to detecting the activation of GST by PDGF, these assays were used to fol…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Questa ricerca è stata sostenuta dalla Fondazione Minerva, l'Organizzazione HFSP, e un Consiglio di Grant europeo della ricerca (Starting Grant 338.265).
Materials
| 1-chloro-2,4-dinitrobenzene | Sigma-Aldrich | 237329 | |
| Acetic acid | Bio Lab | 01070521 | |
| Acetnitrile | J.T.Baker | 9017-03 | |
| Ascorbic acid | Sigma-Aldrich | A4544 | |
| Copper(II) Sulfate pentahydrate | Merck-Millipore | 102790 | |
| Dimethyl sulfoxide | Merck-Millipore | 802912 | |
| Dulbecco's Phosphate Buffered Saline | Biological Industries | 02-023-5A | |
| Ethacrynic acid | Tokyo Chemical Industry Co. Ltd | E0526 | |
| Glutathione-s-transferase M1-1 | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| JS-K | Sigma-Aldrich | J4137 | |
| L-glutathione reduced | Sigma-Aldrich | G4251 | |
| Magnesium Chloride | J.T.Baker | 0162 | |
| nitrate/nitrite colorimetric assay kit | Cayman Chemical | 780001 | |
| Oligonucleotides | W. M. Keck Foundation Biotechnology at Yale University | custom order | |
| PDGF-BB | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| TBTA | Sigma-Aldrich | 678937 | |
| Triethylamine | Sigma-Aldrich | T0886 | |
| Desalting column | GE Healthcare | illustra MicroSpin G-25 Columns | |
| HPLC | Waters | 2695 separation module | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 4.6 mm × 50 mm) | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5μM, 10 mm × 50 mm) | |
| Plate reader | BioTek | synergy H4 hybrid |
References
- Hunter, T. Signaling—2000 and Beyond. Cell. 100, 113-127 (2000).
- Levitzki, A., Klein, S. Signal transduction therapy of cancer. Mol Aspects Med. 31, 287-329 (2010).
- Peri-Naor, R., Motiei, L., Margulies, D. Artificial signal transduction therapy: a futuristic approach to disease treatment. Future Med. Chem. 7, 2091-2093 (2015).
- Peri-Naor, R., Ilani, T., Motiei, L., Margulies, D. Protein-Protein Communication and Enzyme Activation Mediated by a Synthetic Chemical Transducer. J. Am. Chem. Soc. 137, 9507-9510 (2015).
- Corson, T. W., Aberle, N., Crews, C. M. Design and Applications of Bifunctional Small Molecules: Why Two Heads Are Better Than One. ACS Chem. Biol. 3, 677-692 (2008).
- Rutkowska, A., Schultz, C. Protein Tango: The Toolbox to Capture Interacting Partners. Angew. Chem. Int. Ed. 51, 8166-8176 (2012).
- Meyer, C., Köhn, M. A Molecular Tête-à-Tête Arranged by a Designed Adaptor Protein. Angew. Chem. Int. Ed. 51, 8160-8162 (2012).
- Klemm, J. D., Schreiber, S. L., Crabtree, G. R. Dimerization as a Regulatory Mechanism in Signal Transduction. Annu. Rev. Immunol. 16, 569-592 (1998).
- DeRose, R., Miyamoto, T., Inoue, T. Manipulating signaling at will: chemically-inducible dimerization (CID) techniques resolve problems in cell biology. Pflugers Arch. 465, 409-417 (2013).
- Gestwicki, J. E., Marinec, P. S. Chemical control over protein-protein interactions: beyond inhibitors. Comb. Chem. High Throughput. Screen. 10, 667-675 (2007).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. 25, 848-862 (2013).
- Diezmann, F., Seitz, O. DNA-guided display of proteins and protein ligands for the interrogation of biology. Chem. Soc. Rev. 40, 5789-5801 (2011).
- Röglin, L., Ahmadian, M. R., Seitz, O. DNA-Controlled Reversible Switching of Peptide Conformation and Bioactivity. Angew. Chem. Int. Ed. 46, 2704-2707 (2007).
- Röglin, L., Altenbrunn, F., Seitz, O. DNA and RNA-Controlled Switching of Protein Kinase Activity. ChemBioChem. 10, 758-765 (2009).
- Harris, D. C., Chu, X., Jayawickramarajah, J. DNA-Small Molecule Chimera with Responsive Protein-Binding Ability. J. Am. Chem. Soc. 130, 14950-14951 (2008).
- Harris, D. C., Saks, B. R., Jayawickramarajah, J. Protein-Binding Molecular Switches via Host-Guest Stabilized DNA Hairpins. J. Am. Chem. Soc. 133, 7676-7679 (2011).
- Kim, Y., Cao, Z., Tan, W. Molecular assembly for high-performance bivalent nucleic acid inhibitor. Proc. Nat. Acad. Sci. U.S.A. 105, 5664-5669 (2008).
- Han, D., et al. A Logical Molecular Circuit for Programmable and Autonomous Regulation of Protein Activity Using DNA Aptamer-Protein Interactions. J. Am. Chem. Soc. 134, 20797-20804 (2012).
- Motiei, L., Pode, Z., Koganitsky, A., Margulies, D. Targeted Protein Surface Sensors as a Tool for Analyzing Small Populations of Proteins in Biological Mixtures. Angew. Chem. Int. Ed. 53, 9289-9293 (2014).
- Ranallo, S., Rossetti, M., Plaxco, K. W., Vallée-Bélisle, A., Ricci, F. A Modular, DNA-Based Beacon for Single-Step Fluorescence Detection of Antibodies and Other Proteins. Angew. Chem. Int. Ed. 54, 13214-13218 (2015).
- Franzini, R. M., et al. Identification of Structure-Activity Relationships from Screening a Structurally Compact DNA-Encoded Chemical Library. Angew. Chem. Int. Ed. 54, 3927-3931 (2015).
- Unger-Angel, L., et al. Protein recognition by bivalent, ‘turn-on’ fluorescent molecular probes. Chem. Sci. 6, 5419-5425 (2015).
- Nissinkorn, Y., et al. Sensing Protein Surfaces with Targeted Fluorescent Receptors. Chem. Eur. J. 21, 15981-15987 (2015).
- Huber, C. G., Oefner, P. J., Bonn, G. K. High-Resolution Liquid Chromatography of Oligonucleotides on Nonporous Alkylated Styrene-Divinylbenzene Copolymers. Anal. Biochem. 212, 351-358 (1993).
- Lyon, R. P., Hill, J. J., Atkins, W. M. Novel class of bivalent glutathione S-transferase inhibitors. Biochimie. 42, 10418-10428 (2003).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. , 1-16 (2013).

