단백질 - 단백질 상호 작용을 연구하는 도구로 형광 이방성
Summary
단백질 상호 작용은 세포의 기능의 핵심이다. 열량 및 분광 기술은 일반적으로 그 특성을하는 데 사용됩니다. 여기에서 우리는 Shwachman – 다이아몬드 증후군 (SBDS)에 돌연변이 단백질과 신장 인자 같은 1는 GTPase (EFL1) 사이의 상호 작용을 연구하는 도구로 형광 이방성을 설명합니다.
Abstract
단백질 – 단백질 상호 작용은 살아있는 유기체의 기능에 필수적인 역할을한다. 상호 작용이 식별되고 확인되면 그 구조 및 기계적인 레벨을 특성화하는 것이 필요하다. 여러 생화학 및 생물 물리학 적 방법은 이러한 목적을 위해 존재한다. 형광 표지 된 단백질의 형광 강도가 단백질 – 단백질 상호 작용에 따라 일정하게 유지 될 때 그 중에서도 형광 이방성 특히 사용하는 강력한 기술이다. 이 방법에서, 형광 표지 된 단백질을 선택적으로 수신 빔의 상대적인 방향에 따른 형광체의 일부를 여기 적절한 파장의 수직 편광 된 광에 의해 여기된다. 다음과 같이 생성 된 배출량은 누구의 수직 및 수평 평면에 관계 이방성 (r)을 정의하는 방향성을 가지고 : r은 (I VV -I VH가) / (I VV + 2I VH), 나는 절과 I = 위치 <sUB> VH는 각각 수직 및 수평 성분의 형광 강도이다. 형광 이방성 형광 단백질 – 단백질 상호 작용에 따라 변경되는 단백질에 부착 된 형광 물질, 즉 겉보기 분자 크기의 회전 확산 민감하다. 본 텍스트에서, 단백질 – 단백질 상호 작용을 연구 도구로서 형광 이방성의 사용은 상기 Shwachman – 다이아몬드 증후군 (SBDS) 돌연변이 단백질 및 연장 인자와 같은-1는 GTPase (EFL1) 간의 바인딩을 해결하기 위해 예시 하였다. 종래, 형광 물질로 표지 단백질은 티올 기 (시스테인) 또는 단백질의 아미노 그룹 (N- 말단 아민 또는 리신)에서 수행된다. 그러나 SBDS 그것의 사이트에 직접 표시를 허용하지 않았다 여러 시스테인과 라이신을 보유하고있다. 대안 기술 염료 4 '로, 5'- 비스 (1,3,2 dithioarsolan -2- 일) – 플루오 레세 인은 특별히 tetracysteine 모티브 Cys- 라벨을 사용한시스테인 – 프로의 Gly-Cys 또는 Cys이고-는 유전자 재조합 SBDS 단백질의 C 말단에 설계. 실험 데이터의 피팅은이 단백질 사이의 결합 모드에 양적 기계적인 정보를 제공합니다.
Introduction
세포는 끊임없이 서로 상호 작용 생체 거대 분자의 다수 포함되어 있습니다. 이 연관은 신호 전달, 유전자 발현 및 다른 이들 세포 이동의 조절에서 이들의 기능을 담당 세포 경로에 참여 착물을 일으킨다. 세포 내에서 발생하는 모든 단백질 – 단백질 상호 작용은 상호 작용 체라고하는 네트워크를 포함한다. 사카로 마이 세스 세레 비지에서의 단백질의 70 % 이상이 상호 작용 파트너 일을하는 것으로 나타났다. 셀의 상호 작용 체를 이해하고 그 기능은 복잡성과 생물의 다양성에 관련 정보를 제공합니다. 여러 방법은 단백질 – 단백질 상호 작용을 확인하고 특성화하기 위해 설명되었다. 효모 두 하이브리드 2, 단백질 단편 보완 분석 (3), 질량 분석 및 단백질 microarra에 결합 친 화성 정제 4로 넣어 방법을 통해 높은 다른YS는 5,6- 작용을 식별하기 위해 사용된다. 식별되면,이를 검증하기 위해 필요하며, 이는 경우에 따라 달라질 수있다. 일반적으로, 이들 실험은 유전자 결실 또는 단백질 중 하나의 과다 발현에 의해 상호 작용 쌍 등의 각 부재의 수준에서 작용 자체를 방해 포함하고 상기 다른 부재의 특성의 변화 나 함수를 찾고 세포 수준. 이어서, 생물 리 학적 기법 7은 분자 수준에서 단백질 – 단백질 상호 작용을 특성화하기 위해 사용된다. 열량 형광 분광법 정량적 및 기계적으로이를 설명하기 위해 사용되는 동안이를 위해, 단백질 복합체의 구조는 X 선 결정학, 핵 자기 공명 및 극저온 전자 현미경에 의해 결정된다.
본 연구에서는 형광의 이방성이는 GTPase EFL1 및 SBD 간의 상호 작용을 특성화하는 방법을 사용 하였다S 단백질. 이 단백질은 60S 리보솜 서브 유닛 (8)의 표면에서 진핵 개시 인자 (6)의 방출을 촉진하여 리보솜의 합성에 참여하고 있습니다. SBDS 단백질 EFL1는 구아노 신 다이 포스페이트 (10, 11)에 대한 그것의 친화도를 감소시키는 구아닌 뉴클레오타이드 교환 인자로서 Shwachman 증후군 다이아몬드-9 및 행위로 알려진 질환 돌연변이된다. SBDS의 질병 돌연변이는 EFL1과의 상호 작용을 폐지하고, 따라서 그것의 활성화를 방지 할 수 있습니다.
형광의 이방성은 일반적으로 단백질 – 펩타이드 또는 단백질 – 핵산 상호 작용을 연구하는 생물학 분야에 사용된다. 이것은 형광 물질이 부분적으로 편광 된 방사의 편광 결과 여기 있다는 원리에 기초한다. 형광 이방성은 수학 식 1에 의해 정의된다 :
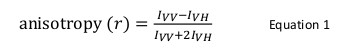
나는 절 내가 VH는 어디에수직 편광 된 광 (12)에 시료가 여기 될 때 수직 (VV) 및 가로 (VH)의 형광 발광 강도를 편광. 형광 이방성 형광의 회전 확산 속도에 영향을주는 요인에 민감하므로, 온도, 용액의 점도 및 형광 물질의 겉보기 분자 크기에 따라 달라진다. 또 다른 단백질과 같은 변화와 상호 작용할 때 형광 증가를 함유하는 단백질의 겉보기 크기이어서 이방성의 변화로 평가 될 수있다. 구체적으로는, 그것의 형광 수명을 용액 상대적으로 천천히 회전 형광 비교적 큰 이방성을 나타내는 것 때문에 큰 I VV의 값 I VH 작은 값을 가질 것이다. 자신의 형광 수명에 빠르게 상대 공중제비 형광, 나는 절 내가 VH는 유사합니다 및 이방성 값이 작은 것 (12) </sup> (그림 1). 또한, 노이즈 측정에 좋은 이방성 신호를 들면, 대상 분자의 회전 상관 시간 유사한 형광 수명을 가진 형광 물질이 필요하다. 그렇지 않으면 정확하게 복잡 프리 단백질과 그 이방성의 차이를 기록 할 수 없다. 예를 들어, 다 100의 저 분자량 화합물에 부착 같은 형광 또는 로다 민과 같은 4 NSEC 가까운 수명과 형광 프로브의 이방성은 0.05이다. kDa의 0.29로 그 이방성 값을 증가 (160)의 분자에 결합; 정확하게 측정 할 수있는 차이. 반면에, 그 증가 분자 크기가 변화 65 내지 1,000 kDa의 결합 반응에 관여 동일한 형광 프로브는 정확하게 측정하기에는 너무 작 0.3-0.28의 이방성이 변화 될 것이다. 이 시나리오에서 400 나노초의 수명이 프로브 (12)에 더 적합 할 것이다.
<p클래스 = "jove_content">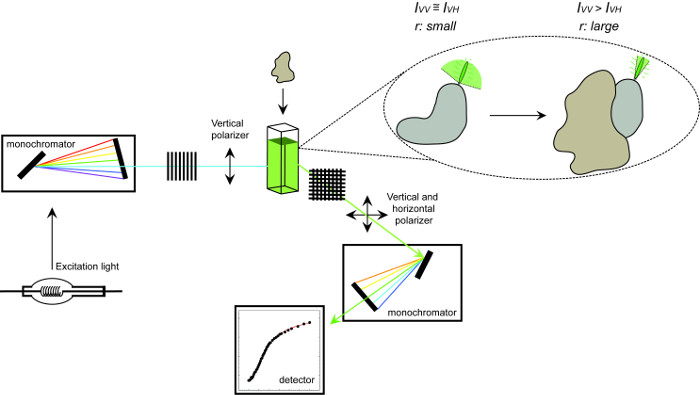
형광 이방성 절차를 측정하는 데 사용되는 장치의 개략도 1.도. 단백질 – 단백질 상호 작용의 실험 측정 형광 이방성을 수행하는 데 사용되는 장치의 개략도. 상호 작용 파트너의 결합에 증가 빠른 디스플레이 작은 이방성을 공중제비 형광. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
형광 응용 연구 분자 중 어느 하나의 형광의 존재를 필요로한다. 형광의 세 가지 유형이 단백질 – 단백질 상호 작용을 연구하는 방법 : 1) 단백질에 존재하는 트립토판 잔류 물, 2) 녹색 형광 단백질 (GFP)와 deriva 같은 화학적으로 부착 된 형광 3) 형광 융합 파트너tives. 대부분의 단백질은 그 구조의 상호 작용을 측정하는 것이 가장 간단한 방법 대응 형광 스펙트럼의 변화를 모니터링함으로써 또는 트립토판 잔기의 형광 강도의 변화를 감시함으로써이다 트립토판 잔기를 갖는다. 그러나, 트립토판 잔기의 분석을 복잡 단백질 모두에 존재할 수있다. 형광 인한 상호 작용을 형광의 속성을 변경하는 한편, 또는 결합 부위 근처에 위치해야하며 상호 자체를 방해 할 수있다. 같은 GFP와 같은 부피가 큰 형광체를 사용할 때 특별한주의가 필요합니다. 이 형광체 중에 결합 연구에 사용 할 수없는 경우는, 관여하는 단백질의 하나 외인성 형광체를 도입 할 필요가있다. 많은 화학적으로 합성 된 형광체는 존재 공유 같은 아민 기 (라이신 또는 N 말단의 측쇄) 및 시스테인의 티올기를 그들의 반응성기를 통해 단백질에 부착 될 수있다. 에프요오도 아세트 아미드 및 말레이 미드가 티올 – 반응성 기 (13) 동안 이소 티오 시아 네이트 및 숙신 이미 딜 에스테르로 luorophore 유도체 아미드 기와 반응. 형광 애플리케이션에서 가장 일반적으로 사용되는 염료는 플루오 레세 인 유도체 및 로다 민 그린 염료, 쿠마린, BODIPY 형광 단 알렉사 형석 염료이다. 상업적으로 입수 가능한 형광체와 그 사용의 자세한 목록은 참고 문헌 14, 15에서 볼 수있다. 성공적인 라벨링, 반응성 그룹은 단백질 표면에 노출되어야하지만 의한 폴리펩티드 전형적으로 존재하는 반응성 관능기의 다수는 부위 특이 적 변형을 얻는 것은 매우 어렵다. 이 연구에 대한 관심의 단백질, SBDS, 여러 사이트 표시 될 수 있습니다 5 무료 시스테인, 33 라이신이 포함되어 있습니다. 불균일 한 표시는 결합에 영향을 미칠 수있는 다른 형광 분자가 결합에 따라 상이한 형광 강도 신호를 유도 할 수있다으로 데이터 분석을 복잡하게한다. 비켜하려면이 문제를 rcome 우리는 플래시 형광, 4 '부위 – 직접 라벨, 5'- 비스 (1,3,2 dithioarsolan -2- 일) – 플루오 레세 SBDS 단백질을 사용 하였다. 이 모티프는 X 시스테인 (16, 17) 이외의 아미노산 서열로 이루어진 플래시 CCXXCC 태그로 알려진 네 개의 이격 시스테인에 대한 높은 친 화성을 갖는 arsenoxide 염료이다. 이 tetracysteine 모티브 함께 단백질의 전체 스크롤의 중단을 방지하기 위해 적당한 링커로 유전 공학에 의해 N- 또는 단백질의 C 말단에 부가된다. 플래시 염료 및 플래시 태그 이루어지는 쌍은 원래 셀 17 거주 지역 사이트 별 라벨 단백질 설계되었지만, 또한 여기에 열거 된 바와 같이 시험 관내에서 정제 된 단백질의 라벨을 사용할 수있다. 또한, 효소 적 전략은 단백질 (18)의 부위 특이 적 작용을 가능하게하기 위해 개발되어왔다.
이 논문에서는 형광 이방성 (A)의 유용성을 설명SA 도구는 단백질 – 단백질 상호 작용을 연구합니다. 정량적 인 정보는 실험 데이터의 피트로부터 얻어 질 수있는 반면 결합은 결합 곡선 형상의 간단한 검사에 의해 평가 될 수있다.
Protocol
Representative Results
Discussion
단백질과 대부분의 생화학 실험에 관계없이 사용되는 기술, 순수 단백질뿐만 아니라, 그들 중 많은 양뿐만 아니라 필요합니다. 그것은 여기에 제시된 경우와 같이이 때문에, 실험의 이러한 유형에 사용되는 단백질은, 이종 발현에 의해 얻어진다. 개화 분광법은 공부 분자에 형광의 존재를 필요로한다. 이들은 대부분의 단백질에 공통적 모두에 존재할 것이기 때문에, 방향족 잔기가 단백질 – 단백?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Authors acknowledge the financial support from CONACyT project numbers 167359 and 177138, and from DGAPA-UNAM project number IN201615.
Materials
| 0.5 mm Glass beads | Biospec Products | 11079105 | |
| Tris Base | Formedium | TRIS01 | Ultra pure |
| Glycerol | Sigma-Aldrich | G5516 | |
| dye 4’, 5’-bis(1,3,2 dithioarsolan-2-yl) fluorescein | ThermoFischer Scientific | LC6090 | This kit contains the dye to label a FlAsH tag |
| Ampiciline | IBI Shelton Scientific, Inc | IB02040 | |
| D(+)-Glucose Anhydrous | Formedium | GLU03 | |
| D(+)-Galactose | Formedium | GAL03 | |
| L-Leucine | Formedium | DOC0157 | |
| L-Tryptofan | Formedium | DOC0189 | |
| Bezamidine hydrochloride | Sigma-Aldrich | B6506-5G | |
| PMSF | Gold Biotechnology, Inc | P-470-25 | Phenylmethylsulfonyl fluoride |
| NaCl | Formedium | NAC02 | Sodium Chloride |
| Glycerol | Tecsiquim, S.A. de C.V. | GT1980-6 | |
| MgCl2 | Merck Millipore Corporation | 1725711000 | Magnesium Chloride |
| Imidazole | Sigma-Aldrich | I2399-500G | |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-100ML | |
| K2HPO4 | Sigma-Aldrich | P3786-500G | Potassium phosphate dibasic |
| NaH2PO4 | Sigma-Aldrich | S3139-500G | Sodium phosphate monobasic |
| Yeast nitrogen base without amino acids | Formedium | CYN0410 | |
| Yeast extract | Formedium | YEM03 | Micro Granulated |
| L-Tyroisne | Formedium | DOC0193 | |
| Adenine sulphate | Formedium | DOC0230 | |
| Casamino acids | Formedium | CAS03 | |
| Tryptone | IBI Shelton Scientific, Inc | IB49182 | |
| IPTG | Formedium | IPTG025 | |
| Name of Material | Company | Catalog Number | Comments/Description |
| Filtration units | Merck Millipore Corporation | UFC901096 | Amicon Ultra-15, membrana PLGC Ultracel-PL, 10 kDa |
| Membrane Filter | Merck Millipore Corporation | GSWP04700 | Membrane Filter, mixed cellulose esters, Hydrophilic, 0.22 µm, 47 mm, white, plain |
| Ni2+ affinity column | QIAGEN | 30760 | Cartridge pre-filled with 5 ml Ni-NTA Superflow |
| Strong Sulfopropyl cation exchanger column | GE Healthcare Life Science | 17-5157-01 | HiTrap SP Sepharose FF 5 ml |
| Size Exclusion column | GE Healthcare Life Science | 28989335 | HiLoad 16/600 Superdex 200 PG |
| Fluorescence cell | Hellma Analytics | 111-057-40 | |
| Name of Equipment | Company | Catalog Number | Comments/Description |
| Spectrophotometer | Agilent Technologies | G6860AA | Cary 60 UV-Vis |
| Shaker | ThermoFischer Scientific | SHKA4000-7 | MaxQ 4000 Benchtop temperature range Ambient-15° to 60°C |
| Centrifuge | ThermoFischer Scientific | 75004271 | Heraeus Megafuge 16R |
| FPLC | Pharmacia Biotech | Discontinued | FPLC system conductivity UV-MM II monitor P500 pump fraction |
| Spectrofluorometer | Olis | No applicable | Olis DM 45 with Polarization Toolbox |
References
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440 (7084), 637-643 (2006).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Michnick, S. W., Hien Ear, P., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods in Enzymology. 470, 336-366 (2010).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Dwane, S., Kiely, P. A. Tools used to study how protein complexes are assembled in signaling cascades. Bioeng Bugs. 2 (5), 247-259 (2011).
- Snider, J., et al. Fundamentals of protein interaction network mapping. Mol Syst Biol. 11 (12), 848 (2015).
- Fersht, A., Baldwin, R. L. . Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. , 191-214 (2002).
- Menne, T. F., et al. The Shwachman-Bodian-Diamond syndrome protein mediates translational activation of ribosomes in yeast. Nat Genet. 39 (4), 486-495 (2007).
- Boocock, G. R., et al. Mutations in SBDS are associated with Shwachman-Diamond syndrome. Nat Genet. 33 (1), 97-101 (2003).
- Garcia-Marquez, A., Gijsbers, A., de la Mora, E., Sanchez-Puig, N. Defective Guanine Nucleotide Exchange in the Elongation Factor-like 1 (EFL1) GTPase by Mutations in the Shwachman-Diamond Syndrome Protein. J Biol Chem. 290 (29), 17669-17678 (2015).
- Gijsbers, A., Garcia-Marquez, A., Luviano, A., Sanchez-Puig, N. Guanine nucleotide exchange in the ribosomal GTPase EFL1 is modulated by the protein mutated in the Shwachman-Diamond syndrome. Biochem Biophys Res Commun. 437 (3), 349-354 (2013).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2010).
- Nishigaki, T., Treviño, C. L., Gòmez, I. . Tools to understand protein-protein interactions. 37, 1-14 (2012).
- Johnson, I. . The Molecular Probes Handbook: A Guide to Fluorescent Probes and Labeling Technologies. , (2010).
- Sabnis, R. W. . Handbook of Fluorescent Dyes and Probes. , (2015).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: synthesis and biological applications. J Am Chem Soc. 124 (21), 6063-6076 (2002).
- Griffin, B. A., Adams, S. R., Tsien, R. Y. Specific covalent labeling of recombinant protein molecules inside live cells. Science. 281 (5374), 269-272 (1998).
- Rashidian, M., Dozier, J. K., Distefano, M. D. Enzymatic labeling of proteins: techniques and approaches. Bioconjug Chem. 24 (8), 1277-1294 (2013).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: A laboratory manual. , (2001).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- West, R. W., Chen, S. M., Putz, H., Butler, G., Banerjee, M. GAL1-GAL10 divergent promoter region of Saccharomyces cerevisiae contains negative control elements in addition to functionally separate and possibly overlapping upstream activating sequences. Genes Dev. 1 (10), 1118-1131 (1987).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Eftink, M. R. Fluorescence methods for studying equilibrium macromolecule-ligand interactions. Methods Enzymol. 278, 221-257 (1997).
- Han, H., et al. Binding of Substrates to the Central Pore of the Vps4 ATPase Is Autoinhibited by the Microtubule Interacting and Trafficking (MIT) Domain and Activated by MIT Interacting Motifs (MIMs). J Biol Chem. 290 (21), 13490-13499 (2015).
- Sanchez-Puig, N., Veprintsev, D. B., Fersht, A. R. Binding of natively unfolded HIF-1alpha ODD domain to p53. Mol Cell. 17 (1), 11-21 (2005).
- Trusch, F., et al. The N-terminal Region of the Ubiquitin Regulatory X (UBX) Domain-containing Protein 1 (UBXD1) Modulates Interdomain Communication within the Valosin-containing Protein p97. J Biol Chem. 290 (49), 29414-29427 (2015).
- Kamp, F., Beyer, K. Binding of alpha-synuclein affects the lipid packing in bilayers of small vesicles. The Journal of Biological Chemsitry. 281, 9251-9259 (2006).
- Bujalowski, W. M., Jezewska, M. J. Fluorescence Intensity, Anisotropy, and Transient Dynamic Quenching Stopped-Flow Kinetics. Spectroscopic Methods of Analysis. 875, 105-133 (2012).
- Asano, N., et al. Direct interaction between EFL1 and SBDS is mediated by an intrinsically disordered insertion domain. Biochem Biophys Res Commun. 443 (4), 1251-1256 (2014).

