La anisotropía de fluorescencia como una herramienta para estudiar las interacciones proteína-proteína
Summary
interacciones de proteínas están en el corazón de la función de la célula. técnicas calorimétricas y espectroscópicas son comúnmente utilizados para caracterizarlos. Aquí describimos anisotropía de fluorescencia como una herramienta para estudiar la interacción entre la proteína mutada en el síndrome de Shwachman-Diamond (SBDS) y el factor de elongación 1 similar GTPasa (EFL1).
Abstract
Las interacciones proteína-proteína desempeñan un papel esencial en la función de un organismo vivo. Una vez que una interacción ha sido identificado y validado es necesario caracterizar en el nivel estructural y mecánica. Existen varios métodos bioquímicos y biofísicos para tal fin. Entre ellos, anisotropía de fluorescencia es una técnica poderosa particularmente cuando se utiliza la intensidad de fluorescencia de una proteína marcada con fluoróforo permanece constante tras la interacción proteína-proteína. En esta técnica, una proteína marcada con fluoróforo se excita con luz polarizada verticalmente de una longitud de onda apropiada que excita selectivamente un subconjunto de los fluoróforos en función de su orientación relativa con el haz entrante. La emisión resultante también tiene una direccionalidad cuya relación en los planos vertical y horizontal define la anisotropía (r) como sigue: r = (I VV -I VH) / (I + 2I VV VH), donde I VV y yo <sub> VH son las intensidades de fluorescencia de los componentes verticales y horizontales, respectivamente. La anisotropía de fluorescencia es sensible a la difusión rotacional de un fluoróforo, es decir, el tamaño molecular aparente de un fluoróforo unido a una proteína, que se altera tras la interacción proteína-proteína. En el presente texto, el uso de la anisotropía de fluorescencia como una herramienta para estudiar las interacciones proteína-proteína se ejemplifica para hacer frente a la unión entre la proteína mutada en el síndrome de Shwachman-Diamond (SBDS) y el factor de elongación como-1 GTPasa (EFL1). Convencionalmente, el etiquetado de una proteína con un fluoróforo se lleva a cabo en los grupos tiol (cisteína) o en los grupos amino (N-terminal de amina o de lisina) de la proteína. Sin embargo, SBDS posee varias cisteínas y lisinas que no permitían sitio etiquetado dirigido de la misma. Como una técnica alternativa, el colorante 4 ', 5'-bis (1,3,2 dithioarsolan-2-il) fluoresceína se utiliza para etiquetar específicamente un motivo de tetracisteína, Cys-Cys-Pro-Gly-Cys-Cys, ingeniería genética en el C-terminal de la proteína recombinante SBDS. El ajuste de los datos experimentales proporcionan información cuantitativa y mecanicista en el modo de unión entre estas proteínas.
Introduction
Las células contienen una multitud de biomacromoléculas que interactúan constantemente entre sí. Esta asociación da lugar a complejos que participan en las vías celulares responsables de su funcionamiento en la transducción de señales, regulación de la expresión génica y la migración celular entre otros. Todas las interacciones proteína-proteína que se producen en una célula comprenden una red conocida como el interactome. En Saccharomyces cerevisiae más de 70% de sus proteínas se ha demostrado que tiene que interactúan socios 1. La comprensión de la interactoma de una célula y sus funciones proporcionar información relevante sobre la complejidad y la diversidad de los organismos vivos. Varios métodos han sido descritos para identificar y caracterizar las interacciones proteína-proteína. Diferentes alta a través de métodos tales como poner dos híbridos de levadura 2, ensayos de complementación de proteínas fragmento 3, 4 purificación por afinidad acoplada a espectrometría de masas y microarra proteínasys se utilizan para identificar una interacción 5,6. Una vez identificados, es necesario para validar y esto puede variar sobre una base de caso por caso. Típicamente, estos experimentos implican la interrupción de la interacción en sí misma en el nivel de los miembros individuales de la pareja de interacción, por ejemplo, por deleción del gen o la sobreexpresión de una de las proteínas y, a continuación en busca de cambios en las propiedades o la función del otro miembro en el nivel celular. Posteriormente, técnicas biofísicas 7 se utilizan para caracterizar la interacción proteína-proteína en el nivel molecular. Con este fin, la estructura de los complejos de proteínas se determinó por cristalografía de rayos X, resonancia magnética nuclear y crio-microscopía electrónica mientras calorimetría y espectroscopía de fluorescencia se utilizan para describir cuantitativamente y de manera mecánica.
En este trabajo, la anisotropía de fluorescencia se usó como una técnica para caracterizar la interacción entre la GTPasa EFL1 y el SBDproteína S. Estas proteínas participan en la síntesis de los ribosomas mediante la promoción de la liberación del factor de iniciación eucariótico 6 de la superficie de las subunidad 60S ribosomal 8. La proteína SBDS está mutado en una enfermedad conocida como el síndrome de Shwachman-Diamond 9 y actúa como un factor de intercambio de nucleótidos de guanina para EFL1 disminuyendo su afinidad por difosfato de guanosina 10,11. mutaciones de la enfermedad en SBDS abolir la interacción con EFL1 de materiales, evitando su activación.
La anisotropía de fluorescencia se utiliza comúnmente en aplicaciones biológicas para estudiar la proteína-péptido o interacciones proteína-ácido nucleico. Se basa en el principio de que un fluoróforo excitado con los resultados de luz polarizada en una emisión parcialmente polarizada. La anisotropía de fluorescencia se define por la Ecuación 1:
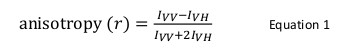
donde VV y VH son laintensidades de fluorescencia de la vertical (VV) y horizontalmente (VH) de emisión polarizadas cuando la muestra se excita con luz polarizada verticalmente 12. La anisotropía de fluorescencia es sensible a los factores que afectan a la tasa de la difusión rotacional del fluoróforo y por lo tanto depende de la temperatura, la viscosidad de la solución y el tamaño molecular aparente del fluoróforo. El tamaño aparente de una proteína que contiene un fluoróforo aumenta cuando interactúa con otra proteína y tal cambio, entonces se puede evaluar como un cambio en la anisotropía. Más específicamente, un fluoróforo que gira lentamente en solución con respecto a su tiempo de vida fluorescente tendrá un valor grande I VV y valor pequeño I VH y por lo tanto exhibirá un relativamente grande anisotropía. Para fluoróforos que caen rápidamente en relación a su tiempo de vida fluorescente, me VV y VH será similar y su valor será pequeña anisotropía 12 </sup> (Figura 1). Además, para una buena señal de anisotropía para la medición del ruido, es necesario disponer de un fluoróforo con una vida de fluorescencia similar al tiempo de correlación rotacional de la molécula de interés. De lo contrario, no es posible registrar con precisión la diferencia de anisotropía entre la proteína libre y que en el complejo. Por ejemplo, la anisotropía de una sonda fluorescente con una vida útil cerca de 4 nseg tales como fluoresceína o rodamina unidos a un compuesto de bajo peso molecular de 100 Da es 0,05. La unión a una molécula de 160 kDa aumentará su valor de anisotropía a 0,29; una diferencia que se puede medir con precisión. Por el contrario, la misma sonda fluorescente que participan en una reacción de unión cuyo aumento en el tamaño molecular varía de 65 a 1000 kDa sólo resultará en un cambio de anisotropía de 0,28 hasta 0,3, que es demasiado pequeña para ser medida con precisión. En este escenario, una sonda con un tiempo de vida de 400 nseg sería más adecuado 12.
<pclass = "jove_content">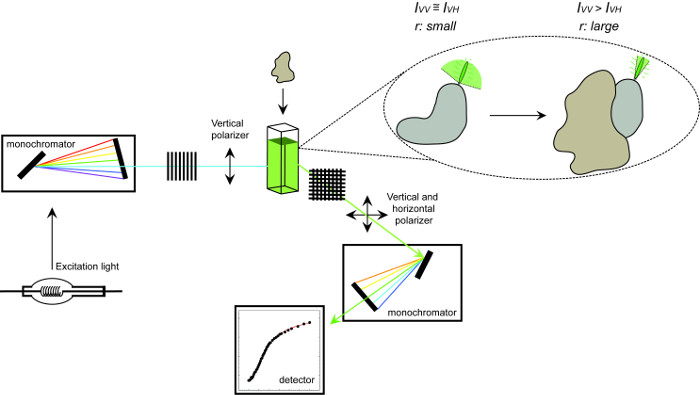
Figura 1. Representación esquemática del equipo utilizado para medir la anisotropía de fluorescencia y el procedimiento. Representación esquemática del equipo utilizado para realizar una medición de anisotropía de fluorescencia proteína-proteína experimento interacción. Fluoróforos que caen rápido pantalla pequeña anisotropía que aumenta tras la unión a un compañero de interacción. Haga clic aquí para ver una versión más grande de esta figura.
aplicaciones de fluorescencia requieren la presencia de un fluoróforo en cualquiera de las moléculas estudiadas. Para el estudio de las interacciones proteína-proteína hay tres tipos de fluoróforos: 1) los residuos de triptófano presentes en las proteínas, 2) fluoróforos unidos químicamente y 3) parejas de fusión fluorescentes tales como proteína verde fluorescente (GFP) y su derivatantes. La mayoría de las proteínas tienen residuos de triptófano en su estructura, por tanto, la manera más fácil de medir una interacción es mediante el control de los cambios en los espectros de fluorescencia correspondiente o mediante el control de los cambios en la intensidad de fluorescencia de los residuos de triptófano. Sin embargo, los residuos de triptófano pueden estar presentes en ambas proteínas que complican el análisis. Por otra parte, por un fluoróforo para cambiar sus propiedades fluorescentes debido a una interacción que tiene que estar situado en o cerca del sitio de unión y que podría interferir con la interacción en sí misma. Esto necesita una atención especial cuando se utiliza fluoróforos voluminosos tales como GFP. Si ninguno de estos fluoróforos se puede utilizar para estudios de unión es necesario, entonces, para introducir fluoróforos extrínsecas a la de las proteínas implicadas. Existen muchos fluoróforos sintetizados químicamente y pueden ser unidos covalentemente a las proteínas a través de sus grupos reactivos tales como los grupos amina (cadena lateral de lisinas o N-terminal) y los grupos tiol en cisteína. Fderivados luorophore con ésteres de isotiocianato y succinimidilo reaccionan con grupos amida mientras yodoacetamida y maleimida son grupos reactivos con tiol 13. Los colorantes más comunes utilizados en aplicaciones de fluorescencia son derivados de la fluoresceína y los colorantes rodamina verde, cumarinas, fluoróforos BODIPY y colorantes Alexa Fluor. Una lista detallada de fluoróforos disponibles en el mercado y su uso se puede encontrar en las referencias 14,15. Para el etiquetado de éxito, el grupo reactivo debe ser expuesto en la superficie de la proteína, pero debido a la gran cantidad de grupos funcionales reactivos típicamente presentes en polipéptidos que es muy difícil de conseguir la modificación específica de sitio. La proteína de interés en este estudio, SBDS, contiene 5 cisteínas libres y 33 lisinas que pueden resultar en el etiquetado de múltiples sitios. etiquetado no uniforme puede afectar a la unión y complicará el análisis de datos como diferentes moléculas de fluoróforo pueden provocar diferentes señales de intensidad de fluorescencia tras la unión. para overcome este problema, hemos utilizado el fluoróforo flash, 4 ', 5'-bis (1,3,2 dithioarsolan-2-il) fluoresceína a la etiqueta sitio directa la proteína SBDS. Este es un colorante de arsenóxido con una alta afinidad por cuatro cisteínas espaciadas en un motivo de saber como Flash-tag consiste en la secuencia CCXXCC donde X es cualquier aminoácido distinto de cisteína 16,17. Este motivo de tetracisteína se añade a la N- o C-terminal de la proteína por ingeniería genética junto con un enlazador adecuado para evitar la interrupción del pliegue general de la proteína. La pareja que consiste en FLASH tinte y Flash-tag fue diseñado originalmente para las proteínas de etiquetas específicas del lugar en las células 17 que vivían, pero también puede ser utilizada para etiquetar proteínas purificadas in vitro como se ejemplifica aquí. Además, las estrategias enzimáticos también se han desarrollado para permitir la funcionalización específica de sitio de proteínas 18.
En este manuscrito se describe la utilidad de anisotropía de fluorescencia de unaherramienta sa para estudiar las interacciones proteína-proteína. La unión puede evaluarse por simple inspección de la forma de la curva de unión mientras que la información cuantitativa puede obtenerse a partir del ajuste de los datos experimentales.
Protocol
Representative Results
Discussion
La mayoría de los experimentos bioquímicos con proteínas requieren no sólo proteína pura, sino también grandes cantidades de ellos, independientemente de la técnica utilizada. Por esta razón, las proteínas utilizadas para este tipo de experimentos se obtienen mediante la expresión heteróloga, como era el caso que aquí se presenta. espectroscopia Florescence requiere la presencia de un fluoróforo en la molécula estudiada. residuos aromáticos constituyen los fluoróforos intrínsecos de una proteína, sin e…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Authors acknowledge the financial support from CONACyT project numbers 167359 and 177138, and from DGAPA-UNAM project number IN201615.
Materials
| 0.5 mm Glass beads | Biospec Products | 11079105 | |
| Tris Base | Formedium | TRIS01 | Ultra pure |
| Glycerol | Sigma-Aldrich | G5516 | |
| dye 4’, 5’-bis(1,3,2 dithioarsolan-2-yl) fluorescein | ThermoFischer Scientific | LC6090 | This kit contains the dye to label a FlAsH tag |
| Ampiciline | IBI Shelton Scientific, Inc | IB02040 | |
| D(+)-Glucose Anhydrous | Formedium | GLU03 | |
| D(+)-Galactose | Formedium | GAL03 | |
| L-Leucine | Formedium | DOC0157 | |
| L-Tryptofan | Formedium | DOC0189 | |
| Bezamidine hydrochloride | Sigma-Aldrich | B6506-5G | |
| PMSF | Gold Biotechnology, Inc | P-470-25 | Phenylmethylsulfonyl fluoride |
| NaCl | Formedium | NAC02 | Sodium Chloride |
| Glycerol | Tecsiquim, S.A. de C.V. | GT1980-6 | |
| MgCl2 | Merck Millipore Corporation | 1725711000 | Magnesium Chloride |
| Imidazole | Sigma-Aldrich | I2399-500G | |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-100ML | |
| K2HPO4 | Sigma-Aldrich | P3786-500G | Potassium phosphate dibasic |
| NaH2PO4 | Sigma-Aldrich | S3139-500G | Sodium phosphate monobasic |
| Yeast nitrogen base without amino acids | Formedium | CYN0410 | |
| Yeast extract | Formedium | YEM03 | Micro Granulated |
| L-Tyroisne | Formedium | DOC0193 | |
| Adenine sulphate | Formedium | DOC0230 | |
| Casamino acids | Formedium | CAS03 | |
| Tryptone | IBI Shelton Scientific, Inc | IB49182 | |
| IPTG | Formedium | IPTG025 | |
| Name of Material | Company | Catalog Number | Comments/Description |
| Filtration units | Merck Millipore Corporation | UFC901096 | Amicon Ultra-15, membrana PLGC Ultracel-PL, 10 kDa |
| Membrane Filter | Merck Millipore Corporation | GSWP04700 | Membrane Filter, mixed cellulose esters, Hydrophilic, 0.22 µm, 47 mm, white, plain |
| Ni2+ affinity column | QIAGEN | 30760 | Cartridge pre-filled with 5 ml Ni-NTA Superflow |
| Strong Sulfopropyl cation exchanger column | GE Healthcare Life Science | 17-5157-01 | HiTrap SP Sepharose FF 5 ml |
| Size Exclusion column | GE Healthcare Life Science | 28989335 | HiLoad 16/600 Superdex 200 PG |
| Fluorescence cell | Hellma Analytics | 111-057-40 | |
| Name of Equipment | Company | Catalog Number | Comments/Description |
| Spectrophotometer | Agilent Technologies | G6860AA | Cary 60 UV-Vis |
| Shaker | ThermoFischer Scientific | SHKA4000-7 | MaxQ 4000 Benchtop temperature range Ambient-15° to 60°C |
| Centrifuge | ThermoFischer Scientific | 75004271 | Heraeus Megafuge 16R |
| FPLC | Pharmacia Biotech | Discontinued | FPLC system conductivity UV-MM II monitor P500 pump fraction |
| Spectrofluorometer | Olis | No applicable | Olis DM 45 with Polarization Toolbox |
References
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440 (7084), 637-643 (2006).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Michnick, S. W., Hien Ear, P., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods in Enzymology. 470, 336-366 (2010).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Dwane, S., Kiely, P. A. Tools used to study how protein complexes are assembled in signaling cascades. Bioeng Bugs. 2 (5), 247-259 (2011).
- Snider, J., et al. Fundamentals of protein interaction network mapping. Mol Syst Biol. 11 (12), 848 (2015).
- Fersht, A., Baldwin, R. L. . Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. , 191-214 (2002).
- Menne, T. F., et al. The Shwachman-Bodian-Diamond syndrome protein mediates translational activation of ribosomes in yeast. Nat Genet. 39 (4), 486-495 (2007).
- Boocock, G. R., et al. Mutations in SBDS are associated with Shwachman-Diamond syndrome. Nat Genet. 33 (1), 97-101 (2003).
- Garcia-Marquez, A., Gijsbers, A., de la Mora, E., Sanchez-Puig, N. Defective Guanine Nucleotide Exchange in the Elongation Factor-like 1 (EFL1) GTPase by Mutations in the Shwachman-Diamond Syndrome Protein. J Biol Chem. 290 (29), 17669-17678 (2015).
- Gijsbers, A., Garcia-Marquez, A., Luviano, A., Sanchez-Puig, N. Guanine nucleotide exchange in the ribosomal GTPase EFL1 is modulated by the protein mutated in the Shwachman-Diamond syndrome. Biochem Biophys Res Commun. 437 (3), 349-354 (2013).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2010).
- Nishigaki, T., Treviño, C. L., Gòmez, I. . Tools to understand protein-protein interactions. 37, 1-14 (2012).
- Johnson, I. . The Molecular Probes Handbook: A Guide to Fluorescent Probes and Labeling Technologies. , (2010).
- Sabnis, R. W. . Handbook of Fluorescent Dyes and Probes. , (2015).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: synthesis and biological applications. J Am Chem Soc. 124 (21), 6063-6076 (2002).
- Griffin, B. A., Adams, S. R., Tsien, R. Y. Specific covalent labeling of recombinant protein molecules inside live cells. Science. 281 (5374), 269-272 (1998).
- Rashidian, M., Dozier, J. K., Distefano, M. D. Enzymatic labeling of proteins: techniques and approaches. Bioconjug Chem. 24 (8), 1277-1294 (2013).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: A laboratory manual. , (2001).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- West, R. W., Chen, S. M., Putz, H., Butler, G., Banerjee, M. GAL1-GAL10 divergent promoter region of Saccharomyces cerevisiae contains negative control elements in addition to functionally separate and possibly overlapping upstream activating sequences. Genes Dev. 1 (10), 1118-1131 (1987).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Eftink, M. R. Fluorescence methods for studying equilibrium macromolecule-ligand interactions. Methods Enzymol. 278, 221-257 (1997).
- Han, H., et al. Binding of Substrates to the Central Pore of the Vps4 ATPase Is Autoinhibited by the Microtubule Interacting and Trafficking (MIT) Domain and Activated by MIT Interacting Motifs (MIMs). J Biol Chem. 290 (21), 13490-13499 (2015).
- Sanchez-Puig, N., Veprintsev, D. B., Fersht, A. R. Binding of natively unfolded HIF-1alpha ODD domain to p53. Mol Cell. 17 (1), 11-21 (2005).
- Trusch, F., et al. The N-terminal Region of the Ubiquitin Regulatory X (UBX) Domain-containing Protein 1 (UBXD1) Modulates Interdomain Communication within the Valosin-containing Protein p97. J Biol Chem. 290 (49), 29414-29427 (2015).
- Kamp, F., Beyer, K. Binding of alpha-synuclein affects the lipid packing in bilayers of small vesicles. The Journal of Biological Chemsitry. 281, 9251-9259 (2006).
- Bujalowski, W. M., Jezewska, M. J. Fluorescence Intensity, Anisotropy, and Transient Dynamic Quenching Stopped-Flow Kinetics. Spectroscopic Methods of Analysis. 875, 105-133 (2012).
- Asano, N., et al. Direct interaction between EFL1 and SBDS is mediated by an intrinsically disordered insertion domain. Biochem Biophys Res Commun. 443 (4), 1251-1256 (2014).

