Fluorescensanisotropi som ett verktyg för att studera protein-proteininteraktioner
Summary
Proteininteraktioner är kärnan i en cell funktion. Kalorimetriska och spektroskopiska tekniker används ofta för att karakterisera dem. Här beskriver vi fluorescensanisotropi som ett verktyg för att studera interaktionen mellan proteinet muterat i Shwachman-Diamond syndrom (SBD) och förlängningsfaktor liknande ett GTPas (EFL1).
Abstract
Protein-proteininteraktioner spelar en viktig roll i funktionen av en levande organism. När en interaktion har identifierats och validerats är det nödvändigt att karakterisera det på strukturella och mekanistiska nivå. Flera biokemiska och biofysikaliska metoder finns för detta ändamål. Bland dem är fluorescensanisotropi en kraftfull teknik används i synnerhet när fluorescensintensiteten för en fluorofor-märkt protein förblir konstant vid protein-proteininteraktion. I denna teknik är en fluorofor-märkt protein exciteras med vertikalt polariserat ljus av en lämplig våglängd som selektivt exciterar en delmängd av fluoroforema enligt deras relativa orientering med den inkommande strålen. Den resulterande utsläpp har också en rikt vars förhållande i de vertikala och horisontella plan definierar anisotropi (r) enligt följande: r = (I VV -I VH) / (I VV + 2I VH), där jag vv och jag <sub> VH är fluorescensintensiteterna hos de vertikala och horisontella komponenter, respektive. Fluorescensanisotropi är känslig för rotations diffusion av en fluorofor, nämligen den skenbara molekylstorleken hos en fluorofor fäst till en protein, som förändras vid protein-proteininteraktion. I denna text, har användningen av fluorescens anisotropi som ett verktyg för att studera protein-proteininteraktioner exemplifieras att ta itu med bindningen mellan proteinet muterat i Shwachman-Diamond syndrom (SBD) och förlängningsfaktor lika ett GTPas (EFL1). Konventionellt är märkning av ett protein med en fluorofor som utförs på de tiolgrupper (cystein) eller i de aminogrupper (de N-terminala amin eller lysin) av proteinet. Emellertid SBD besitter flera cysteiner och lysiner som inte tillåter sätesriktad märkning av den. Som en alternativ teknik, färgämnet 4 ', var 5'-bis (1,3,2 dithioarsolan-2-yl) fluorescein användas för att specifikt märka en tetracysteine motiv, Cys-Cys-Pro-Gly-Cys-Cys, gentekniskt i C-terminalen av det rekombinanta SBD-protein. Montering av de experimentella data tillgänglig kvantitativ och mekanistisk information om bindningsläge mellan dessa proteiner.
Introduction
Celler innehåller en mångfald av biomakromolekyler som ständigt interagerar med varandra. Denna förening ger upphov till komplex som deltar i de cellulära vägar som ansvarar för deras funktion i signalöverföring, reglering av genuttryck och cell migration bland andra. Alla protein-proteininteraktioner som sker i en cell innefattar ett nätverk som kallas interactome. I Saccharomyces cerevisiae har visats mer än 70% av dess proteiner för att ha interagerande partners 1. Förstå interactome av en cell och deras funktioner ger relevant information på komplexiteten och mångfalden av levande organismer. Flera metoder har beskrivits för att identifiera och karakterisera protein-proteininteraktioner. Olika hög genomströmningshastighet metoder såsom jäst två-hybrid-2, proteinfragmentkompletteringsanalyser 3, affinitetsrening 4 kopplad till masspektrometri och protein microarrays används för att identifiera en interaktion 5,6. När detta har skett, är det nödvändigt att bekräfta det och kan variera från fall till fall. Normalt är dessa experiment involverar att störa interaktionen sig på samma nivå som de enskilda medlemmarna i samspelet paret, t ex genom gendeletion eller överuttryck av ett av proteinerna, och sedan letar efter förändringar i egenskaper eller funktion hos andra medlem på cellnivå. Därefter biofysiska tekniker 7 används för att karakterisera interaktion protein-protein på molekylnivå. För detta ändamål, är strukturen av proteinkomplex bestämdes genom röntgenkristallografi, kärnmagnetisk resonans och Cryo-elektronmikroskopi medan kalorimetri och fluorescensspektroskopi används för att kvantitativt och mekanistiskt beskriva dem.
I detta arbete, var fluorescensanisotropi används som en teknik för att karakterisera interaktionen mellan GTPas EFL1 och SBDS-proteinet. Dessa proteiner deltar i syntesen av ribosomer genom att främja frisättningen av eukaryota inledande faktor 6 från ytan av 60S ribosomala subenheten 8. Den SBD-protein är muterat i en sjukdom som är känd som den Shwachman-Diamond syndrom 9 och fungerar som en guaninnukleotidutbytesfaktor för EFL1 minska dess affinitet för Guanosindifosfat 10,11. Sjukdoms mutationer i SBD avskaffa interaktionen med EFL1 och därmed förhindra dess aktivering.
Fluorescensanisotropi används allmänt i biologiska tillämpningar för att studera protein-peptid eller protein-nukleinsyra-interaktioner. Den bygger på principen att en fluorofor exciteras med polariserat ljus resulterar i en delvis polariserad emission. Fluorescensanisotropi definieras av ekvation 1:
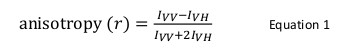
där jag Vv och jag VH ärfluorescensintensiteter av vertikalt (VV) och horisontellt (VH) polariserad emission när provet exciteras med vertikalt polariserat ljus 12. Fluorescensanisotropi är känslig för faktorer som påverkar graden av rotations diffusion av fluoroforen och sålunda beror på temperaturen, viskositeten hos lösningen och den synbara molekylstorleken av fluoroforen. Den skenbara storleken av ett protein innehållande en fluorofor ökar när det interagerar med ett annat protein och en sådan förändring kan därefter utvärderas som en ändring i anisotropi. Mer specifikt kommer en fluorofor som roterar långsamt i lösning i förhållande till dess fluorescerande livslängd har en stor jag VV värde och liten jag VH värde och därför kommer att uppvisa en relativt stor anisotropi. För fluoroforer som tumlar snabbt i förhållande till deras fluorescerande livstid, jag vv och jag VH kommer att likna och deras anisotropi värdet kommer att vara liten 12 </sup> (Figur 1). Dessutom, för en god anisotropi signalbrusmätning, är det nödvändigt att ha en fluorofor med en fluorescenslivstid liknar rotationskorrelationstiden av molekylen av intresse. Annars är det inte möjligt att på ett korrekt skillnaden i anisotropi mellan fritt protein och att i komplexet. Till exempel, är anisotropin hos en fluorescerande sond med en livstid nära 4 nsek såsom fluorescein eller rodamin bundna till en lågmolekylär förening med 100 Da 0,05. Bindning till en molekyl av 160 kDa kommer att öka sin anisotropi värde till 0,29; en skillnad som kan mätas noggrant. Däremot kommer samma fluorescerande prob involverad i en bindningsreaktion, vars ökning i molekylstorleken varierar från 65 till 1.000 kDa endast resultera i en anisotropi förändring av 0,28 till 0,3, vilket är för liten för att mätas noggrant. I detta scenario skulle en sond med en livslängd på 400 ns vara lämpligare 12.
<pclass = "jove_content">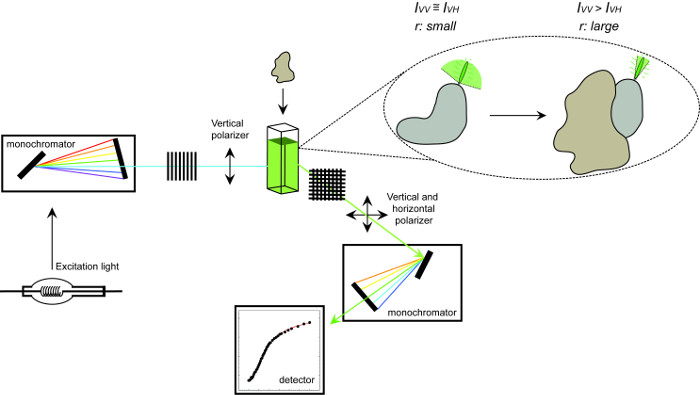
Figur 1. Schematisk representation av den utrustning som används för att mäta fluorescens anisotropi och proceduren. Schematisk representation av den utrustning som används för att utföra en protein-proteininteraktion experiment mätning av fluorescens anisotropi. Fluoroforer som tumlar snabbt display liten anisotropi som ökar vid bindning till en interaktion partner. Klicka här för att se en större version av denna siffra.
Fluorescensapplikationer kräver närvaro av en fluorofor i någon av molekylerna som studerats. För att studera protein-proteininteraktioner finns tre typer av fluoroforer: 1) de tryptofanrester närvarande i proteinerna, 2) kemiskt bundna fluoroforer och 3) fluorescerande fusionspartners såsom grönt fluorescerande protein (GFP) och dess Derivahavare. De flesta proteiner har tryptofanrester på dess struktur, således det enklaste sättet att mäta en interaktion är genom att övervaka förändringarna i den motsvarande fluorescensspektra eller genom övervakning av förändringar i fluorescensintensiteten hos de tryptofanrester. Emellertid kan tryptofangrupper vara närvarande i båda proteinerna komplicerar analysen. Å andra sidan, för en fluorofor att ändra sina fluorescerande egenskaper beroende på en interaktion den behöver för att vara placerad på eller i närheten av bindningsstället och det kan störa interaktionen självt. Detta kräver särskild uppmärksamhet vid användning av skrymmande fluoroforer såsom GFP. Om ingen av dessa fluoroforer kan användas för bindningsstudier är det nödvändigt, då att införa yttre fluoroforer till ett av de proteiner som är inblandade. Många kemiskt syntetiserade fluoroforer existerar och kan fästas kovalent till proteiner via deras reaktiva grupper såsom amingrupper (sidokedjan i lysiner eller N-terminalen) och tiolgrupperna i cystein. Fluorophore derivat med isotiocyanat och succinimidylestrar reagerar med amidgrupper medan jodacetamid och maleimid är tiol-reaktiva grupper 13. De vanligaste färgämnen som används i fluorescensapplikationer är derivat av fluorescein och rodamin gröna färgämnen, kumariner, BODIPY fluoroforer och Alexa Fluor färgämnen. En detaljerad lista över kommersiellt tillgängliga fluoroforer och deras användning kan hittas i referenser 14,15. För framgångsrik märkning, måste den reaktiva gruppen att exponeras på ytan av proteinet, men på grund av det stora antalet reaktiva funktionella grupper som vanligen är närvarande i polypeptider är det mycket svårt att få platsspecifik modifiering. Proteinet av intresse i denna studie, SBD innehåller 5 fria cysteiner och 33 lysiner som kan resultera i flera plats märkning. Icke-enhetlig märkning kan påverka bindningen och kommer att försvåra dataanalys som olika fluoroforen molekyler kan framkalla olika fluorescerande intensitetssignaler vid bindning. att overcome detta problem använde vi Flash fluorofor, 4 ', 5'-bis (1,3,2 dithioarsolan-2-yl) fluorescein till plats direkt etikett SBD-protein. Detta är en arsenoxide färgämne med en hög affinitet för fyra åtskilda cysteiner i ett motiv vet som Flash-tag som består av sekvensen CCXXCC där X är vilken aminosyra som helst annan än cystein 16,17. Denna tetracysteine motiv sättes till N- eller C-terminalen av proteinet med genteknik tillsammans med en lämplig linker för att förhindra störningar av den totala vikningen av proteinet. Paret som består av flash-färgämne och FLASH-tagg var ursprungligen avsedd att platsspecifika etikett proteiner i levande celler 17 men det kan också användas för att märka renade proteiner in vitro såsom den exemplifieras här. Dessutom har enzymatiska strategier också utvecklats för att göra det möjligt för platsspecifik funktionalisering av proteiner 18.
I detta manuskript beskriver vi nyttan av fluorescens anisotropi ensa verktyg för att studera protein-proteininteraktioner. kan bedömas genom enkel inspektion av bindande kurvform bindande medan kvantitativ information kan erhållas från passningen av experimentella data.
Protocol
Representative Results
Discussion
De flesta biokemiska experiment med proteiner kräver inte bara rent protein utan också stora mängder av dem, oberoende av vilken teknik som används. Av denna anledning är de proteiner som används för denna typ av experiment som erhållits genom heterologt uttryck, som det var fallet som presenteras här. Blomningstid spektroskopi kräver närvaro av en fluorofor i den studerade molekylen. Aromatiska rester utgör inneboende fluoroforer av ett protein, men med hjälp av sin signal för att studera protein-proteini…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Authors acknowledge the financial support from CONACyT project numbers 167359 and 177138, and from DGAPA-UNAM project number IN201615.
Materials
| 0.5 mm Glass beads | Biospec Products | 11079105 | |
| Tris Base | Formedium | TRIS01 | Ultra pure |
| Glycerol | Sigma-Aldrich | G5516 | |
| dye 4’, 5’-bis(1,3,2 dithioarsolan-2-yl) fluorescein | ThermoFischer Scientific | LC6090 | This kit contains the dye to label a FlAsH tag |
| Ampiciline | IBI Shelton Scientific, Inc | IB02040 | |
| D(+)-Glucose Anhydrous | Formedium | GLU03 | |
| D(+)-Galactose | Formedium | GAL03 | |
| L-Leucine | Formedium | DOC0157 | |
| L-Tryptofan | Formedium | DOC0189 | |
| Bezamidine hydrochloride | Sigma-Aldrich | B6506-5G | |
| PMSF | Gold Biotechnology, Inc | P-470-25 | Phenylmethylsulfonyl fluoride |
| NaCl | Formedium | NAC02 | Sodium Chloride |
| Glycerol | Tecsiquim, S.A. de C.V. | GT1980-6 | |
| MgCl2 | Merck Millipore Corporation | 1725711000 | Magnesium Chloride |
| Imidazole | Sigma-Aldrich | I2399-500G | |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-100ML | |
| K2HPO4 | Sigma-Aldrich | P3786-500G | Potassium phosphate dibasic |
| NaH2PO4 | Sigma-Aldrich | S3139-500G | Sodium phosphate monobasic |
| Yeast nitrogen base without amino acids | Formedium | CYN0410 | |
| Yeast extract | Formedium | YEM03 | Micro Granulated |
| L-Tyroisne | Formedium | DOC0193 | |
| Adenine sulphate | Formedium | DOC0230 | |
| Casamino acids | Formedium | CAS03 | |
| Tryptone | IBI Shelton Scientific, Inc | IB49182 | |
| IPTG | Formedium | IPTG025 | |
| Name of Material | Company | Catalog Number | Comments/Description |
| Filtration units | Merck Millipore Corporation | UFC901096 | Amicon Ultra-15, membrana PLGC Ultracel-PL, 10 kDa |
| Membrane Filter | Merck Millipore Corporation | GSWP04700 | Membrane Filter, mixed cellulose esters, Hydrophilic, 0.22 µm, 47 mm, white, plain |
| Ni2+ affinity column | QIAGEN | 30760 | Cartridge pre-filled with 5 ml Ni-NTA Superflow |
| Strong Sulfopropyl cation exchanger column | GE Healthcare Life Science | 17-5157-01 | HiTrap SP Sepharose FF 5 ml |
| Size Exclusion column | GE Healthcare Life Science | 28989335 | HiLoad 16/600 Superdex 200 PG |
| Fluorescence cell | Hellma Analytics | 111-057-40 | |
| Name of Equipment | Company | Catalog Number | Comments/Description |
| Spectrophotometer | Agilent Technologies | G6860AA | Cary 60 UV-Vis |
| Shaker | ThermoFischer Scientific | SHKA4000-7 | MaxQ 4000 Benchtop temperature range Ambient-15° to 60°C |
| Centrifuge | ThermoFischer Scientific | 75004271 | Heraeus Megafuge 16R |
| FPLC | Pharmacia Biotech | Discontinued | FPLC system conductivity UV-MM II monitor P500 pump fraction |
| Spectrofluorometer | Olis | No applicable | Olis DM 45 with Polarization Toolbox |
References
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440 (7084), 637-643 (2006).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Michnick, S. W., Hien Ear, P., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods in Enzymology. 470, 336-366 (2010).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Dwane, S., Kiely, P. A. Tools used to study how protein complexes are assembled in signaling cascades. Bioeng Bugs. 2 (5), 247-259 (2011).
- Snider, J., et al. Fundamentals of protein interaction network mapping. Mol Syst Biol. 11 (12), 848 (2015).
- Fersht, A., Baldwin, R. L. . Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. , 191-214 (2002).
- Menne, T. F., et al. The Shwachman-Bodian-Diamond syndrome protein mediates translational activation of ribosomes in yeast. Nat Genet. 39 (4), 486-495 (2007).
- Boocock, G. R., et al. Mutations in SBDS are associated with Shwachman-Diamond syndrome. Nat Genet. 33 (1), 97-101 (2003).
- Garcia-Marquez, A., Gijsbers, A., de la Mora, E., Sanchez-Puig, N. Defective Guanine Nucleotide Exchange in the Elongation Factor-like 1 (EFL1) GTPase by Mutations in the Shwachman-Diamond Syndrome Protein. J Biol Chem. 290 (29), 17669-17678 (2015).
- Gijsbers, A., Garcia-Marquez, A., Luviano, A., Sanchez-Puig, N. Guanine nucleotide exchange in the ribosomal GTPase EFL1 is modulated by the protein mutated in the Shwachman-Diamond syndrome. Biochem Biophys Res Commun. 437 (3), 349-354 (2013).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2010).
- Nishigaki, T., Treviño, C. L., Gòmez, I. . Tools to understand protein-protein interactions. 37, 1-14 (2012).
- Johnson, I. . The Molecular Probes Handbook: A Guide to Fluorescent Probes and Labeling Technologies. , (2010).
- Sabnis, R. W. . Handbook of Fluorescent Dyes and Probes. , (2015).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: synthesis and biological applications. J Am Chem Soc. 124 (21), 6063-6076 (2002).
- Griffin, B. A., Adams, S. R., Tsien, R. Y. Specific covalent labeling of recombinant protein molecules inside live cells. Science. 281 (5374), 269-272 (1998).
- Rashidian, M., Dozier, J. K., Distefano, M. D. Enzymatic labeling of proteins: techniques and approaches. Bioconjug Chem. 24 (8), 1277-1294 (2013).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: A laboratory manual. , (2001).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- West, R. W., Chen, S. M., Putz, H., Butler, G., Banerjee, M. GAL1-GAL10 divergent promoter region of Saccharomyces cerevisiae contains negative control elements in addition to functionally separate and possibly overlapping upstream activating sequences. Genes Dev. 1 (10), 1118-1131 (1987).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Eftink, M. R. Fluorescence methods for studying equilibrium macromolecule-ligand interactions. Methods Enzymol. 278, 221-257 (1997).
- Han, H., et al. Binding of Substrates to the Central Pore of the Vps4 ATPase Is Autoinhibited by the Microtubule Interacting and Trafficking (MIT) Domain and Activated by MIT Interacting Motifs (MIMs). J Biol Chem. 290 (21), 13490-13499 (2015).
- Sanchez-Puig, N., Veprintsev, D. B., Fersht, A. R. Binding of natively unfolded HIF-1alpha ODD domain to p53. Mol Cell. 17 (1), 11-21 (2005).
- Trusch, F., et al. The N-terminal Region of the Ubiquitin Regulatory X (UBX) Domain-containing Protein 1 (UBXD1) Modulates Interdomain Communication within the Valosin-containing Protein p97. J Biol Chem. 290 (49), 29414-29427 (2015).
- Kamp, F., Beyer, K. Binding of alpha-synuclein affects the lipid packing in bilayers of small vesicles. The Journal of Biological Chemsitry. 281, 9251-9259 (2006).
- Bujalowski, W. M., Jezewska, M. J. Fluorescence Intensity, Anisotropy, and Transient Dynamic Quenching Stopped-Flow Kinetics. Spectroscopic Methods of Analysis. 875, 105-133 (2012).
- Asano, N., et al. Direct interaction between EFL1 and SBDS is mediated by an intrinsically disordered insertion domain. Biochem Biophys Res Commun. 443 (4), 1251-1256 (2014).

