Kortlægning af bindingssted en aptamer på ATP Brug mikroskala Thermophoresis
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
Interaktion mellem molekyler er grundlaget for naturen. Derfor forskere i mange områder af grundforskning og anvendt forskning forsøge at forstå de grundlæggende principper om molekylære interaktioner af forskellig art. Individuel Thermophoresis (MST) gør det muligt for forskere at udføre hurtig, præcis, omkostningseffektiv, og kvalitet-kontrollerede karakterisering af molekylære interaktioner i opløsning, med frit valg af buffere. Der er allerede mere end 1.000 publikationer MST, fra 2016 alene, der beskriver forskellige former for analyser, herunder bibliotekets screeninger, bindende event valideringer, konkurrence analyser og eksperimenter med flere bindende partnere 1-8. Generelt MST tillader studiet af de klassiske bindende parametre, såsom bindingsaffinitet (pM til mM), støkiometri og termodynamik, af enhver form for molekylær interaktion. En stor fordel ved MST er evnen til at studere bindingsbegivenheder uafhængigt af størrelsen af de interaktionspartnere. Selv udfordrende interaktioner mellem små nucleinsyre aptamerer (15-30 nt) og mål såsom små molekyler, lægemidler, antibiotika eller metabolitter kan kvantificeres.
Aktuelle state-of-the-art teknologier til at karakterisere aptamer målgruppen interaktioner er enten lab-intens og meget komplekse eller undlader at kvantificere aptamer-lille molekyle interaktioner 9,10. Surface (SPR) -baserede assays 11,12 og virkelig label-fri kalorimetriske tilgange, såsom Isotermisk Titrering kalorimetri (ITC) 13-15, isokratisk eluering 16, ligevægt fi ltrering 17,18, in-line sondering 19, gel- skift analyser, stopped- fl ow fl uorescence spektroskopi 20,21, fluorescens anisotropi (FA) 22,23, single-molekyle fl uorescence billedbehandling 24,25, og Bio-lags interferometri (BLI) 26 er også enten upræcise eller uforenelig med aptamer-lille molekyle interaktioner. Andre principal spørgsmål af disse metoder er lav følsomhed, højt forbrug prøve, immobilisering, masse begrænsninger transport på overflader, og / eller buffer restriktioner. Kun få af disse teknologier giver integrerede styringer til sammenlægning og adsorption effekter.
MST er et effektivt redskab for forskerne at overvinde denne begrænsning for at undersøge samspillet mellem aptamerer og små molekyler 27-29, samt andre mål, såsom proteiner 30-33. Teknologien bygger på flytning af molekyler gennem temperaturgradienter. Denne rettet bevægelse, kaldet "thermophoresis," afhænger af størrelse, ladning, og hydrering skallen af molekylet 34,35. Bindingen af en ligand til molekylet vil direkte ændre mindst en af disse parametre, hvilket resulterer i en ændret thermophoretic mobilitet. Ligander med små størrelser kan ikke have en betydelig indvirkning i form af størrelse ændring fra den ubundne til den bundne tilstand, men de kan have dr amatic virkninger på hydrering skal og / eller beregning. Ændringerne i thermophoretic bevægelse af molekyler efter interaktioner med bindingspartneren muliggør kvantificering af grundlæggende bindingsparametre 2,7,34,36,37.
Som afbildet i figur 1A, MST anordning består af en infrarød laser fokuseres på prøven inden for de glaskapillarer anvendelse af de samme optikker som for fluorescens detektion. Kan overvåges thermophoretic bevægelse af proteiner via den iboende fl uorescence af tryptophaner 6 eller af en fluorescens-mærket interaktion partner 3,8, mens laseren etablerer en temperaturgradient (AT på 2-6 ° C). Den resulterende temperaturforskel i rummet, AT, fører til udtømning eller akkumulering af molekyler i området for forhøjet temperatur, som kan kvantificeres ved Soret fi cient (S T):
g "/>
c varmt repræsenterer koncentrationen i den opvarmede region, og C Kold er koncentrationen i den indledende koldt region.
Som vist i figur 1B, en typisk MST eksperimentresultaterne i en MST bevægelse profil (tidssporet), der består af forskellige faser, som kan adskilles ved deres respektive tidsplaner. Den indledende fluorescens måles i de første 5 s i fravær af temperaturgradienten at definere den præcise start fluorescens og for at kontrollere for fotoblegning eller photoenhancement. Temperaturen Jump (T-jump) repræsenterer den fase, hvor fluorescens ændringer før thermophoretic bevægelse. Denne indledende fald i fluorescens afhænger varme-afhængige ændringer i fl uorophore kvantumudbytte. Den thermophoresis fase følger, hvor fluorescens falder (eller stiger) på grund af den thermophoretic bevægelse af molekylerne indtil steady-state fordelingen er nået.Kan observeres den omvendte TJump og samtidig tilbagediffusion af fl uorescent molekyler som vist i figur 1 B, efter at laseren er slukket. For at få adgang grundlæggende bindende parametre, er forskellige molære forhold af interaktionspartnere analyseret og sammenlignet. Typisk er 16 forskellige forhold undersøgt i en MST eksperiment, hvorimod den optiske synlige molekyle holdes konstant og leveres med en stigende mængde af umærket ligand. Samspillet mellem de to bindende partnere inducerer ændringer i thermophoresis, og dermed i det normaliserede fl uorescence, F norm, der beregnes som følger:
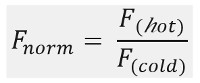
F varmt og F koldt repræsenterer gennemsnit fl uorescence intensiteter på defineret tidspunkter af MST spor. Bindingsaffiniteter (K d eller EC 50 værdier) kan beregnes ved Curve fitting (figur 1C).
Samlet set MST er et stærkt værktøj til at studere molekylære interaktioner af nogen art. Dette håndskrift tilbyder en protokol til at karakterisere udfordrende samspil mellem det lille molekyle adenosin trifosfat (ATP, 0,5 kDa) og 25-nt kort ssDNA aptamer DH25.42 (7,9 kDa). I løbet af manuskriptet, er bindingsstedet for aptameren på ATP-molekylet kortlagt ned til adenin gruppe af ATP.
Protocol
Representative Results
Discussion
Kvalitet kontrol:
Uspecifik fastklæbning / adsorption af prøvemateriale til overflader, samt aggregering effekter, har en dramatisk indflydelse på kvaliteten af de affinitet data. Men kun få state-of-the-art teknologier giver præcise og hurtige muligheder for at overvåge og undgå disse effekter. MST tilbyder integrerede kvalitetskontroller, der registrerer og bidrage til at overvinde disse problemer, der giver mulighed for trinvis optimering af tekniske setup. Vigtig information om…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Forfatterne har ingen bekræftelser.
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochimie. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochimie. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochimie. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochimie. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

