Картирование участок связывания аптамер на АТФ Использование Microscale Термофорез
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
Взаимодействие между молекулами является основой природы. Таким образом, ученые во многих областях фундаментальных и прикладных исследований пытаются понять основные принципы молекулярных взаимодействий различных видов. Microscale Термофорез (MST) позволяет ученым осуществлять быстрый, точный, экономичный, и контроль качества характеристику молекулярных взаимодействий в растворе, со свободным выбором буферов. Есть уже более 1000 публикаций с использованием MST, с 2016 года в одиночку, описывая различные виды анализов, в том числе библиотеки скринингов, связывание валидаций событий, анализы конкуренции, а также эксперименты с несколькими партнерами связывания 1-8. В общем, МСТ позволяет исследовать классических параметров связывания, такие как аффинность связывания (стр.м.в мМ), стехиометрии и термодинамики, любого вида молекулярного взаимодействия. Большим преимуществом MST является возможность изучения связывания событий независимо от размера партнеров взаимодействия. Даже чалlenging взаимодействия между малыми аптамеров нуклеиновых кислот (15-30 нт) и задачи, такие как небольшие молекулы, лекарственные средства, антибиотики или метаболиты могут быть определены количественно.
Текущее состояние-оф-современные технологии для характеристики аптамеров-мишени взаимодействия либо лабораторного интенсивной и очень сложной или не в состоянии количественно аптамеров-малая молекула взаимодействиях 9,10. Поверхностного плазмонного резонанса (SPR) основе анализов 11,12 и действительно этикетки свободные калориметрических подходы, такие как изотермической калориметрии (титрования ITC), 13-15 изократическим элюирования 16, равновесному инфильтрации 17,18, рядный зондирования 19, гелеобразные анализах сдвига, stopped- потока флуоресценции спектроскопии 20,21, анизотропии флуоресценции (FA) 22,23, одной молекулы флуоресценции изображений 24,25 и Био-слой интерферометрии (BLI) 26 также являются либо неточными или несовместим с аптамеров-небольшой молекулы взаимодействия. Другое principaл вопросы этих методов низкая чувствительность, высокое потребление пробы, иммобилизация, ограничение массовых перевозок на поверхностях, и / или ограничения буфера. Лишь немногие из этих технологий обеспечивают интегрированные средства управления для агрегации и адсорбции эффектов.
MST представляет собой мощный инструмент для ученых , чтобы преодолеть это ограничение для изучения взаимодействия между аптамеров и малыми молекулами 27-29, а также другие цели , такие как белки 30-33. Технология опирается на движение молекул через температурных градиентов. Это направленное движение, называемое "термофорезу," зависит от размера, заряда и гидратной оболочки молекулы 34,35. Связывание лиганда с молекулой будет непосредственно изменять, по меньшей мере один из этих параметров, в результате чего в измененном термофоретического мобильности. Лиганды с малыми размерами не могут иметь значительное влияние с точки зрения изменения размера от несвязанного в связанном состоянии, но они могут иметь Др amatic воздействие на оболочку гидратации и / или заряда. Изменения в термофоретического движение молекул после взаимодействия с партнером по связыванию позволяет количественно оценить основные параметров связывания 2,7,34,36,37.
Как показано на рисунке 1А, устройство МСТ состоит из инфракрасного лазера фокусировалось на образец в стеклянных капиллярах с использованием тех же оптических систем для флуоресцентной детекции. Термофоретическая движение белков с помощью собственной флуоресценции триптофанов 6 или флуоресцентно меченого партнерского взаимодействия 3,8 можно отслеживать в то время как лазер создает температурный градиент (& Delta ; t 2-6 ° С). Полученная разность температур в пространстве, & Delta ; t, приводит к истощению или накопление молекул в области повышенной температуры, которая может быть определена количественно с помощью Соре коэффици- циент (С Т):
г "/>
С горячей представляет собой концентрацию в нагретой области, и с холодом концентрация в начальной холодной области.
Как показано на фигуре 1В, типичный результаты эксперимента MST в профиле движения MST (время трассировки), состоящий из различных этапов, которые могут быть разделены с помощью их соответствующих временных рамок. Первоначальная измеряют флуоресценцию в первые 5 секунд в отсутствие градиента температуры, чтобы определить точную начальную флуоресценцию и для проверки наличия фотообесцвечиванию или photoenhancement. Скачок температуры (T-Jump) представляет собой фазу, в которой изменения флуоресценции до термофоретического движения. Это начальное снижение флуоресценции зависит от тепловых зависящих от изменений фл uorophore квантового выхода. Фаза термофорезу следует, в которых флуоресценция уменьшается (или увеличивается) в связи с термофоретического движения молекул вплоть до стационарного распределения достигается.Обратное TJump и сопутствующее назад диффузия фл uorescent молекул можно наблюдать , как показано на рисунке 1В после того, как лазер выключен. Для того, чтобы получить доступ к основным параметрам связывания, различные молярные отношения партнеров взаимодействия анализируются и сравниваются. Как правило, 16 различных соотношений изучаются в одном эксперименте MST, в то время как оптический видимый молекулы поддерживается постоянным и поставляется с увеличением количества немеченого лиганда. Взаимодействие между этими двумя связывающими партнерами вызывает изменения в ТФ, и , таким образом , в нормированной флуоресценции, F норма, которая рассчитывается следующим образом :
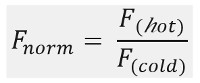
F горячей и холодной F представляют собой усредненные интенсивности uorescence Fl на де определены моменты времени следов MST. Аффинности связывания (K D или 50 значений EC) можно рассчитать по CURVе фитинга (рис 1C).
В целом, MST является мощным инструментом для изучения молекулярных взаимодействий любого рода. Эта рукопись предлагает протокол характеризовать сложные взаимодействия между молекулой небольшой аденозинтрифосфата (АТФ, 0,5 кДа) и 25-нт короткий оцДНК аптамеров DH25.42 (7,9 кДа). В течение рукописи, сайт связывания аптамеров на молекулы АТФ отображается вплоть до аденин группы АТФ.
Protocol
Representative Results
Discussion
Контроль качества:
Неспецифические прилипание / адсорбции материала образца на поверхности, а также агрегации эффектов, оказывают серьезное влияние на качество данных сродством. Тем не менее, лишь немногие внедренный технологии обеспечивают точные и быстрые варианты ко…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Авторы не имеют никаких подтверждений.
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochimie. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochimie. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochimie. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochimie. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

