Kartläggning av bindningsstället för en aptamer på ATP Använda Micro Thermophoresis
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
Interaktion mellan molekyler utgör grunden för naturen. Därför forskare inom många områden av grundforskning och tillämpad forskning försöker förstå de grundläggande principerna för molekylära interaktioner av olika slag. Micro Thermophoresis (MST) gör det möjligt för forskare att utföra snabba, exakta och kostnadseffektiva, och kvalitetskontrollerad karakterisering av molekylära interaktioner i lösning, med ett fritt val av buffertar. Det finns redan mer än 1.000 publikationer med hjälp av MST, från 2016 ensam, som beskriver olika typer av analyser, inklusive biblioteks filmvisningar, bindande händelse valideringar, konkurrensanalyser och experiment med flera bindningspartner 1-8. I allmänhet, MST tillåter studiet av de klassiska bindningsparametrar, såsom bindningsaffinitet (pM till mM), stökiometri, och termodynamik, av något slag av molekylär interaktion. En stor fördel med MST är förmågan att studera bindningshändelser oberoende av storleken av interaktionspartners. även chalnande interaktioner mellan små nukleinsyra aptamers (15-30 nt) och mål såsom små molekyler, läkemedel, antibiotika, eller metaboliter kan kvantifieras.
Nuvarande state-of-the-art teknik för att karakterisera aptamer-mål interaktioner är antingen lab-intensiv och mycket komplexa eller misslyckas med att kvantifiera aptamer-småmolekylära interaktioner 9,10. Surface Plasmon Resonance (SPR) -baserad analyser 11,12 och verkligen etikettfria kalorimetriska metoder, såsom Isotermiska Titrering Calorimetry (ITC) 13-15, isokratisk eluering 16, jämvikts fi ltration 17,18, in-line sondering 19, gel- skift analyser stopped- fl öde fl uorescence spektroskopi 20,21, fluorescensanisotropi (FA) 22,23, enda molekyl fl uorescence avbildning 24,25, och Bio-skikt interferometri (BLI) 26 är också antingen oprecisa eller oförenlig med aptamer-liten molekyl interaktioner. andra huvl frågor dessa metoder är låg känslighet, hög prov konsumtion, immobilisering, begränsningar masstransport på ytor, och / eller begränsningar buffert. Endast ett fåtal av dessa tekniker ger integrerade kontroller för aggregering och adsorption effekter.
MST utgör ett kraftfullt verktyg för forskare att övervinna denna begränsning för att studera samspelet mellan aptamers och små molekyler 27-29, liksom andra mål såsom proteiner 30-33. Tekniken bygger på förflyttning av molekyler genom temperaturgradienter. Detta riktad rörelse, kallad "thermophoresis" beror på storlek, laddning, och hydrering skal av molekylen 34,35. Bindningen av en ligand till molekylen kommer att direkt förändra åtminstone en av dessa parametrar, vilket resulterar i ett förändrat thermophoretic rörlighet. Ligander med små storlekar kan inte ha en betydande inverkan i termer av storlek förändring från den obundna till det bundna tillståndet, men de kan ha dr amatic effekter på vätske skal och / eller laddning. Förändringarna i thermophoretic rörelse av molekyler efter växelverkan med bindningspartnern möjliggör kvantifiering av grundläggande bindningsparametrar 2,7,34,36,37.
Såsom avbildas i figur 1 A, MST Anordningen består av en infraröd laser fokuseras på provet inom de glaskapillärer med användning av samma optik som för fluorescensdetektion. Den thermophoretic rörelsen av proteiner via den inre fl uorescence av tryptofaner 6 eller av en fluorescensmärkt interaktionspartner 3,8 kan övervakas medan lasern upprättas en temperaturgradient (AT för 2-6 ° C). Den resulterande temperaturskillnaden i rymden, AT, leder till utarmning eller ackumulering av molekyler i området för förhöjd temperatur, som kan kvantifieras genom det Soret koef fi räcklig (S T):
g "/>
c hett representerar koncentrationen i det uppvärmda området, och c kallt är koncentrationen i det inledande kall regionen.
Såsom visas i figur 1B, en typisk MST experimentresultat i en MST rörelseprofil (tidskurva), som består av olika faser, vilka kan separeras med deras respektive tidsskalor. Den initiala fluorescens mäts i de första 5 si frånvaro av temperaturgradienten att definiera exakt börjar fluorescens och för att kontrollera fotoblekning eller photoenhancement. Temperatur Jump (T-Jump) representerar fas där fluorescens ändringarna innan thermophoretic rörelse. Denna inledande minskning i fluorescens beror på värmeberoende förändringar av fl uorophore kvantutbyte. Den thermophoresis fas följer, i vilken fluorescens minskar (eller ökar) på grund av den thermophoretic rörelsen av molekylerna tills steady-state distributions uppnås.Kan observeras omvända TJump och samtidig tillbaka diffusion av fl uorescent molekyler som visas i figur 1B efter lasern är avstängd. För att få tillgång grundläggande bindningsparametrar används olika molära förhållanden av interaktionspartners analyseras och jämföras. Normalt är 16 olika förhållanden studerats i en MST experiment, medan den optiska synlig molekylen hålls konstant och matas med en ökande mängd av omärkt ligand. Samspelet mellan de två bindningspartners framkallar förändringar i thermophoresis, och därmed i den normaliserade fl uorescence, F norm, som beräknas enligt följande:
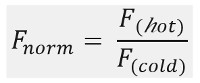
F varm och F kall representerar genomsnitt fl uorescence intensiteter vid de fi nierade punkter i MST spåren gång. Bindningsaffiniteter (K d eller EC 50-värden) kan beräknas genom curve montering (Figur 1C).
Totalt sett är MST ett kraftfullt verktyg för att studera molekylära interaktioner av något slag. Detta manuskript ger ett protokoll för att karakterisera utmanande samverkan mellan den lilla molekylen adenosintrifosfat (ATP, 0,5 kDa) och 25-nt kort ssDNA aptamer DH25.42 (7,9 kDa). Under loppet av manuskriptet, är bindningsstället för aptameren på ATP-molekylen mappas ner till adenin grupp av ATP.
Protocol
Representative Results
Discussion
Kvalitetskontroller:
Ospecifik klibbning / adsorption av provmaterial till ytor, liksom aggregering effekter, har en dramatisk inverkan på kvaliteten av de affinitetsdata. Men bara några state-of-the-art teknik erbjuder noggranna och snabba möjligheter att övervaka och undvika dessa effekter. MST erbjuder integrerade kvalitetskontroller som upptäcker och bidrar till att övervinna dessa frågor, gör det möjligt att stegvis optimering av den tekniska installationen. Viktig information om …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Författarna har inga bekräftelser.
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochimie. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochimie. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochimie. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochimie. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

