Los estudios de estructura-función en las células madre embrionarias de ratón Usando mediada por recombinasa Cambio de Cassette
Summary
Las proteínas a menudo contienen varios dominios que pueden ejercer diferentes funciones celulares. Genes knock-out (KO) no tienen en cuenta esta diversidad funcional. Aquí, se presenta un enfoque basado en la estructura-función de intercambio de casete de recombinación mediada (RMCE) en células madre embrionarias de KO que permite la disección molecular de varios dominios funcionales o variantes de una proteína.
Abstract
ingeniería de genes en embriones de ratón o células madre embrionarias (mESCs) permite el estudio de la función de una proteína dada. Las proteínas son los caballos de batalla de la célula y a menudo consisten en varios dominios funcionales, que pueden ser influenciados por modificaciones posteriores a la traducción. El agotamiento de la proteína completa en ratones condicional o constitutiva knock-out (KO) no tiene en cuenta esta diversidad funcional y la regulación. se informó previamente Una línea mESC y un modelo de ratón derivada, en la que se insertó un sitio de acoplamiento para FLPe intercambio casete de recombinación mediada (RMCE) dentro del locus ROSA26 (R26),. Aquí, se presenta en un enfoque de estructura-función que permite la disección molecular de las distintas funcionalidades de una proteína multidominio. Con este fin, los ratones compatible-RMCE deben ser cruzadas con ratones KO y mESCs KO compatibles RMCE-a continuación deben ser aislados. A continuación, un panel de construcciones de rescate putativos se puede introducir en el locus R26 a través de RMCE targeting. Los ADNc de rescate candidatos se pueden insertar fácilmente entre sitios RMCE de los ataques de vectores de clonación que utilizan la recombinación. A continuación, mESCs KO se transfectan con el vector de direccionamiento en combinación con un plásmido de expresión de la recombinasa FLPe. RMCE reactiva el gen de resistencia a neomicina sin promotor en los sitios de acoplamiento ROSA26 y permite la selección del evento orientación correcta. De esta manera, se obtienen altas eficiencias de focalización cercanos al 100%, lo que permite la inserción de múltiples construcciones de rescate putativo de una manera semi-alto rendimiento. Por último, una multitud de construcciones de rescate impulsadas-R26 puede ser probado por su capacidad para rescatar el fenotipo que se observó en mESCs KO parentales. Presentamos un estudio de estructura-función de prueba de principio en la catenina p120 (p120ctn) mESCs KO utilizando la diferenciación del endodermo en cuerpos embrioides (EBS) como la lectura fenotípica. Este enfoque permite la identificación de dominios importantes, las vías aguas abajo putativo, y el punto de enfermedad relevantemutaciones que subyacen fenotipos KO para una determinada proteína.
Introduction
Se estima que los genomas de mamíferos contienen alrededor de 20.000 genes codificadores de proteínas. Splicing alternativo y modificaciones postraduccionales aumentan aún más el repertorio de proteínas. Las proteínas tienen una estructura modular 1 y a menudo contienen varios dominios de interacción, que permiten su reclutamiento en diferentes complejos de proteínas y su participación en múltiples procesos celulares 2. Un ejemplo es la proteína multifuncional llamada p120ctn. p120ctn está codificada por el gen Ctnnd1 y consta de un gran dominio central armadillo repetición flanqueado por un N-terminal y una región C-terminal. El dominio armadillo de p120ctn se une a un dominio yuxtamembrana altamente conservada de cadherinas clásicas, que están implicadas en la adhesión célula-célula, pero también se une al represor transcripcional Kaiso. El dominio N-terminal de p120ctn interactúa con diferentes quinasas, fosfatasas, pequeñas RhoGTPases, y asociada a los microtúbulos proteins 3. Curiosamente, como resultado de corte y empalme alternativo, isoformas p120ctn pueden ser generados a partir de cuatro codones de inicio alternativos 4. p120ctn isoforma 1A es la más larga, ya que se traduce de la más-5' codón de inicio y contiene el segmento N-terminal de longitud completa. En p120ctn isoformas 3 y 4, este segmento N-terminal se suprime parcialmente y completamente, respectivamente. La comprensión del papel exacto de proteínas (o isoformas de proteínas) y sus dominios en diferentes funciones celulares sigue siendo un reto.
La orientación de genes en mESCs permite el estudio de la función de una proteína a través de la supresión genética del gen correspondiente y ha contribuido ampliamente a la identificación de genes y las vías de desarrollo importantes y relevantes de la enfermedad. Este avance en la genética inversa fue el resultado de los avances en los campos de aislamiento mESC y la orientación de genes debido a la recombinación homóloga 5 </sarriba>. La recombinación homóloga es un proceso en el que fragmentos de ADN se intercambian entre dos restos nucleicos similares o idénticos después de doble hebra (ds) se rompe de ADN. Normalmente, HR es ineficiente debido a roturas de ADN bicatenario son poco frecuentes. Recientemente, la eficiencia del gen homología dirigida de orientación podría aumentarse usando nucleasas específicas de sitio 6, 7, pero, por desgracia, estos son propensos a los efectos fuera del objetivo 8. Una técnica más fiable para permitir la orientación de genes es RMCE, que se basa en los sistemas de recombinación específica de sitio tales como Cre / loxP o FLPe / FRT. LoxP y la secuencia Frt se encuentran en bacteriófago P1 y Saccharomyces cerevisiae, respectivamente, y se componen de 34 pares de bases, incluyendo una secuencia de pares de bases asimétrica 8 que determina la orientación del sitio. Por otra parte, la orientación de, por ejemplo, dos sitios loxP dentro de un tramo de ADN determinará si el ADN floxed convierte escindió o inversed tras la recombinación mediada por Cre 9. Por otra parte, Cre también puede inducir una translocación si dos sitios están localizados en diferentes cromosomas. RMCE se aprovecha de sitios de recombinación heterospecific que no reaccionan de forma cruzada y que están incrustados en un locus genómico. En presencia de un plásmido donante que contiene un fragmento de ADN flanqueado por los mismos sitios heterospecific, la recombinasa se insertar este fragmento de ADN en el locus genómico compatible-RMCE debido a la translocación de doble simultáneo (Figura 1). Aquí, los clones solamente correctamente orientados RMCE pueden hacer de resistencia a fármacos gracias a un promotor en el vector entrante que restaura una "atrapado" promotor-menos gen de resistencia a neomicina (Neo R) presente en el genoma R26 de las células de conexión (Figura 1) 10, 11. Esto resulta en una eficiencia muy alta focalización, a menudo cerca de 100% 11, </ sup> 12. En conclusión, la selección de base RMCE es altamente eficiente y se puede utilizar para estudios de estructura-funciones; sin embargo, se requiere un locus genómico pre-ingeniería.
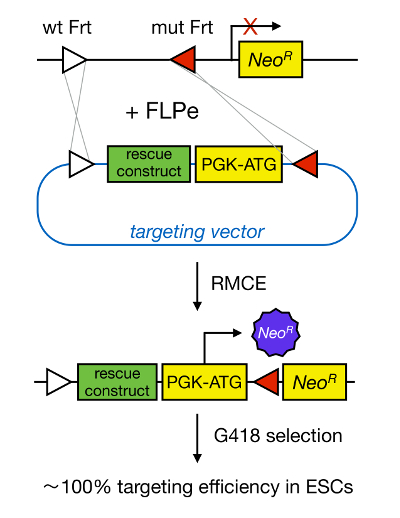
Figura 1. Representación esquemática de la orientación de RMCE mediada. RMCE permite el intercambio de segmentos de ADN a partir de un vector de dirección entrante a un locus genómico definido si ambos albergan dos sitios FRT heterospecific (representados por triángulos blanco y rojo). Además, el locus genómico de ingeniería contiene un gen truncado de resistencia a neomicina (Neo R) sin promotor y. Al proporcionar un promotor y el codón de inicio en el fragmento de ADN entrante, eventos de recombinación solamente correctas restaurar la resistencia a neomicina, dando como resultado altas eficiencias de focalización. Haga clic aquí para ver una versión más grande de tsu figura.
la ingeniería del genoma en mESCs permite la generación de ratones compatible-RMCE. En 1981, dos grupos tuvieron éxito en la captura de células pluripotentes de la masa celular interna (ICM) de blastocistos y en el mantenimiento de ellos en la cultura 13, 14. mESCs son capaces de auto-renovación y diferenciación en todos los tipos de células embrionarias y adultas, incluyendo el linaje de células germinales. Por lo tanto, la orientación de genes en mESCs permite estudios inversa genéticos mediante el desarrollo de ratones KO constitutivos o condicionales (utilizando el sistema cre / loxP). Sin embargo, la manera clásica para aislar células ES de ratón es muy ineficiente. Varias mejoras importantes han aumentado en gran medida la tasa de éxito para derivar líneas mESC, incluyendo el uso de un suero de reemplazo definido (SR) medio 15, alternando entre medio mESC que contenía suero bovino SR y fetal (FBS) 16, y el uso de fármacocompuestos lógicos como pluripotin o 2i 17. Pluripotin, una molécula sintética pequeña, permite la propagación de mESCs en un estado no diferenciado en ausencia del factor inhibidor de la leucemia (LIF) y fibroblastos embrionarios de ratón (MEFs) 18. Por último, se ha demostrado que mESCs se pueden aislar con una eficacia muy alta (cerca de 100%) cuando un / FBS protocolo alternancia medio SR se combina con LIF y pluripotin 19, 20. Estos protocolos permiten el aislamiento eficiente de mESCs KO compatible con RMCE que posteriormente pueden ser utilizados para estudios de estructura-función.
Este documento describe un método que permite identificar los dominios o residuos clave dentro de una proteína que son responsables de los procesos celulares específicos. Con este fin, una línea de tecnologías avanzadas que permiten el aislamiento eficiente mESC, montaje vector de dirección y orientación mESC era crearre. Como tales paneles grandes, con isoformas de proteínas, mutantes de dominio, y efectores aguas abajo pueden ser introducidas en mESCs KO y se pueden evaluar por su capacidad para rescatar el fenotipo vitro KO en.
Protocol
Representative Results
Discussion
Nuestro método de aislamiento mESC es fácil de usar y no requiere de técnicas y equipos avanzados, tales como la microcirugía de los blastocistos. Por lo tanto, esta tecnología se puede acceder a una gran parte de la comunidad científica. Cualquier persona con experiencia básica cultivo celular puede propagarse excrecencias ICM y establecer líneas mESCs. Sin embargo, el enrojecimiento y la manipulación de los blastocistos requiere algo de práctica. Una pipeta boca se utiliza para transferir los blastocistos y …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Agradecemos a Jinke D'Hont, Frederique Van Rockeghem, Natalie Farla, Kelly Lemeire y Riet De Rycke por su excelente soporte técnico. Agradecemos también a Eef PARTHOENS, Evelien Van Hamme y Amanda Goncalves del Fondo para el Bioimagen Núcleo del Centro de Investigación inflamación por su ayuda experta. Reconocemos los miembros de nuestro grupo de investigación valiosa para los debates. Este trabajo fue apoyado por la política belga Ciencia (BELSPO Interuniversitario de atracción Postes – Premio IAP VII-07 DevRepair; https://devrepair.be), por la Fundación Reina Elisabeth médica, Bélgica (GSKE 2008-2010; http: // www .fmre-gske.be), y mediante la acción concertada de investigación (GOA) 01G01908 de la Universidad de Gante, Bélgica (http://www.ugent.be/en/ghentuniv). SG es un compañero postdoctoral de los fondos de Investigación Flandes (FWO-V).
Materials
| ROSALUC Mice | made in house | frozen sperm available upon request | |
| R26-iPSC mice | made in house | frozen sperm available upon request | |
| Vessel probe | Fine Science Tools | 10160-13 | to check for copulation plugs |
| M2 medium | Sigma-Aldrich | M7167 | make aliquots and store at -20°C |
| Fine forceps (Dumont #5 Standard tip Student forceps) | Fine Science Tools | 11251-10 | spray with 70% EtOH before use (do not autoclave) |
| 23G needles | Fine-ject | 8697 | |
| 1-ml syringes | Soft-ject | 6680 | |
| 60-mm bacterial grade plates (for flushing) | Gosselin | BB60-01 | |
| Mouth pipette | made in house | see discussion | |
| Mouse embryonic fibroblasts (MEFs, TgN (DR4)1 Jae strain) | ATTC | SCRC-1045 | |
| TgN (DR4)1 Jae mice | The Jackson Laboratory | 3208 | |
| Mitomycin C | Sigma-Aldrich | M0503 | |
| Phosphate buffered saline (PBS) without calcium or magnesium | Gibco | 14190-094 | |
| Tg(DR4)1Jae/J mice | JAX | 3208 | mice that contain four drug-selectable genes and DR4 MEFS confers resistance to neomycin, puromycin, hygromycin and 6-thioguanine |
| 0.1% Gelatin | Sigma-Aldrich | G1393 | Dissolve 0.5 g in 500 ml distilled water, autoclave and store at 4°C. |
| Trypsin (0.25%) | Gibco | 25200-056 | |
| 2 μM pluripotin | Cayman Chemical | 10009557 | |
| pRMCE-DV1 | BCCM/LMBP collection | LMBP 08870 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| cre-excised pRMCE-DV1 | BCCM/LMBP collection | LMBP 08195 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| pCAG-FlpE-IRES-Puro-pA | Addgene | 20733 | |
| heat-shock competent DH5α bacteria | made in house | ||
| Gateway pDONR221 vector | Thermo Fisher | 12536-017 | contains kanamycin-resistance gene |
| BP clonase II mix | Thermo Fisher | 11789-020 | |
| LR clonase II mix | Thermo Fisher | 11791-020 | |
| Luria Broth (LB) | |||
| Ampicillin | |||
| Applied Biosystems 3730XL DNA Analyzer | Thermo Fisher | 3730XL | |
| G418 | Thermo Fisher | 11811-023 | |
| Lipofectamine 2000 transfection reagent | Thermo Fisher | 11668027 | |
| Lipofectamine LTX transfection reagent | Thermo Fisher | 15338100 | |
| Effectene transfection reagent | Qiagen | 301425 | |
| GATEWAY pENTR 1A vector | Thermo Fisher | A10462 | recombination-compatible vector |
| mouse monoclonal anti-p120ctn antibody | BD Transduction Laboratories | 610134 | |
| mouse monoclonal anti-Ecadherin antibody | BD Transduction Laboratories | 610181 | |
| General equipment: | |||
| Sterile dissection tools | fine scissors and forceps for dissecting the uterus | ||
| Sterile pipettes: 5 ml, 10 ml and 25 ml | |||
| 15-ml and 50-ml conical centrifuge tubes | |||
| 96-well culture plates V-shaped bottom and flat bottom) | |||
| Culture dishes: 24 wells, 12 wells and 6 wells | |||
| Multichannel pipettes (to pipette 30, 50, 100 and 200 μl) | |||
| Sterile multichannel reservoirs | |||
| Access to a laminar air flow | |||
| Access to an incubator at 37°C with 5% CO2 | |||
| Access to an inverted microscope | |||
| Access to a bench-top centrifuge | |||
| Access to a stereo microscope with transmitted-light | |||
| Culture media: | |||
| MEF Medium: | stored at 4°C; warm 30 min at 37°C before use | ||
| Dulbecco’s modified Eagle’s medium (DMEM) | Gibco | 41965-062 | |
| 10% fetal bovine serum (FBS) | Sigma-Aldrich | F-7524 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| Sodium pyruvate (0.4 mM) | Gibco | 11360-039 | |
| penicillin (100 U/ml) | Gibco | 15140-122 | |
| streptomycin (100 µg/ml) | Gibco | 15140-122 | |
| SR-based mESC medium: | stored at 4°C; warm 30 min at 37°C before use | ||
| DMEM/F12 | Gibco | 31330-038 | mixed in a 1:1 ratio |
| 15% knock-out serum replacement (SR ) | Gibco | 10828–028 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| 0.1 mM non-essential amino acids | Gibco | 11140-050 | |
| penicillin (100 U/ml) | Gibco | 15140-122 | |
| streptomycin (100 µg/ml) | Gibco | 15140-122 | |
| β-mercaptoethanol (0.1 mM) | Sigma-Aldrich | M 3148 | |
| 2,000 U/ml recombinant mouse LIF | (IRC/VIB Protein Service facility) | ||
| FBS-based mESC medium (similar to SR-based mESC medium): | stored at 4°C; warm 30 min at 37°C before use | ||
| Knockout DMEM | Gibco | 10829-018 | |
| 15% FBS | Hyclone | SH30070.03E | |
| Differention Medium | stored at 4°C; warm 30 min at 37°C before use | ||
| Iscove's Modified Dulbecco's Medium (IMDM) | Gibco | 21980-032 | |
| 15% FBS | Hyclone | SH30070.03E | |
| 5% CD Hybridoma Medium(1x) liquid | Gibco | 11279-023 | |
| 2 mM L-glutamine | Gibco | 25030-024 | |
| 0.4 mM 1-thioglycerol | Sigma-Aldrich | M-6145 | |
| 50 μg/ml ascorbic acid | Sigma-Aldrich | A-4544 | |
| penicillin (100 U/ml) | Gibco | 15140-122 | |
| streptomycin (100 µg/ml) | Gibco | 15140-122 | |
| 2i | |||
| 1 μM Erk inhibitor PD0325901 | Axon Medchem | Axon 1408 | |
| 3 μM Gsk3 inhibitor CHIR99021 | Axon Medchem | Axon 1386 |
References
- Gul, I. S., Hulpiau, P., Saeys, Y., van Roy, F. Metazoan evolution of the armadillo repeat superfamily. Cell Mol Life Sci. , (2016).
- Valenta, T., Hausmann, G., Basler, K. The many faces and functions of beta-catenin. EMBO J. 31, 2714-2736 (2012).
- Pieters, T., van Hengel, J., van Roy, F. Functions of p120ctn in development and disease. Front Biosci. 17, 760-783 (2012).
- Keirsebilck, A., et al. Molecular cloning of the human p120ctn catenin gene (CTNND1): Expression of multiple alternatively spliced isoforms. Genomics. 50, 129-146 (1998).
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6, 507-512 (2005).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Yang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 154, 1370-1379 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31, 822-826 (2013).
- Branda, C. S., Dymecki, S. M. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell. 6, 7-28 (2004).
- Sandhu, U., et al. Strict control of transgene expression in a mouse model for sensitive biological applications based on RMCE compatible ES cells. Nucleic Acids Res. 39, 1 (2010).
- Haenebalcke, L., et al. Efficient ROSA26-based conditional and/or inducible transgenesis using RMCE-compatible F1 hybrid mouse embryonic stem cells. Stem Cell Rev. 9, 774-785 (2013).
- Pieters, T., et al. p120 Catenin-Mediated Stabilization of E-Cadherin Is Essential for Primitive Endoderm Specification. PLoS Genet. 12, e1006243 (2016).
- Evans, M. J., Kaufman, M. H. Establishment in culture of pluripotential cells from mouse embryos. Nature. 292, 154-156 (1981).
- Martin, G. R. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells. Proc Natl Acad Sci U S A. 78, 7634-7638 (1981).
- Cheng, J., Dutra, A., Takesono, A., Garrett-Beal, L., Schwartzberg, P. L. Improved generation of C57BL/6J mouse embryonic stem cells in a defined serum-free media. Genesis. 39, 100-104 (2004).
- Bryja, V., et al. An efficient method for the derivation of mouse embryonic stem cells. Stem Cells. 24, 844-849 (2006).
- Ying, Q. L., et al. The ground state of embryonic stem cell self-renewal. Nature. 453, 519-523 (2008).
- Chen, S., et al. Self-renewal of embryonic stem cells by a small molecule. Proc Natl Acad Sci U S A. 103, 17266-17271 (2006).
- Pieters, T., et al. Efficient and User-Friendly Pluripotin-based Derivation of Mouse Embryonic Stem Cells. Stem Cell Rev. 8, (2012).
- Yang, W., et al. Pluripotin combined with leukemia inhibitory factor greatly promotes the derivation of embryonic stem cell lines from refractory strains. Stem Cells. 27, 383-389 (2009).
- Haenebalcke, L., et al. The ROSA26-iPSC Mouse: A Conditional, Inducible, and Exchangeable Resource for Studying Cellular (De)Differentiation. Cell Rep. 3, (2013).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25, 3745-3746 (1997).
- Nyabi, O., et al. Efficient mouse transgenesis using Gateway-compatible ROSA26 locus targeting vectors and F1 hybrid ES cells. Nucleic Acids Res. 37, 55 (2009).
- Katzen, F. Gateway((R)) recombinational cloning: a biological operating system. Expert Opin Drug Discov. 2, 571-589 (2007).
- Schaft, J., Ashery-Padan, R., vander Hoeven, F., Gruss, P., Stewart, A. F. Efficient FLP recombination in mouse ES cells and oocytes. Genesis. 31, 6-10 (2001).
- Spencer, H. L., et al. E-cadherin inhibits cell surface localization of the pro-migratory 5T4 oncofetal antigen in mouse embryonic stem cells. Mol Biol Cell. 18, 2838-2851 (2007).
- Stryjewska, A., et al. Zeb2 Regulates Cell Fate at the Exit from Epiblast State in Mouse Embryonic Stem Cells. Stem Cells. , (2016).
- Nagy, A., Gertsenstein, M., Vintersten, K., Behringer, R. . Manipulating the mouse embryo: A laboratory manual. , (2003).
- van den Berghe, V., et al. Directed migration of cortical interneurons depends on the cell-autonomous action of Sip1. Neuron. 77, 70-82 (2013).
- Li, J., et al. The EMT transcription factor Zeb2 controls adult murine hematopoietic differentiation by regulating cytokine signaling. Blood. , (2016).

