Struktur-funksjon studier i mus embryonale stamceller Bruke recombinase-mediert Cassette Veksling
Summary
Proteiner inneholder ofte flere domener som kan utøve ulike cellulære funksjoner. Gene knock-outs (KO) betrakter ikke dette funksjonelle mangfold. Her rapporterer vi en rekombinasjon-mediert kassett utveksling (RMCE) -basert struktur-funksjonsmetoden i KO-embryonale stamceller som gjør det mulig for den molekylære disseksjon av forskjellige funksjonelle domener eller varianter av et protein.
Abstract
Gene teknikk i museembryoer eller embryonale stamceller (mESCs) gjør det mulig å studere funksjonen av et gitt protein. Proteiner er arbeidshester av cellen og ofte bestå av flere funksjonelle domener, som kan påvirkes av post-translasjonelle modifikasjoner. Uttømming av hele proteinet i kondisjon eller konstitutiv knock-out (KO) mus som ikke tar hensyn til denne funksjonell diversitet og regulering. En Mesc linje og en avledet musemodell, hvor en festestedet for FLPe rekombinasjon-mediert kassett utveksling (RMCE) ble innført i ROSA26 (R26) locus, ble tidligere rapportert. Her rapporterer vi på en struktur-funksjon tilnærming som gjør det mulig for molekylær disseksjon av de ulike funksjonene i en multidomain protein. For å oppnå dette, må RMCE-kompatible mus krysses med KO mus og deretter RMCE-kompatible KO mESCs må isoleres. Deretter kan et panel av antatte rednings konstruksjoner innføres i R26 locus via RMCE targeting. Kandidat-rednings cDNA kan enkelt settes inn mellom RMCE områder av den målsøkende vektor ved hjelp av rekombinasjon kloning. Deretter blir KO mESCs transfektert med den målsøkende vektor i kombinasjon med et FLPe rekombinase ekspresjonsplasmid. RMCE reaktiverer den promoter-mindre neomycin-resistensgenet i ROSA26 tilkoblings områder og muliggjør valg av den riktige målretting arrangementet. På denne måten blir høy målretting virkningsgrader nær 100% oppnådd, som tillater innføring av flere mulige rednings konstruksjoner i en semi-høy gjennomstrømning måte. Endelig kan et mangfold av R26-drevne rednings konstruksjoner bli testet for deres evne til å redde den fenotype som ble observert i foreldre KO mESCs. Vi presenterer et bevis-for-prinsipp struktur-funksjon studie med p120 catenin (p120ctn) KO mESCs ved hjelp endoderm differensiering i embryoidlegemene (EBS) som fenotypiske avlesning. Denne tilnærmingen muliggjør identifikasjon av viktige områder, antatte nedstrøms trasé, og sykdomsrelevant punktmutasjoner som ligger til grunn KO fenotyper for et gitt protein.
Introduction
Det er anslått at pattedyr-genomer inneholder omtrent 20.000 protein-kodende gener. Alternativ spleising og posttranslasjonelle modifikasjoner ytterligere øke protein repertoaret. Proteiner har en modulær konstruksjon 1 og ofte inneholde flere interaksjons domener, noe som tillater deres rekruttering i forskjellige proteinkomplekser og deres deltagelse i flere cellulære prosesser 2. Et eksempel er den multifunksjonelle proteiner kjent p120ctn. p120ctn kodes for av Ctnnd1 gen og består av et stort sentralt armadillo gjenta domene flankert av en N-terminal og en C-terminal region. Armadillo domene av p120ctn binder seg til en høyt konservert juxtamembrane domene av klassiske cadheriner som er involvert i celle-celle-adhesjon, men det binder også til den transkripsjon repressoren Kaiso. Det N-terminale domenet av p120ctn samvirker med forskjellige kinaser, fosfataser, små RhoGTPases og mikrotubul-assosiert proteins tre. Det er interessant som et resultat av alternativ spleising, p120ctn isoformer kan genereres fra fire alternative startkodoner 4. p120ctn isoform 1A er den lengste, som det er oversatt fra den mest-5' startkodon og inneholder full-lengde N-terminal segmentet. I p120ctn isoformer 3 og 4 er denne N-terminale segment slettes helt henholdsvis delvis og. Å forstå den nøyaktige rolle av proteiner (eller protein isoformene) og deres domenene i forskjellige cellulære funksjoner er fortsatt en utfordring.
Gen-målsøking i mESCs gjør det mulig å studere funksjonen til et protein gjennom den genetiske delesjon av det tilsvarende genet og har bidratt mye til identifisering av utviklingsmessig viktige og sykdom-relevante gener og veier. Dette gjennombrudd i revers genetikk var et resultat av fremskrittene innen feltene Mesc isolasjon og gen-målsøking som følge av homolog rekombinasjon 5 </sopp>. Homolog rekombinasjon er en prosess i hvilken DNA-fragmenter blir utvekslet mellom to lignende eller identiske nukleinsyrer grupper etter dobbelt-trådet (ds) DNA-brudd. Normalt er HR ineffektiv fordi dsDNA pauser er sjeldne. Nylig, kan effektiviteten av homologi-rettet gen-målsøking økes ved å bruke stedsspesifikke nuklease 6, 7, men dessverre disse er tilbøyelige til off-target effekter 8. En mer pålitelig teknikk for å muliggjøre gen-målsøking er RMCE, som er basert på setespesifikke rekombinasjon systemer som Cre / loxP eller FLPe / Frt. LoxP og Frt sekvens er funnet i bakteriofag P1 og Saccharomyces cerevisiae, henholdsvis, og består av 34 bp, inkludert et asymmetrisk 8 bp-sekvens som bestemmer orienteringen av området. På den annen side, kan orienteringen av, for eksempel, to loxP-seter innenfor et DNA strekning vil avgjøre hvorvidt den floxed DNA blir kuttet ut eller jegnversed ved Cre-formidlet rekombinasjon 9. Videre kan Cre også indusere en trans hvis to stedene ligger på forskjellige kromosomer. RMCE utnyttet heterospecific rekombinasjonsseter som ikke kryssreagerer og som er innebygd i et genom-locus. I nærvær av en donor plasmid som inneholder et DNA-fragment flankert av de samme heterospecific steder, vil den rekombinasen sette inn dette DNA-fragment i RMCE-kompatible genomiske lokuset på grunn av dobbel-samtidig translokasjon (figur 1). Her kan bare korrekt RMCE rettede kloner gjengi medikamentresistens, takket være en promoter på den innkommende vektoren som gjenoppretter en "fanget" promoter-mindre neomycin resistensgenet (neo R) som er tilstede i R26 genomet av dokkings-celler (Figur 1) 10, 11. Dette resulterer i en meget høy effektivitet målretting, ofte nær 100% 11 </ sup> 12. Som konklusjon, er RMCE basert målretting svært effektiv og kan anvendes for struktur-funksjoner studier; Men det krever en pre-utviklet genomisk locus.
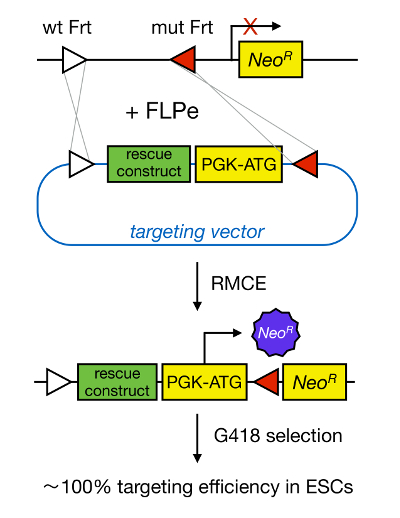
Figur 1. Skjematisk representasjon av RMCE-mediert målretting. RMCE tillater utveksling av DNA-segmenter fra en innkommende målsøkende vektor til en definert genomisk locus hvis begge huse to heterospecific FRT steder (vist med hvite og røde trekanter). I tillegg inneholder det konstruerte genomiske lokuset en promoterfrie og avkortet neomycin-resistens (Neo R) genet. Ved å tilveiebringe en promoter og startkodon i den innkommende DNA-fragment, kun korrekt rekombinasjonshendelser gjenopprette neomycinresistens, som resulterer i høye målretting effektivitet. Klikk her for å se en større versjon av thans skikkelse.
Genome engineering i mESCs tillater generering av RMCE-kompatibel mus. I 1981 ble to grupper lyktes å pluripotente celler fra den indre cellemasse (ICM) av blastocyster, og i å opprettholde dem i kultur 13, 14. mESCs er i stand til selvfornyelse og differensiering i alle typer av embryonale og voksne celler, inkludert bakterie-cellelinjen. Derfor, gen-målsøking i mESCs muliggjør revers-genetiske studier gjennom utviklingen av konstitutive eller betingede (ved bruk av Cre / loxP-system) KO-mus. Imidlertid er den klassiske metoden for å isolere muse-ES-celler er svært ineffektiv. Flere store forbedringer har sterkt øket suksessraten for utledning av Mesc linjer, inkludert bruk av et definert serum erstatning (SR) medium 15, alternerende mellom Mesc medium inneholdende SR og føtalt bovint serum (FBS) 16, og bruken av farmakologiskelogiske forbindelser slik som pluripotin eller 2i 17. Pluripotin, en liten syntetisk molekyl, tillater forplantning av mESCs i en udifferensiert tilstand i fravær av leukemi-inhiberende faktor (LIF) og muse-embryonale fibroblaster (MEFs) 18. Endelig har det vist seg at mESCs kan isoleres med en meget høy virkningsgrad (tilnærmet 100%) ved en SR / FBS-medium alternering protokoll er kombinert med LIF og pluripotin 19, 20. Disse protokollene muliggjøre effektiv isolering av RMCE-kompatible KO mESCs som senere kan brukes for struktur-funksjonsstudier.
Dette dokumentet beskriver en metode som gjør det mulig å identifisere de viktigste domener eller ester i et protein som er ansvarlig for spesifikke cellulære prosesser. For dette formål er en rørledning av avansert teknologi som muliggjør effektiv Mesc isolasjon, målsøkende vektor montasje, og Mesc målretting var skaped. Som sådanne store paneler med proteinisoformer, domene mutanter, og nedstrøms effektorer kan innføres i KO mESCs og kan evalueres for deres evne til å redde in vitro KO fenotype.
Protocol
Representative Results
Discussion
Vår Mesc isolasjonsmetoden er brukervennlig og krever ikke avanserte ferdigheter eller utstyr, som for eksempel mikro av blastocyster. Dermed er denne teknologien tilgjengelig for en stor andel av det vitenskapelige samfunn. Alle med grunnleggende cellekultur erfaring kan forplante ICM utvekster og etablere mESCs linjer. Men spyling og håndtering av blastocyster krever litt øvelse. En munn pipette brukes til å overføre blastocyster, og består av en mikropipette, en mikropipette holder, rør, og en sugemunnstykke <…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Vi takker Jinke D'Hont, Frederique Van Rockeghem, Natalie Farla, Kelly Lemeire, og Riet De RYCKE for deres utmerkede teknisk støtte. Vi takker også EEF Parthoens, Evelien Van Hamme og Amanda Goncalves fra Bioimaging Kjerne Facility av Betennelse Research Center for deres eksperthjelp. Vi erkjenner medlemmer av vår forskningsgruppe for verdifulle diskusjoner. Dette arbeidet ble støttet av den belgiske Science Policy (Belspo Interuniversity attraksjon polakker – Award IAP VII-07 DevRepair, https://devrepair.be), av dronning Elisabeth Medical Foundation, Belgia (GSKE 2008-2010; http: // www .fmre-gske.be), og ved å samordne forskning handlinger (GOA 01G01908) av Ghent universitet, Belgia (http://www.ugent.be/en/ghentuniv). SG er en postdoktor av Flandern forskningsmidler (FWO-V).
Materials
| ROSALUC Mice | made in house | frozen sperm available upon request | |
| R26-iPSC mice | made in house | frozen sperm available upon request | |
| Vessel probe | Fine Science Tools | 10160-13 | to check for copulation plugs |
| M2 medium | Sigma-Aldrich | M7167 | make aliquots and store at -20°C |
| Fine forceps (Dumont #5 Standard tip Student forceps) | Fine Science Tools | 11251-10 | spray with 70% EtOH before use (do not autoclave) |
| 23G needles | Fine-ject | 8697 | |
| 1-ml syringes | Soft-ject | 6680 | |
| 60-mm bacterial grade plates (for flushing) | Gosselin | BB60-01 | |
| Mouth pipette | made in house | see discussion | |
| Mouse embryonic fibroblasts (MEFs, TgN (DR4)1 Jae strain) | ATTC | SCRC-1045 | |
| TgN (DR4)1 Jae mice | The Jackson Laboratory | 3208 | |
| Mitomycin C | Sigma-Aldrich | M0503 | |
| Phosphate buffered saline (PBS) without calcium or magnesium | Gibco | 14190-094 | |
| Tg(DR4)1Jae/J mice | JAX | 3208 | mice that contain four drug-selectable genes and DR4 MEFS confers resistance to neomycin, puromycin, hygromycin and 6-thioguanine |
| 0.1% Gelatin | Sigma-Aldrich | G1393 | Dissolve 0.5 g in 500 ml distilled water, autoclave and store at 4°C. |
| Trypsin (0.25%) | Gibco | 25200-056 | |
| 2 μM pluripotin | Cayman Chemical | 10009557 | |
| pRMCE-DV1 | BCCM/LMBP collection | LMBP 08870 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| cre-excised pRMCE-DV1 | BCCM/LMBP collection | LMBP 08195 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| pCAG-FlpE-IRES-Puro-pA | Addgene | 20733 | |
| heat-shock competent DH5α bacteria | made in house | ||
| Gateway pDONR221 vector | Thermo Fisher | 12536-017 | contains kanamycin-resistance gene |
| BP clonase II mix | Thermo Fisher | 11789-020 | |
| LR clonase II mix | Thermo Fisher | 11791-020 | |
| Luria Broth (LB) | |||
| Ampicillin | |||
| Applied Biosystems 3730XL DNA Analyzer | Thermo Fisher | 3730XL | |
| G418 | Thermo Fisher | 11811-023 | |
| Lipofectamine 2000 transfection reagent | Thermo Fisher | 11668027 | |
| Lipofectamine LTX transfection reagent | Thermo Fisher | 15338100 | |
| Effectene transfection reagent | Qiagen | 301425 | |
| GATEWAY pENTR 1A vector | Thermo Fisher | A10462 | recombination-compatible vector |
| mouse monoclonal anti-p120ctn antibody | BD Transduction Laboratories | 610134 | |
| mouse monoclonal anti-Ecadherin antibody | BD Transduction Laboratories | 610181 | |
| General equipment: | |||
| Sterile dissection tools | fine scissors and forceps for dissecting the uterus | ||
| Sterile pipettes: 5 ml, 10 ml and 25 ml | |||
| 15-ml and 50-ml conical centrifuge tubes | |||
| 96-well culture plates V-shaped bottom and flat bottom) | |||
| Culture dishes: 24 wells, 12 wells and 6 wells | |||
| Multichannel pipettes (to pipette 30, 50, 100 and 200 μl) | |||
| Sterile multichannel reservoirs | |||
| Access to a laminar air flow | |||
| Access to an incubator at 37°C with 5% CO2 | |||
| Access to an inverted microscope | |||
| Access to a bench-top centrifuge | |||
| Access to a stereo microscope with transmitted-light | |||
| Culture media: | |||
| MEF Medium: | stored at 4°C; warm 30 min at 37°C before use | ||
| Dulbecco’s modified Eagle’s medium (DMEM) | Gibco | 41965-062 | |
| 10% fetal bovine serum (FBS) | Sigma-Aldrich | F-7524 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| Sodium pyruvate (0.4 mM) | Gibco | 11360-039 | |
| penicillin (100 U/ml) | Gibco | 15140-122 | |
| streptomycin (100 µg/ml) | Gibco | 15140-122 | |
| SR-based mESC medium: | stored at 4°C; warm 30 min at 37°C before use | ||
| DMEM/F12 | Gibco | 31330-038 | mixed in a 1:1 ratio |
| 15% knock-out serum replacement (SR ) | Gibco | 10828–028 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| 0.1 mM non-essential amino acids | Gibco | 11140-050 | |
| penicillin (100 U/ml) | Gibco | 15140-122 | |
| streptomycin (100 µg/ml) | Gibco | 15140-122 | |
| β-mercaptoethanol (0.1 mM) | Sigma-Aldrich | M 3148 | |
| 2,000 U/ml recombinant mouse LIF | (IRC/VIB Protein Service facility) | ||
| FBS-based mESC medium (similar to SR-based mESC medium): | stored at 4°C; warm 30 min at 37°C before use | ||
| Knockout DMEM | Gibco | 10829-018 | |
| 15% FBS | Hyclone | SH30070.03E | |
| Differention Medium | stored at 4°C; warm 30 min at 37°C before use | ||
| Iscove's Modified Dulbecco's Medium (IMDM) | Gibco | 21980-032 | |
| 15% FBS | Hyclone | SH30070.03E | |
| 5% CD Hybridoma Medium(1x) liquid | Gibco | 11279-023 | |
| 2 mM L-glutamine | Gibco | 25030-024 | |
| 0.4 mM 1-thioglycerol | Sigma-Aldrich | M-6145 | |
| 50 μg/ml ascorbic acid | Sigma-Aldrich | A-4544 | |
| penicillin (100 U/ml) | Gibco | 15140-122 | |
| streptomycin (100 µg/ml) | Gibco | 15140-122 | |
| 2i | |||
| 1 μM Erk inhibitor PD0325901 | Axon Medchem | Axon 1408 | |
| 3 μM Gsk3 inhibitor CHIR99021 | Axon Medchem | Axon 1386 |
References
- Gul, I. S., Hulpiau, P., Saeys, Y., van Roy, F. Metazoan evolution of the armadillo repeat superfamily. Cell Mol Life Sci. , (2016).
- Valenta, T., Hausmann, G., Basler, K. The many faces and functions of beta-catenin. EMBO J. 31, 2714-2736 (2012).
- Pieters, T., van Hengel, J., van Roy, F. Functions of p120ctn in development and disease. Front Biosci. 17, 760-783 (2012).
- Keirsebilck, A., et al. Molecular cloning of the human p120ctn catenin gene (CTNND1): Expression of multiple alternatively spliced isoforms. Genomics. 50, 129-146 (1998).
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6, 507-512 (2005).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Yang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 154, 1370-1379 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31, 822-826 (2013).
- Branda, C. S., Dymecki, S. M. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell. 6, 7-28 (2004).
- Sandhu, U., et al. Strict control of transgene expression in a mouse model for sensitive biological applications based on RMCE compatible ES cells. Nucleic Acids Res. 39, 1 (2010).
- Haenebalcke, L., et al. Efficient ROSA26-based conditional and/or inducible transgenesis using RMCE-compatible F1 hybrid mouse embryonic stem cells. Stem Cell Rev. 9, 774-785 (2013).
- Pieters, T., et al. p120 Catenin-Mediated Stabilization of E-Cadherin Is Essential for Primitive Endoderm Specification. PLoS Genet. 12, e1006243 (2016).
- Evans, M. J., Kaufman, M. H. Establishment in culture of pluripotential cells from mouse embryos. Nature. 292, 154-156 (1981).
- Martin, G. R. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells. Proc Natl Acad Sci U S A. 78, 7634-7638 (1981).
- Cheng, J., Dutra, A., Takesono, A., Garrett-Beal, L., Schwartzberg, P. L. Improved generation of C57BL/6J mouse embryonic stem cells in a defined serum-free media. Genesis. 39, 100-104 (2004).
- Bryja, V., et al. An efficient method for the derivation of mouse embryonic stem cells. Stem Cells. 24, 844-849 (2006).
- Ying, Q. L., et al. The ground state of embryonic stem cell self-renewal. Nature. 453, 519-523 (2008).
- Chen, S., et al. Self-renewal of embryonic stem cells by a small molecule. Proc Natl Acad Sci U S A. 103, 17266-17271 (2006).
- Pieters, T., et al. Efficient and User-Friendly Pluripotin-based Derivation of Mouse Embryonic Stem Cells. Stem Cell Rev. 8, (2012).
- Yang, W., et al. Pluripotin combined with leukemia inhibitory factor greatly promotes the derivation of embryonic stem cell lines from refractory strains. Stem Cells. 27, 383-389 (2009).
- Haenebalcke, L., et al. The ROSA26-iPSC Mouse: A Conditional, Inducible, and Exchangeable Resource for Studying Cellular (De)Differentiation. Cell Rep. 3, (2013).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25, 3745-3746 (1997).
- Nyabi, O., et al. Efficient mouse transgenesis using Gateway-compatible ROSA26 locus targeting vectors and F1 hybrid ES cells. Nucleic Acids Res. 37, 55 (2009).
- Katzen, F. Gateway((R)) recombinational cloning: a biological operating system. Expert Opin Drug Discov. 2, 571-589 (2007).
- Schaft, J., Ashery-Padan, R., vander Hoeven, F., Gruss, P., Stewart, A. F. Efficient FLP recombination in mouse ES cells and oocytes. Genesis. 31, 6-10 (2001).
- Spencer, H. L., et al. E-cadherin inhibits cell surface localization of the pro-migratory 5T4 oncofetal antigen in mouse embryonic stem cells. Mol Biol Cell. 18, 2838-2851 (2007).
- Stryjewska, A., et al. Zeb2 Regulates Cell Fate at the Exit from Epiblast State in Mouse Embryonic Stem Cells. Stem Cells. , (2016).
- Nagy, A., Gertsenstein, M., Vintersten, K., Behringer, R. . Manipulating the mouse embryo: A laboratory manual. , (2003).
- van den Berghe, V., et al. Directed migration of cortical interneurons depends on the cell-autonomous action of Sip1. Neuron. 77, 70-82 (2013).
- Li, J., et al. The EMT transcription factor Zeb2 controls adult murine hematopoietic differentiation by regulating cytokine signaling. Blood. , (2016).

