Kvantifiera mikroorganismer vid låga koncentrationer använder digitala holografisk mikroskopi (DHM)
Summary
Digitala holografisk mikroskopi (DHM) är en volymetriska teknik som tillåter imaging prover 50-100 X tjockare än brightfield mikroskopi på jämförbar upplösning, med fokusering utförs efterbearbetning. DHM används här för att identifiera, räkna, och spårning mikroorganismer vid mycket låga tätheter och jämfört med optisk densitet mätningar, plattan, antal och direkt.
Abstract
Korrekt upptäcka och räknar glesa bakteriell prover har många tillämpningar i mat, dryck och farmaceutiska beredningsindustrin i medicinsk diagnostik, och för livet upptäckt av robotic uppdrag till andra planeter och månar i solsystemet. För närvarande räknas glesa bakteriell prover av kultur bordläggning eller epifluorescence mikroskopi. Kultur plattor kräver lång inkubation times (dagar till veckor), och epifluorescence mikroskopi kräver omfattande färgning och koncentrationen i provet. Här visar vi hur du använder off-axeln digital holografisk mikroskopi (DHM) att räkna upp bakterier i mycket utspädd kulturer (100-104 celler/mL). Först diskuteras byggandet av den anpassade DHM, tillsammans med detaljerade instruktioner om att bygga en låg kostnad instrument. Principerna för holografi diskuteras, och en statistisk modell används för att uppskatta hur länge videor bör vara att identifiera celler, baserat på optisk prestandaegenskaper för instrumentet och koncentrationen av bakteriell lösningen (tabell 2) . Video upptäckt av celler 105, 104, 103och 100 celler/mL demonstreras i realtid med hjälp av un-rekonstruerade hologram. Rekonstruktion av amplitud och fas bilder demonstreras med en öppen källkod-paketet.
Introduction
Bestämning av korrekt bakteriell räknas i mycket utspädda prover är avgörande i många applikationer: några exempel är vatten och mat kvalitet analys1,2,3; patogener i blod, ryggmärgsvätska eller sputum4,5. tillverkning av farmaceutiska produkter, inklusive sterilt vatten6; och miljörörelsen analys i näringsfattiga miljöer såsom den öppna ocean och sediment7,8,9. Det ökar också intresset för upptäckt av möjliga bevarade mikrobiellt liv på de isiga moonsna av Jupiter och Saturnus, särskilt Europa10,11 och Enceladus12,13, 14, som är känt för att ha under markytan flytande världshaven. Eftersom inga uppdrag sedan Viking i 1978 har försökt att hitta bevarade liv på en annan planet, har det varit begränsad utveckling av teknik och instrument för bakteriell identifiering och inventering under utrymme uppdrag15.
Traditionella metoder för plattspridning hitta endast odlingsbara celler, som kan representera en minoritet av arter i miljömässiga stammar, ibland < 1%16. Plattorna kräver dagar eller veckor för inkubation för maximal framgång, beroende på belastningen. Epifluorescence mikroskopi har till stor del ersatt plattan räknas som den gyllene standarden för snabb och noggrann mikrobiell uppräkning. Nucleic-acid-märkning fluorescerande färger såsom 4′, 6-diamidin-2-fenylindol dihydroklorid (DAPI), SYBR Green eller akridin orange som binder till nukleinsyror är typiska färgämnen används17,18,19 , även om många studier använder fluorescerande indikatorer gram logga20,21,22,23,24. Med dessa metoder utan före koncentrationen steg leder till detektionsgränser (LoDs) ~ 105 celler per mL. Förbättringar i LoD är möjligt med hjälp av filtrering. Ett flytande prov är vakuum-filtrerad på ett membran, oftast polykarbonat och helst svart att minska bakgrunden. Låg-bakgrund färgämnen som DNA fläckarna nämns ovan kan appliceras direkt på de filter25. För noggrann inventering av ögat, ~ 105 celler krävs per filter, vilket betyder att för prover mer utspädd än ~ 105 celler per mL, betydande provvolymer måste samlas in och filtreras. Laserskanning enheter har utvecklats för att systematiskt utforska alla regioner i filtret och därmed minska antalet celler krävs för inventering, driver detektionsgränser ner till ~ 102 celler per mL26. Men dessa är inte tillgängliga i de flesta laboratorier, och kräver sofistikerade hårdvara samt mjukvara som tillåter expert bekräftelse som observerats partiklar är bakterier och inte skräp.
För referens, vuxna med sepsis brukar börja visar symtom på < 100 celler/mL blod och spädbarn på < 10 celler/mL. En blodprovstagning från en vuxen tar 10 mL, och från ett spädbarn, 1 mL. PCR-baserade metoder hämmas genom närvaron av mänskliga och icke-patogena floran DNA och PCR-hämmande komponenter i blodet27,28. Trots en mängd ny teknik fortfarande kulturer den gyllene standarden för diagnos av blodinfektioner, särskilt i mer lantliga områden eller utvecklingsländer. För påvisande av liv på andra planeter, kan termodynamiska beräkningar uppskatta energi budgeten för livet och därmed den förväntade möjligt biomassan. 1 – 100 celler/mL förväntas vara thermodynamically rimlig på Europa29. Det kan lätt ses från dessa siffror att upptäckten av ett mycket litet antal celler i stora mängder vattenlösning är ett viktigt olösta problem.
I detta papper, vi visar upptäckt av Serratia marcescens och Shewanella oneidensis (wild-type och icke-rörliga mutant) vid koncentrationer av 105, 104, 103och 100 celler/mL med hjälp av en off-axeln Digital holografisk Mikroskop (DHM). Den viktigaste fördelen med DHM över traditionella ljusmikroskopi är den samtidiga avbildning av en tjock provvolymen med hög upplösning — i detta genomförande, i provkammaren var 0,8 mm tjock. Dessa prov kammare konstruerades av den mjuka-litografin av Polydimetylsiloxan (PDMS) från en precision-frästa aluminium mögel med en tolerans på ± 50 µm. Detta är en cirka 100-faldig förbättring i djup i fältet över high-power ljusmikroskopi. DHM ger också kvantitativa fas information, vilket möjliggör mätningar av optiska ljuspassagelängden (produkt av brytningsindex och tjocklek). DHM och liknande metoder har använts för övervakning av bakterier och jäst cellcykeln och beräkning av bakteriell torr massa30,31,32. scattering skillnader kan även användas för att differentiera bakteriestammar33.
De instrument som vi använder är specialbyggda specifikt för användning med mikroorganismer, som tidigare publicerade34,35, och dess design och konstruktion är visade och diskuterade. Aqueous lösningar levereras kontinuerligt till en 0,25 µL volym provkammare via sprutpump; flödet bestäms av kamerans bildfrekvens för att säkerställa avbildning av den hela provvolymen. En statistisk beräkning förutspår antalet provvolymer som måste avbildas för att upptäcka ett betydande antal celler vid en given koncentration.
För cell-påvisande program var rekonstruktion av hologram i amplitud och fas bilder inte skyldig; analysen utfördes på raw hologrammet. Detta sparar betydande dataresurser och diskutrymme: ett 500 Mb hologram video blir 1-2 Tb när rekonstrueras. Dock diskutera vi återuppbyggnad genom djup av provet att bekräfta att hologram motsvarar önskad arten. Ett viktigt inslag i DHM är dess förmåga att övervaka både intensitet och fas av bilderna. Organismer som är nästan transparent i intensitet (såsom de flesta biologiska celler) visas tydligt i fas. Eftersom det är en etikett-fri teknik, används inga färgämnen. Detta är en endvantage för möjliga rymdfärder program, eftersom färgämnen inte kan överleva villkoren för ett uppdrag och – ännu viktigare — inte kan antas för att arbeta med utomjordiska organismer, som inte får använda DNA eller RNA för kodning. Det är också en fördel för arbete i extrema miljöer såsom Arktis och Antarktis, där färgämnen kan vara svårt att få till fjärrplatsen och försämras vid förvaring. Rekonstruktion av bilderna i fas och amplitud utförs med hjälp av en programvara med öppen källkod paket som vi har gjort tillgänglig på GitHub (schampo) eller ImageJ.
Protocol
Representative Results
Discussion
Numeriska rekonstruktion av hologram: för numeriska återuppbyggnaden av hologram, används metoden kantiga spektrum (ASM). Detta innebär faltningen av hologrammet med Greens funktion för DHMEN. Den komplexa wavefront av bilden på en viss fokalplan kan beräknas genom att anställa Fourier faltning sats enligt följande:
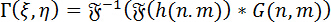 (1)
(1)
Där <img alt="Equation 3" src="/files/ftp_upload/56343/5…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Författarna erkänner Gordon och Betty Moore Foundation bidrag 4037 och 4038 till California Institute of Technology för att finansiera detta arbete.
Materials
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
References
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples – Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn’s moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).

