Dissection multi-signalisation Complexes protéiques par Purification d’affinité de complémentation bimoléculaire (BiCAP)
Summary
Cet article décrit le protocole pour la Purification d’affinité de complémentation bimoléculaire (BiCAP). Cette nouvelle méthode facilite l’isolement spécifique et la caractérisation de la protéomique en aval de toute deux protéines qui interagissent, tandis que hors ONU complexée protéines individuelles ainsi que des complexes formés avec des partenaires de liaison concurrentes.
Abstract
L’Assemblée de complexes protéiques est un mécanisme central qui sous-tendent le règlement de nombreuses voies de signalisation de cellulaire. Un objectif majeur de la recherche biomédicale est déchiffrer comment ces complexes de protéine dynamique agissent pour intégrer les signaux provenant de plusieurs sources afin de diriger une réponse biologique spécifique, et comment cela devient déréglementé dans bien des cas de maladie. Malgré l’importance de ce mécanisme biochimique essentiel, il y a un manque de techniques expérimentales qui peuvent faciliter la déconvolution spécifique et sensible de ces complexes de signalisation moléculaires multiples.
Ici, cette lacune est adressée par la combinaison d’un dosage de complémentation protéique avec une conformation spécifique nanobody, que nous avons appelé bimoléculaire Purification d’affinité de complémentation (BiCAP). Cette nouvelle technique facilite l’isolation spécifique et la caractérisation de la protéomique en aval de n’importe quelle paire de protéines qui interagissent, à l’exclusion des protéines individuelles ONU complexée et complexes qui se forment avec des partenaires de liaison concurrentes.
La technique de BiCAP s’adapte à un large éventail d’essais expérimentaux en aval, et le haut degré de spécificité offerte par cette technique permet plus nuancées enquêtes sur la mécanique de l’assemblage complexe de protéine est actuellement possible à l’aide techniques de purification d’affinité standard.
Introduction
Assemblage complexe de protéine est un processus clé dans le maintien de la spécificité spatio-temporelle de nombreuses signalisation des voies1,2. Bien que la nature essentielle de ce rôle réglementaire est largement reconnue, il y a un manque de techniques expérimentales disponibles à scruter ces complexes. La plupart des études interactomique mettre l’accent sur les interactions avec les protéines individuelles, ou l’enrichissement séquentiel des composants complexes. Nous présentons ici une technique pour l’isolation d’un dimère de protéine spécifique tout en excluant les portions individuelles de la protéines composant, mais aussi les complexes formés avec liant les partenaires3en compétition. Nous avons appelé cette technique bimoléculaire Purification d’affinité de complémentation (BiCAP), telle qu’elle est une combinaison d’un dosage de complémentation de fragment protéique déjà existantes, bimoléculaire complémentation de Fluorescence (BiFC), avec la nouvelle utilisation d’un conformation spécifique nanobody recombinante vers GFP et de ses dérivés (voir Table des matières).
Une analyse de complémentation de fragment de protéine typique se fonde sur l’expression des protéines « appât » et « proie » fusionnés pour fractionner des fragments de reporters comme luciférase,4,5de la β-galactosidase ou protéine fluorescente verte (GFP)6 ( Figure 1 a). Par le biais de l’interaction des protéines appât et proies, les domaines de journaliste de split sont encouragés à replier dans une structure fonctionnelle, qui permet l’interaction de l’appât et de protéines pour être visualisées ou quantifié la proie. BiCAP a été adapté d’une version de cette technique qui fait utiliser des fragments de la variante GFP Venus. Protéine fluorescente complémentation dosages sont une méthode populaire pour la visualisation des interactions protéines-protéines dans une cellule en direct, mais jusqu’à présent ont été limitées à cette un fonction7. BiCAP représente une avancée significative à cet égard, car cette technique permet non seulement pour la visualisation, mais aussi l’isolement et l’interrogatoire de l’interaction protéine-protéine qui en résulte.
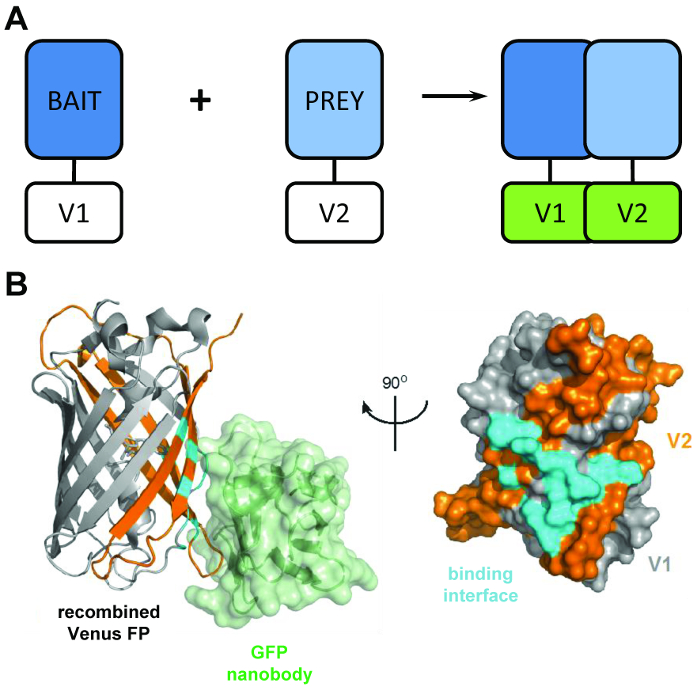
Figure 1 : le capital structurel derrière la technique BiCAP. (A) un schéma décrivant l’entité de sécurité derrière l’affichage de complémentation de fluorescence bimoléculaire les protéines « appât » et « proie » marquées par le N-terminal V1 ou V2 C-terminal des fragments de la protéine pleine longueur de Vénus. (B) analyse structurale de l’interface d’interaction (cyan) entre la GFP nanobody (vert) et Vénus recombinée, montrant la position de la V1 (gris) et des fragments de V2 (orange) (APB adhésion 3OGO). Ce chiffre est republié fromCroucher Al3 reproduit avec la permission de l’AAAS. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Le BiCAP technique fait usage de deux fragments non fluorescent de Vénus (nommé V1 et V2), qui associent avec un faible degré d’affinité, à moins qu’une interaction se produit entre leurs partenaires en matière de fusion. Dans ce cas, les domaines deux split replier dans la structure fonctionnelle de la β-baril du fluorophore (Figure 1 b)6. L’innovation majeure de BiCAP provient de l’introduction de la recombinaison nanobody GFP, qui reconnaît un épitope en trois dimensions sur le β-Canon de GFP (et des variantes telles que Vénus) qui n’est présent que sur le fluorophore recombiné et correctement plié ( Figure 1 b)8. Fondamentalement, la GFP nanobody ne lie pas à un des fragments individuels Venus. Ceci facilite l’isolement des dimères de protéine qu’après que les deux protéines ont formé un complexe de leur propre gré, conduisant à des résultats plus représentatives que celles acquises des méthodes qui rendent l’utilisation de chimiquement induite, forcé les interactions9.
BiCAP est une technique puissante qui se concentre spécifiquement sur les complexes de protéines multiples, qui peuvent potentiellement être combinées avec un certain nombre d’applications en aval afin d’améliorer la granularité de notre compréhension du rôle à jouer ces complexes dans la transduction du signal . Elle englobe également la caractéristique importante de ce qui permet la visualisation des interactions de protéine in situ. A ce jour, BiCAP a été démontrée comme une méthode efficace d’analyse de l’interactome des récepteurs tyrosine kinase (RTK) dimères3, mais la capacité d’adaptation de cette méthode signifie qu’il pourra être adopté dans presque n’importe quel contexte d’interaction de protéine.
Protocol
Representative Results
Discussion
BiCAP est une méthode puissante pour isoler les dimères de protéine spécifique tout en excluant les composants individuels et leurs concurrents de partenaires de liaison3. BiCAP repose sur l’adaptation d’un dosage de complémentation protéique de fluorescence appelé BiFC6. Les méthodes existantes, y compris les essais de ligature BiFC et de proximité, ont été largement utilisés pour visualiser et quantifier les interactions de protéine dans des cellules viva…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Entiereté est un camarade de NSW Institut du Cancer et D.N.S était auparavant un Cancer Institute NSW Fellow. Les résultats de la recherche présentées dans ce manuscrit ont été financés par le Cancer Institute NSW (13/RSF/1-02 et 09/CDF/2-39), NHMRC (projet Grant GNT1052963), Science Foundation Ireland (11/GSS/B2157), NSW Office of Science et recherche médicale, les famille Bourse et Mostyn Family Foundation. J.F.H. et R.S. ont été destinataires d’une sentence Postgraduate australien.
Materials
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid – Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) – 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).

