해 다중 단백질 신호 복합물 분자 보완성 친 화력 정화 (BiCAP)에 의해
Summary
이 원고는 분자 보완성 친 화력 정화 (BiCAP)에 대 한 프로토콜을 설명합니다. 이 새로운 방법은 경쟁 바인딩 파트너와 함께 형성 된 복합물 뿐만 아니라 유엔 complexed 개별 단백질을 제외 하는 동안 특정 격리 및 어떤 두 상호 작용 단백질의 다운스트림 proteomic 특성화를 촉진 한다.
Abstract
단백질 복합물의 조립은 많은 세포 신호 통로의 규칙을 기본 중앙 메커니즘입니다. 어떻게 이러한 동적 단백질 복합물 특정 생물학 응답을 직접 하기 위해 여러 소스 로부터 신호를 통합 하 고 어떻게이 많은 질병 설정에서 폐지 된다 생물 의학 연구의 주요 초점 해독 됩니다. 이 주요 생 화 학적 메커니즘의 중요성에도 불구 하 고이 다 분자 신호 복합물의 구체적이 고 민감한 deconvolution을 촉진할 수 있다 실험적인 기술의 부족이 이다.
여기이 단점 분자 보완성 친 화력 정화 (BiCAP) 불리는 우리 나 란 특정 nanobody는 단백질 보완성 분석 결과의 조합을 통해 해결 됩니다. 이 새로운 기술은 특정 격리 및 유엔 complexed 개별 단백질과 경쟁 바인딩 파트너와 함께 형성 하는 복합물의 배타에 상호 작용 단백질의 어떤 쌍 든 지의 다운스트림 proteomic 특성화를 촉진 한다.
BiCAP 기술은 다양 한 다운스트림 실험적인 분석 실험에 이며이 기술에서 제공 하는 특이성의 고차 수 더 nuanced 조사 단백질 복잡 한 어셈블리의 역학에 현재 가능한 것 보다 사용 하 여 표준 선호도 정화 기술입니다.
Introduction
단백질 복잡 한 어셈블리 많은 신호 경로1,2의 spatiotemporal 특이성을 유지 하는 핵심 과정 이다. 이 규제 역할의 중요 한 특성은 넓게 인식 된다, 그러나 실험적인 기술이이 단지 유심히 사용할 수의 부족이 이다. 대부분 상호작용체학 연구 개별 단백질, 또는 개별 복잡 한 부품의 순차 농축 상호 작용에 초점. 여기 선물이 특정 단백질 이합체의 고립에 대 한 기술 파트너3바인딩 경쟁 형성 복합물 뿐만 아니라 구성 요소 단백질의 개별 moieties 제외 하는 동안. 우리는이 기술을 라는 분자 보완성 친 화력 정화 (BiCAP), 그것은 기존 단백질 조각 보완성 분석 결과의 분자 형광 보완성 (BiFC), 조합의 소설 사용을 GFP와 그 파생 상품 나 란 특정 재조합 nanobody (참조 테이블의 재료).
전형적인 단백질 조각 보완성 분석 결과 분할 luciferase4, β-galactosidase5, 녹색 형광 단백질 (GFP)6 ( 등 기자 들의 파편을 융합 하는 “미끼” 및 “먹이” 단백질의 표현에 사용 그림 1A). 미끼와 먹이 단백질의 상호 작용을 통해 분할 기자 도메인, 미끼의 상호 작용을 허용 하는 기능 구조로 refold 및 시각에 계량 단백질을 먹이 것이 좋습니다. BiCAP 만든이 기술은의 버전에서 적응 시켰다 GFP 변종 비너스의 조각을 사용. 형광 단백질 보완성 분석 실험은 살아있는 세포에 있는 단백질 단백질 상호 작용을 시각화를 위한 인기 있는 방법 하지만 지금까지이 하나의 함수7로 제한 되었습니다. BiCAP 나타냅니다 중요 한 사전을 이와 관련,이 기술은 뿐만 아니라 시각화, 하지만 또한 격리와 심문 결과 단백질 단백질 상호 작용의 수 있습니다.
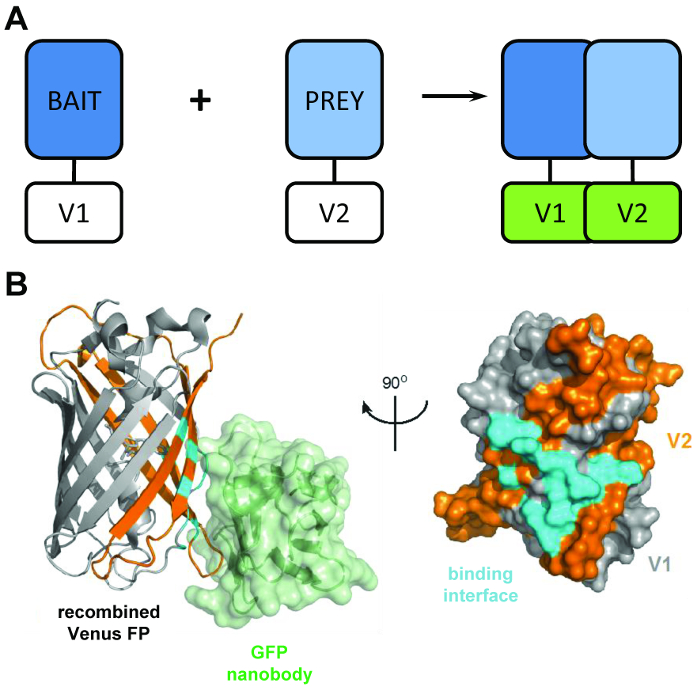
그림 1: BiCAP 기술 뒤에 구조 교장. (A) 회로도 분자 형광 보완성 표시 뒤에 교장 개요 ‘미끼’ 그리고 ‘먹이’ 단백질 태그로 N 맨끝 V1 또는 v 2의 C-터미널 전체 길이 금성 단백질의 파편. GFP nanobody (녹색)과 재결합 금성, (회색) V1 및 V2 (오렌지) 조각 (PDB 취득 3OGO)의 위치를 보여주는 사이 상호 작용 인터페이스 (사이안)의 (B) 구조 분석. 이 그림은 재 fromCroucher 외3 Reprinted AAAS 허가입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
기법 BiCAP 선호도 낮은 수준의 상호 작용 그들의 퓨전 파트너 사이 발생 하지 않는 한 연관 금성 (v 1과 v 2 라는)의 2 개의 비 형광 파편의 사용 합니다. 이 경우 두 개의 분할 도메인 refold fluorophore (그림 1B)6의 기능 β 배럴 구조. BiCAP의 주요 혁신 재조합 GFP nanobody, β-총 신 GFP (금성 같은 변종)의 재결합 올바르고 접힌 fluorophore ( 에만에 3 차원 epitope를 인식의 소개에서 온다 그림 1B)8. 결정적인 GFP nanobody 개별 비너스 조각 중 하나에 바인딩할 하지 않습니다. 이 두 가지 단백질의 그들의 자신의 지, 그 취득 하는 방법에서 화학적으로 유도 된, 강제 상호 작용9의 사용 보다 더 대표적인 결과 복잡 한 형성 후에 단백질 이합체의 분리를 촉진 한다.
BiCAP 다운스트림 응용 프로그램 역할 이러한 단지 신호 변환에서의 우리의 이해의 세분성을 향상 시킬 수와 잠재적으로 결합 될 수 있는 다중 단백질 복합물에 특히 초점을 맞추고 있는 강력한 기술입니다. . 그것은 또한 단백질 상호 작용 제자리의 시각화를 허용의 중요 한 기능을 포함. 날짜 하려면, BiCAP 수용 체 티로신 키 니 아 제 (RTK) 이합체3위하여 분석의 효과적인 방법으로 입증 되었습니다 하지만이 방법의 적응성 거의 모든 단백질 상호 작용의 맥락에서 채택 될 수 있다.
Protocol
Representative Results
Discussion
BiCAP 개별 구성 요소와 그들의 경쟁 바인딩 파트너3을 제외 하는 동안 특정 단백질 이합체를 격리 하기 위한 강력한 방법입니다. BiCAP은 BiFC6라는 형광 단백질 보완성 분석 결과의 적응을 기반으로 합니다. BiFC와 근접 결 찰 분석을 포함 하 여 기존 방법 시각화 하 고 라이브 셀7상호 작용 단백질 정량에 광범위 하 게 사용 되었습니다 하지만 격리…
Divulgations
The authors have nothing to disclose.
Acknowledgements
D.R.C 암 연구소 NSW 동료 이며 D.N.S 이전 암 연구소 NSW 동료 이었다. 이 원고에 제시 하는 연구 결과 암 연구소 NSW (13/FRL/1-02과 09/CDF/2-39), NHMRC (프로젝트 그랜트 GNT1052963), 과학 재단 아일랜드 (11/SIRG/B2157), 뉴 사우스 웨일즈 과학 및 의료 연구, 이용 가족에 의해 투자 되었다 친목 그리고 모스 틴 가족 재단입니다. J.F.H.와 텍 오스트레일리아 대학원 포상의 수령인 이었다.
Materials
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid – Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) – 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).

