Dissekera multi signalering proteinkomplex av Bimolecular komplettering affinitet rening (BiCAP)
Summary
Detta manuskript beskriver protokollet för Bimolecular komplettering affinitet rening (BiCAP). Denna nya metod underlättar specifika isolering och nedströms proteomiska karakterisering av några två samverkande proteiner, medan exklusive FN-komplex enskilda proteiner samt komplex bildas med konkurrerande bindande partner.
Abstract
Montering av proteinkomplex är en central mekanism som ligger bakom regleringen av många cell signalering vägar. Ett stort fokus på biomedicinsk forskning dechiffrera hur dessa dynamiska proteinkomplex agera för att integrera signaler från flera källor för att rikta en specifik biologisk reaktion, och hur detta blir avregleras i många sjukdomar inställningar. Trots betydelsen av denna viktiga biokemiska mekanism finns det en brist på experimentella metoder som kan underlätta den specifika och känsliga deconvolution av dessa multi molekylär signalering komplex.
Här åtgärdas denna brist genom kombinationen av en protein komplettering analysen med en konformation-specifika nanobody, som vi har kallat Bimolecular komplettering affinitet rening (BiCAP). Denna nya teknik underlättar specifika isolering och nedströms proteomiska karakterisering av alla par av samverkande proteiner, med uteslutande av FN-komplex enskilda proteiner och komplex som bildas med konkurrerande bindande partner.
Den BiCAP tekniken är anpassningsbar till ett brett utbud av nedströms experimentella analyser, och den höga graden av specificitet som ges genom denna teknik tillåter mer nyanserad utredningar av mekaniken i protein komplex sammansättning än för närvarande använder standard affinitet rening tekniker.
Introduction
Protein komplex sammansättning är en viktig process för att upprätthålla många signalering vägar1,2spatiotemporal specificitet. Medan kritiska arten av denna reglerande roll är allmänt erkänd, finns det en brist på experimentella tekniker tillgängliga att granska dessa komplex. De flesta interactomics studier fokuserar på interaktioner med enskilda proteiner eller sekventiella anrikningen av enskilda komplexa komponenter. Här presenterar vi en teknik för isolering av ett specifikt protein dimer medan exklusive de enskilda delarna av den komponenten proteiner samt komplex bildade med konkurrerande bindande partner3. Vi har kallat denna teknik Bimolecular komplettering affinitet rening (BiCAP), eftersom det är en kombination av en tidigare existerande protein fragment komplettering analys, Bimolecular fluorescens komplettering (BiFC), med ny användning av en exteriör-specifika rekombinant nanobody mot GFP och dess derivat (se tabellen för material).
En typisk protein-fragment komplettering analysen bygger på uttrycket av ”bete” och ”offer” proteiner smält för att dela upp fragment av reportrar som luciferas4, β-galaktosidas5eller grönt fluorescerande protein (GFP)6 ( Figur 1A). Genom samverkan mellan bete och byte proteinerna uppmuntras split reporter domäner refold till en funktionell struktur, vilket gör att samspelet mellan betet och bytesdjur proteiner som ska visualiseras eller kvantifieras. BiCAP var anpassad från en version av denna teknik som gjord använda av fragment av GFP varianten Venus. Fluorescerande protein komplettering analyser är en populär metod för att visualisera protein-protein interaktioner i en levande cell, men hittills har begränsats till detta en funktion7. BiCAP utgör ett betydande framsteg i detta avseende, eftersom denna teknik inte bara möjliggör visualisering, men också isolering och förhör av den resulterande protein-protein interaktionen.
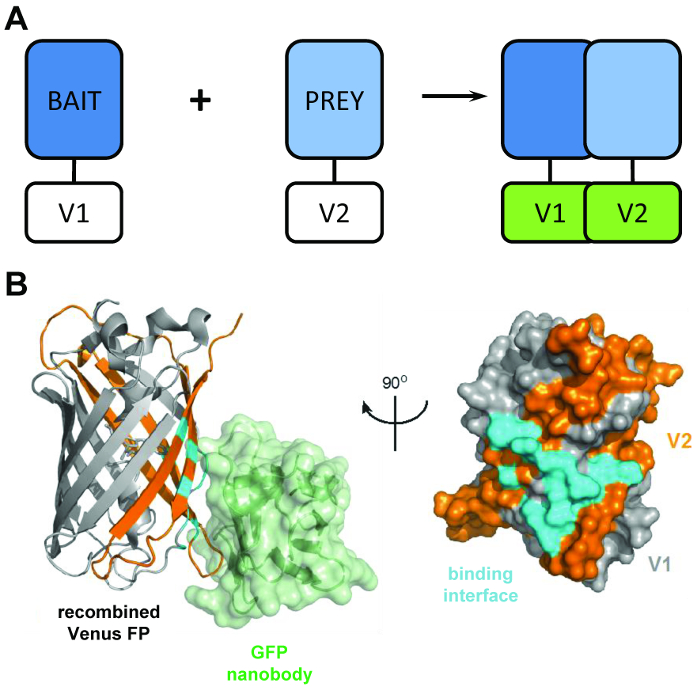
Figur 1: den strukturella viktigaste bakom tekniken som BiCAP. (A) en schematisk disposition huvudmannen bakom bimolecular fluorescens komplettering visar ‘bete’ och ‘offer’ proteinerna taggade med N-terminala V1 eller C-terminal V2 fragment av proteinet fullängds Venus. (B) strukturell analys av interaktion gränssnittet (cyan) mellan GFP nanobody (grön) och rekombinerat Venus, visar positionen för V1 (grå) och V2 (orange) fragment (PDB anslutning 3OGO). Denna siffra är ompublicerade fromCroucher et al.3 Reprinted med tillstånd från AAAS. Klicka här för att se en större version av denna siffra.
Den BiCAP teknik gör användningen av två icke-fluorescerande fragment av Venus (heter V1 och V2) som associeras med en låg grad av samhörighet såvida inte en interaktion sker mellan deras fusion partners. I detta fall refold de två delade domänerna i funktionella β-fat strukturen av fluorophore (figur 1B)6. Den viktigaste nyheten i BiCAP kommer från införandet av den rekombinanta GFP-nanobody, som erkänner en tredimensionell epitop på β-fat av god Jordbrukarsed (och varianter såsom Venus) som finns endast på korrekt rekombinerat och vikta fluorophore ( Figur 1B)8. Avgörande, binder den GFP nanobody inte till antingen enskilda Venus fragment. Detta underlättar isolering av protein dimerer först efter de två proteinerna har bildat ett komplex av egen vilja, vilket leder till mer representativt resultat än sådana som förvärvats från metoder som gör användningen av kemiskt inducerad, Tvingad interaktioner9.
BiCAP är en kraftfull teknik som specifikt fokuserar på flera proteinkomplex, som potentiellt kan kombineras med ett antal efterföljande program för att förbättra precisionen av vår förståelse av dessa komplex roll i Signaltransduktion . Det omfattar också den viktiga funktionen att låta visualisering av protein interaktioner i situ. Hittills har BiCAP har visats som en effektiv metod för att analysera interactome receptor tyrosine kinase (RTK) dimerer3, men anpassningsförmåga i denna metod innebär att den kan antas i nästan alla sammanhang med protein i interaktion.
Protocol
Representative Results
Discussion
BiCAP är en kraftfull metod för att isolera specifika protein dimerer medan exklusive de enskilda komponenterna och deras konkurrerande bindande partner3. BiCAP bygger på anpassning av en fluorescens protein komplettering analysen kallas BiFC6. Befintliga metoder, inklusive BiFC och närhet ligering analyser, har använts i stor utsträckning att visualisera och kvantifiera proteininteraktioner i levande celler7, men lämnade inte ett effektivt s?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
D.R.C är en Cancer Institute NSW Fellow och D.N.S var tidigare en Cancer Institute NSW Karl. Forskningsresultaten presenteras i detta manuskript finansierades av Cancer Institute NSW (13/FRL/1-02 och 09/CDF/2-39), NHMRC (Project Grant GNT1052963), Science Foundation Ireland (11/SIRG/B2157), NSW Office of Science och medicinsk forskning, värdfamilj Gemenskap och Mostyn Family Foundation. J.F.H. och R.S. var mottagare av en australiensisk forskarutbildning Award.
Materials
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid – Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) – 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).

