Bimolecular tamamlayıcı benzeşme arıtma (BiCAP) tarafından parçalanmış çok protein sinyal kompleksleri
Summary
Bu el yazması Bimolecular uluslara benzeşme arıtma (BiCAP) protokolünü açıklar. BM complexed bireysel proteinler hem de konut projeleri hariç rakip bağlama ortaklarıyla oluşan iken bu roman yöntemi belirli yalıtım ve aşağı akım proteomik karakterizasyonu herhangi bir iki etkileşen proteinlerin kolaylaştırır.
Abstract
Protein kompleksleri Meclisi birçok hücre yolları sinyal düzenlenmesi temel merkezi bir mekanizmadır. Biyomedikal araştırma önemli bir odak noktası bu dinamik protein kompleksleri sinyalleri birden fazla kaynaktan doğrudan belirli bir biyolojik tepki için entegre hareket nasıl ve ne kadar bu birçok hastalığın ayarlarında kuralsız olur deşifre. Bu anahtar biyokimyasal mekanizması önem rağmen bu çok moleküler sinyal kompleksleri Özel ve hassas deconvolution kolaylaştırabilir Deneysel teknikler eksikliği vardır.
Burada bu eksiklik bir protein uluslara tahlil Bimolecular uluslara benzeşme arıtma (BiCAP) olarak adlandırdığı bir uyum özel nanobody ile birleşimi yoluyla ele alınmaktadır. Bu roman tekniği etkileşen proteinler, BM complexed bireysel proteinler ve rakip bağlama ortakları ile oluşturduğu kompleksler hariç herhangi bir çiftin malzemelerin belirli yalıtım ve aşağı akım proteomik kolaylaştırır.
BiCAP teknik aşağı akım deneysel deneyleri geniş bir dizi için uyarlanabilir ve bu tekniği ile tanınan özgüllüğü yüksek derecede Şu anda mümkün olduğundan daha farklı yaklaşımlar içeren soruşturmalar protein kompleksi derleme mekaniği sağlar kullanarak Standart benzeşme arıtma teknikleri.
Introduction
Protein kompleksi derleme birçok sinyal verme yolları1,2kronolojik zamanmekansal özgüllük korumada önemli bir süreçtir. Bu düzenleyici rol kritik doğası tanınmış ise Deneysel teknikler bu komplekslerin ince eleyip sık dokumak kullanılabilir bir eksikliği. En interactomics çalışmaları bireysel proteinler veya karmaşık bileşenleri tek tek sıralı zenginleştirme ile etkileşimleri üzerine odaklanır. Burada bireysel moieties bileşen proteinler hem de konut projeleri hariç ortakları3bağlama rekabet ile oluşan iken bir teknik belirli protein dimer yalıtım için mevcut. Biz bu teknik çağrıda bulundular Bimolecular uluslara benzeşme arıtma (BiCAP), olduğu gibi bir arada bir önceden varolan protein parçası uluslara testin, Bimolecular floresan uluslara (BiFC), roman kullanımı ile bir GFP ve türevleri doğru biçimi özgü rekombinant nanobody (bkz: malzemeler tablo).
Luciferase4, β-galaktozidaz5veya yeşil flüoresan protein (GFP)6 ( gibi gazetecilere parçaları bölmek erimiş “yem” ve “av” proteinlerin ifade tipik protein-fragment uluslara tahlil dayanan Şekil 1A). Yem ve av protein etkileşimi, split muhabir etki alanları yem etkileşim sağlayan bir işlev yapısı içinde refold ve proteinler görüntülenir veya sayısal için av için teşvik edilir. BiCAP yapılan bu teknik bir sürümünden adapte GFP değişken Venüs parçaları kullanın. Floresan protein uluslara deneyleri canlı hücrede protein-protein etkileşimleri görselleştirmek için popüler bir yöntemdir, ancak şimdiye kadar bu bir işlev7‘ ye sınırlı olmuştur. Bu teknik sadece görselleştirme, ama aynı zamanda yalıtım ve sorgulama elde edilen protein-protein etkileşim sağlar gibi BiCAP bu bağlamda, önemli bir ilerleme temsil eder.
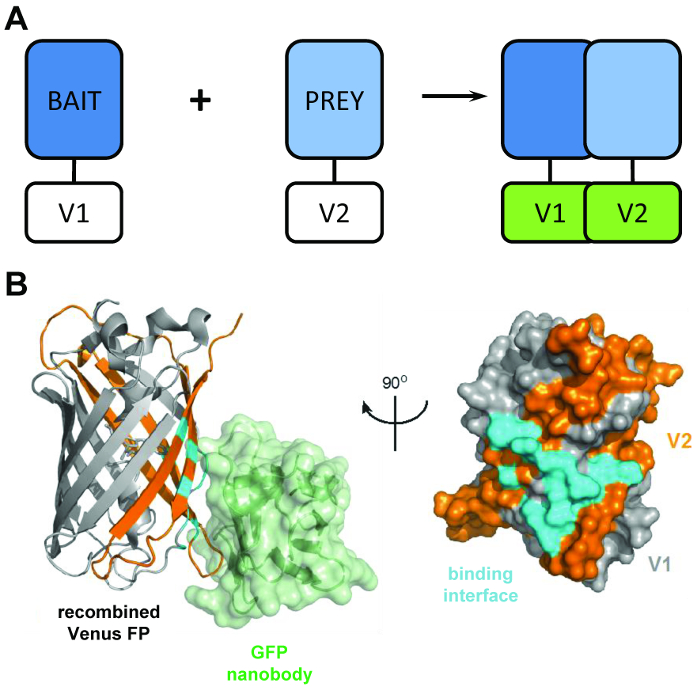
Şekil 1: BiCAP teknik arkasında yapısal müdür. (A) bimolecular floresan uluslara gösteren arkasında asıl özetleyen bir şematik ‘yem’ ve ‘av’ proteinler N-terminal V1 veya C-terminal V2 ile tam uzunlukta Venüs protein parçaları öğesini. (B) yapısal analizi GFP nanobody (yeşil) ve (gri) V1 ve V2 (turuncu) parçaları (PDB katılım 3OGO) konumunu gösteren recombined Venüs arasındaki etkileşim arayüzü (Camgöbeği). Bu rakam yayımlanamaz fromCroucher vd3 Reprinted AAAS izniyle olduğunu. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Füzyon eşleri arasında bir etkileşim oluşur sürece benzeşme düşük bir derece ile ilişkilendirmek iki floresan sigara parçaları (V1 ve V2 olarak adlandırılır) Venüs’ün Kullanım tekniği yapar BiCAP. Bu durumda, iki bölünmüş etki alanı fluorophore (Şekil 1B)6fonksiyonel β-varil yapısına refold. BiCAP anahtar yenilik β-sadece düzgün recombined ve katlanmış fluorophore ( üzerinde mevcut olan varil üzerinde GFP (ve Venüs gibi varyantların) üç boyutlu epitope tanır rekombinant GFP nanobody getirilmesi gelir Şekil 1B)8. En önemlisi, GFP nanobody ya bireysel Venüs parçaları bağlanmaz. Sadece iki protein o yapmak metotlarından alınan kimyasal kaynaklı, zorla etkileşim9/ kullanma daha fazla temsilcisi sonuç için önde gelen kendi iradesiyle bir kompleks oluşmuş sonra bu protein dimer yalıtım kolaylaştırır.
BiCAP özellikle potansiyel taneciklik’Bu komplekslerin sinyal iletimi içinde oynadıkları rolü anlayışımızı geliştirmek için aşağı akım uygulamaları bir dizi ile birlikte çok protein kompleksleri üzerinde duruluyor güçlü bir tekniktir . Ayrıca protein etkileşimler içinde in situgörselleştirme sağlayan önemli bir özelliği kapsar. Bugüne kadar BiCAP reseptör Tirozin kinaz (RTK) dimer3interactome analiz etkili bir yöntem gösterilmiştir, ama bu yöntemin uyum yeteneği hemen hemen her protein etkileşimi bağlamında kabul edilmesi anlamına gelir.
Protocol
Representative Results
Discussion
BiCAP ise tek tek bileşenleri ve onların rakip bağlama ortaklar3hariç belirli protein dimer izole için güçlü bir yöntemdir. BiCAP BiFC6adı verilen bir floresan protein uluslara tahlil adaptasyonu üzerinde temel alır. Tüp ligasyonu deneyleri, BiFC ve yakınlık da dahil olmak üzere mevcut yöntemler görselleştirmek ve protein etkileşimleri canlı hücreleri7ölçmek için kapsamlı bir şekilde kullanılmış ama yalıtma ve oluşmuş…
Divulgations
The authors have nothing to disclose.
Acknowledgements
D.R.C Kanser Enstitüsü NSW adam ve D.N.S daha önce Kanser Enstitüsü NSW bursu aldı. Bu el yazması sunulan araştırma bulguları Kanser Enstitüsü NSW (13/FRL/1-02 ve 09/CDF/2-39), NHMRC (proje Grant GNT1052963), Bilim Vakfı İrlanda (11/SIRG/B2157), NSW Office Bilim ve tıbbi araştırma, Konuk aile tarafından finanse edilmiştir Arkadaş grubu ve Mostyn Aile Vakfı. J.F.H. ve R.S. alıcılar bir Avustralya yüksek lisans Ödülü…
Materials
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid – Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) – 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).

