수집 및 RNA 분석에 대 한 큰 동물에서 림프절의 처리: 큰 동물 종의 림프 노드 Transcriptomic 연구에 대 한 준비
Summary
이 프로토콜은 림프절 조직 식별 하 고 가축과 야생 동물, 샘플링 방법에서 림프절의 절단 단계를 포함 하 여 큰 동물에서의 프로 파일링 transcriptomic RNA의 격리에 대 한 절차의 개요를 제공 사후 수집 보존 및 RNA 분석 처리에 대 한 여러 동물, 그리고 고려 사항 플러스 대표 결과 걸쳐 일관성을 제공 합니다.
Abstract
큰 동물 (가축과 야생 동물) 동물 매개 병원 체, 세라, 진 균 bovis, 살 모 넬 라, 대장균, 등의 중요 한 공기 통으로 봉사 하 고 pathogenesis의 연구에 대 한 유용 및 자연 호스트에서 박테리아의 확산입니다. 호스트 면역 반응에서 림프절의 핵심 기능으로, 림프절 조직 역할을 다운스트림 transcriptomic 분석에 대 한 RNA의 감염의 과정을 통해 세포에 유전자 발현의 시간적 변화를 평가 합니다. 이 문서는 림프 노드 컬렉션, 조직 샘플링 및 다운스트림 RNA 아메리카 들소 (비 손 비 손에서에서 제공 하는 추가 예제와 함께 모델로, 가축 (보스 토 러 스)를 사용 하 여 가축, 처리 과정의 개요를 선물 한다 ). 프로토콜 시체의 위치, 식별 및 여러 주요 사이트에서 림프절의 제거에 대 한 정보를 포함합니다. 또한, 생 검 샘플링 방법론 여러 동물에서 샘플링의 일관성에 대 한 수 있도록 제공 됩니다. 샘플 보존에 대 한 몇 가지 고려 사항, RNA 시퀀싱 및 RT-PCR 같은 다운스트림 방법론에 대 한 적합 한 RNA의 생성을 포함 하 여 설명 합니다. 큰 동물 대 마우스 시간 과정 연구에 내재 된 긴 지연으로 인해 들소와 소 림프절 조직에서 대표적인 결과의 검토의 맥락에서이 조직 유형에 저하의 시간 과정을 설명 하기 위해 제공 됩니다. 다른 조직에서 RNA 저하에 이전 방법론 작동 합니다. 전반적으로,이 프로토콜은 유용 두 수의 연구에 큰 동물 샘플 transcriptome 프로젝트를 시작 하 고 분자 생물학 조직 샘플링 vivo에서 그리고 생체 외에서 처리 기법을 학습에 관심이 있을 것입니다.
Introduction
림프절의 transcriptome의 RNA 시퀀싱 분석 병원 체의 다양 한 동물의 면역 반응을 하 기회를 제공 한다. 이 방법론은 생쥐에서 광범위 하 게 이용 되어, 하는 동안 분석은 최근 큰 포유류1,2로 확대 되었습니다. 가축 또는 큰 동물 림프 노드 호스트 감염 뿐만 아니라 백신 또는 유전 자화 율 연구에 그들의 사용을 위해 그리고 인간 연구에 대 한 모델 시스템으로 서 뿐만 아니라 신약 개발에 대 한 대상의 식별에 대 한 응답 특성을 사용할 수 있습니다. 동물 매개 질병에. 예를 들어 brucellosis (동물 매개 세균성 질병 그 영향은 절반 백만 사람들이 전 세계 매년), 경우에 크게 증가 비용, 연구 양에서 또는 염소는 더 인간 감염 및 인간 백신 관련 실험실 동물 모델 보다 개발입니다. 마우스 감염 모델 정리 reticuloendothelial 시스템 감염 하지만 하지는 특징적인 임상 증상3.
실험실 동물 연구에 비해 큰 동물 실험에서 조직의 반드시 수확 과정 포함는 안락사의 보존에 대 한 잠재적인 도전을 선물 한다 조직 컬렉션 사이 긴 지연 높은-품질 한 RNA 그대로 RNA 생물학 관련 transcriptomic 데이터의 생성을 위해 필수적 이다. 조직 샘플에서 높은-품질 RNA의 생성은 특히 큰 동물 병원 체 연구에 대 한 중요 한 실시 제약 시설에서. 이러한 연구는 본질적으로 더 그들은 뿐만 아니라 시설 승인 및 고도로 훈련 직원 필요 하지만 상당한 금융 비용을 작업에 따라 수백 수천 달러의 수만에서 배열할 수 있다 수행를 수행 하기 어렵다. 이러한 유형의 연구는 또한 크로스-징계 협업 및 그들의 복잡성을 추가 하는 그들의 완성에 대 한 크로스-징계 지식을 포함 한다. 따라서,에 대 한 교육, 개발, 샘플 수집 및 보존에 대 한 효율적인된 시스템을 준수 하는 이득을 제공 한다 중요 한 조직의 다운스트림 분자 연구에 대 한 감염 된 동물에서.
큰 림프 노드의 컬렉션 murine 림프절의 유사한 샘플링에 비해 조직 컬렉션에 대 한 추가적인 도전을 선물 한다. 샘플 절단에 대 한 준비 관련 내부 구조를 포함 한 림프절의 해부학에 대 한 기본적인 이해를 필요로 합니다. 림프 노드의 구조는 림프 lobules sinuses 림프로 가득 둘러싸인 구성 됩니다. 이러한 구조는 거친, 섬유질 캡슐 포함. 4 림프 lobule “기본 해 부와 기능적인 단위 림프 노드의” 이며 낭, 깊은 외피 단위 및 골 수 코드와 sinuses4 (그림 1A)로 구성 됩니다. B와 T 세포는 각각 낭과 깊은 외피 단위에 가정. 이러한 구조 3D 비 계를 제공 하 고 있는 림프 톨 항 원 또는 항 원 제시 세포 사이 상호 작용 촉진.
조잡 한, 낭과 깊은 대뇌 피 질의 단위 확인할 수 있습니다 잘라 표면에 그들은 빽으로 채널을 포함 하 고 보다 더 섬세 한 망상 채널의 구성 되어 나타나고 라이터는 sinuses 표시 (그림 1B). 규칙에 따라 병리학 표면 피 질 (낭), paracortex (깊은 외피 단위)와 골 수 (골 수 코드와 sinuses) 림프절의 영역을 참조 하십시오. 모든 3 개의 영역의 적절 한 시험 간주 되었습니다 최고로 림프절5에 대 한 일상적인 병 적인 시험 지침에 연습. 참고 일관성, 크기, 및 단일 동물 내 에서도 림프절의 색상에 상당한 변이가 있다. 동물 나이, 그들의 림프절 크기 감소와 젊은 동물의 그들 보다는 더 확고 한 되, 일반적으로 그들의 결합 조직 및 림프 정상의 감소에서 증가로 인해6,7을 구조 하는 경향이 됩니다.
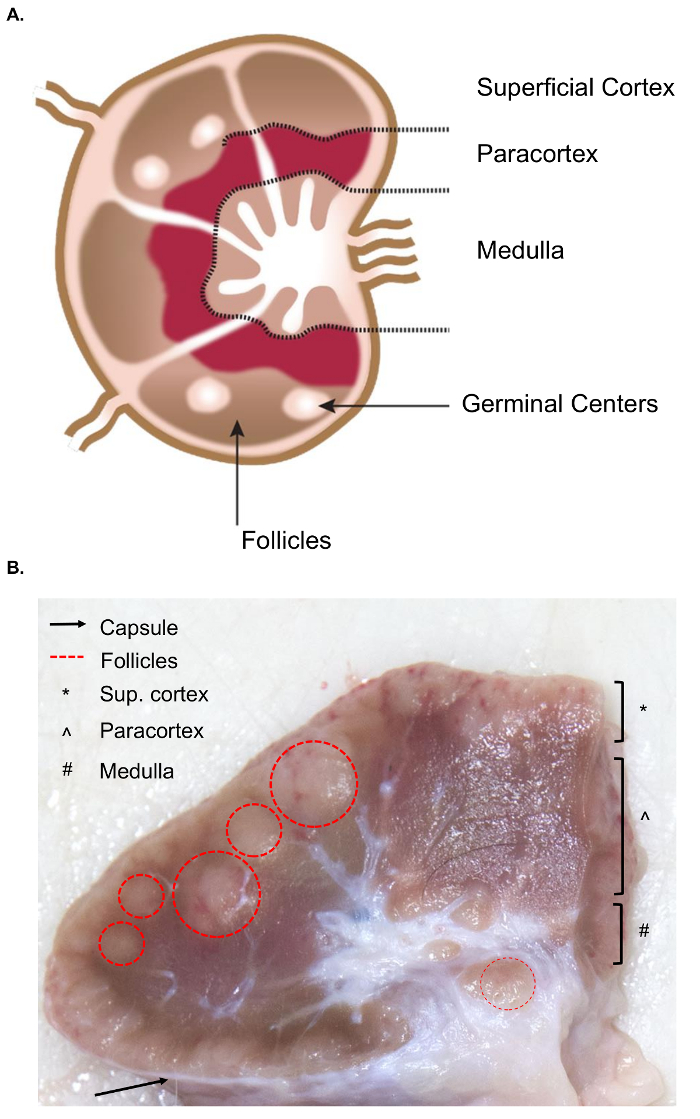
그림 1. 림프절의 해부학. (A)이 만화 이미지 표시, 림프절의 해부학 키 구조를 묘사. (B)이 여전히이 이미지 횡단면으로 잘라 소 림프 노드를 보여 줍니다. 육안으로 볼 수 있습니다 관련 구조/레이어 강조 표시 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
실험적인 질문에 따라 다른 림프절 수집 및 분석에 대 한 관심이 될 것입니다. 주변 림프절은 피하 조직에 깊이 있는. 가축, 주변 또는 표면 림프절 임상 및 실험 연습에서 자주 사용 한다 포함 귀 밑, submandibular, retropharyngeal, prescapular, prefemoral (precrural)와 표면 사 타 구니 (supramammary 여성, 남성에서 음 낭에서) ( 그림 2)입니다. 표 1주요 표면 림프절의 속성 가축 시스템8에 설명 된 대로 설명 되어 있습니다. 아래 소의 세균성 전염병에 대 한 몇 가지 잠재적인 림프 노드 컬렉션 계획 조사에 대 한 시작 지점으로 제공 됩니다.
세라 abortus/세라 melitensis: 표준을 B. abortus위한 necropsies-가축 및 B. melitensis감염-감염 된 염소 국립 동물 질병 센터에서 supramammary, prescapular, 및 선 림프절 조직 복구 모두 호스트 RNA 식 프로 파일링에 대 한 RNA 준비를 위한 세균성 열거형에 대 한 분쇄. B. abortus 정기적으로 실험적으로 감염 된 가축9. 에 이러한 림프절의 각 재기 될 수 있다 B. melitensis에 이러한 림프 노드 형식 각각에 있는 박테리아의 존재를 검출 될 수 있다-우리의 연구 (Boggiatto 그 외 여러분, 미 출판)에서 RNA 기반 방법론을 사용 하 여 감염 된 후 적어도 9 개월까지 염소를 감염. 살 모 넬 라 sp.: prescapular, subiliac (prefemoral), 그리고 mesenteric 림프절 살 모 넬 라 보급10,,1112 에 대 한 가축 시체를 프로 파일링 하는 동안 유용 하 게 되었습니다, transcriptomic 연구에 대 한 잠재적인 관심의 것입니다. E. 콜라이 O157:H7: (중간 소장 원심 소장 위치에서) Mesenteric 림프절 박테리아 감염 된 송아지 (에 감염 된 성인 가축)13의 가끔 회복의 사이트 수 있습니다. 렙 (Leptospira sp.): 박테리아의 만성 지 속성 유선14배출 림프절에서 관찰 되었습니다. 진 균 bovis : 가축, 박테리아 종 아리15의 mediastinal 및 tracheobronchial 림프절에서 복구 된 후 실험 감염 되었습니다. 또한, 림프절 RNA 돼지 재생산과 호흡 증후군 바이러스2등 큰 동물 호스트 응답을 검사 하 이용 되어 있다. 그림 2 는 가축 시체에서 이러한 주요 림프절의 하위 집합의 위치를 보여 줍니다.
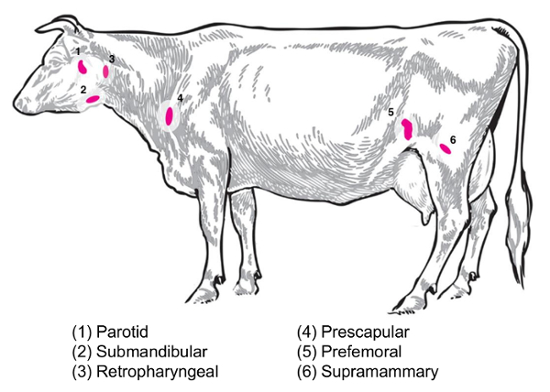
그림 2: 선택 된 림프 노드 위치를 묘사 하는 만화 보스 토 러 스 . 번호가 매겨진된 림프절은 주석 처리 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
이 문서와 관련 된 비디오, 선물이 큰 동물 감염의 transcriptomic 연구에 관련 된 분자 생물학에 대 한 유익 하도록 설계 된 RNA 연구에 대 한 큰 동물 림프절의 격리에 대 한 프로토콜. 먼저, 우리 제공 격리 절차의 개요, 림프절에 대 한 예제로 소과 들소 조직에서 샘플링을 사용 하 여. 와 결합 하 여이 데모 비디오에 표시 된 대로, RNA 격리에 대 한 재현 조직 샘플링에 대 한 워크플로우가 이다. 다음, 우리는 감염 된 림프절, 안전, 일관성, 그리고 RNA 품질에 초점을 맞춘의 처리에 대 한 중요 한 고려 사항을 설명합니다.
산성화 페 놀 guanidine isothiocyanate 시와 조직에서 RNA의 준비 Chomczynski 및 Sacchi16,17, 실리 카 기반 스핀 열의 존재에 대해 정화의 원래 방법에 따라 Vogelstein 길18의 원래의 작품에 따라 chaotropic 대리인. 우리는 또한 transcriptomics에 대 한 대체 방법으로 보존 하는 가축 림프절에서 RNA의 복구에 대 한 가능성을 검사 합니다. 마지막으로, 우리는 안락사와 들소에서 복구 된 RNA 프로 파일에 샘플링 사이의 시간 증가의 효과 묘사 하는 대표적인 실험을 포함 하 여 큰 동물 necropsies에서 RNA 품질에 시간 변수의 영향을 탐구 하 고 소 림프 노드입니다. 이 문서 뿐만 아니라 분자 생물학 하지만 transcriptomic 연구를 개시 하는 또한 하 수의 연구자에 유용할 것 이다.
Protocol
Representative Results
Discussion
마우스, 쥐, 또는 사후 인간의 샘플 transcriptomic 연구와 관련 된 프로토콜의 대부분에 의하여 초점 있다. 그러나, 가축과 야생 동물에 대 한 조사는 인간의 보건에 모두에 적용 가능한 동물 용 의약품, 동물 매개 질병에 관한 질병에 면역 반응의 특성에 대 한 기회의 넓은 범위를 제공합니다. 이 프로토콜은 큰 동물, 가축, 들소, 염소, 양 등에서 높은 무결성 조직에서 RNA 추출에 대 한 주요 고려 사항?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
저자는 제임스 포 세 모든 videography 및 비디오 처리; 그의 뛰어난 작품에 대 한 감사 하 고 싶습니다. 디지털된 가축 이미지;의 세대에 그의 뛰어난 작품에 대 한 마이클 마티 릴리 아는 발터에 대 한 그녀의 RNA 추출 및 Bioanalyzer 실행; 미치 파머와 도움이 검토 및 림프 노드 이미지;에 대 한 피드백에 대 한 칼 리 Kanipe 동물 보호 그리고 그들의 노력 및 축산 지원의 모든 국립 동물 질병 센터와 차례의 준비에서 수의 직원 들.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
References
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

