Coleta e processamento de linfonodos de animais de grandes porte para a análise do RNA: preparando-se para estudos de linfonodo Transcriptomic de espécies de animais grandes
Summary
Este protocolo fornece uma visão geral dos procedimentos para o isolamento do RNA para o perfil de transcriptomic do nó de linfa de tecidos de animais de grandes porte, incluindo etapas na identificação e excisão dos gânglios linfáticos do gado e animais selvagens, abordagens de amostragem para fornecer consistência em vários animais e considerações mais resultados representativos para a preservação de pós coleta e processamento para a análise do RNA.
Abstract
Animais de grandes porte (gado e animais selvagens) servem como importantes reservatórios de agentes patogénicos zoonóticos, incluindo Brucella, Mycobacterium bovis, Salmonellae Escherichia colie são úteis para o estudo da patogênese e/ou propagação das bactérias em hospedeiros naturais. Com a função chave de linfonodos na resposta imune do hospedeiro, linfonodo tecidos servem como uma fonte potencial de RNA para análises de transcriptomic a jusante, a fim de avaliar as mudanças temporais na expressão gênica em células no decorrer de uma infecção. Este artigo apresenta uma visão geral do processo de coleção de nó de linfa, amostragem de tecido e a jusante do RNA processamento em gado, usando bovinos (Bos taurus) como um modelo, com exemplos adicionais provenientes do Bisão-americano (bison Bison ). O protocolo inclui informações sobre a localização, identificação e remoção dos gânglios linfáticos de diversos sites chaves no corpo. Além disso, uma metodologia de amostragem de biópsia é apresentada que permite a uma consistência de amostragem em vários animais. Várias considerações para preservação de amostra são discutidas, incluindo a geração de RNA apropriado para jusante metodologias como a sequenciação do ARN e RT-PCR. Devido os longos atrasos inerentes a estudos de curso de tempo grandes animais vs rato, apresentam-se resultados representativos de bison e tecidos de bovinos do nó de linfa para descrever o tempo de degradação neste tipo de tecido, no contexto de uma revisão da trabalho metodológico anterior na degradação de RNA em outros tecidos. Em geral, este protocolo será útil para ambos os pesquisadores veterinários começando transcriptome projetos em grandes amostras de animais e biólogos moleculares interessados em aprender técnicas para processamento de em vitro e em vivo amostragem de tecido.
Introduction
A sequenciação do ARN da transcriptoma de linfonodos fornece a oportunidade para caracterizar a resposta imune dos animais para uma variedade de agentes patogénicos. Enquanto esta metodologia tem sido utilizada extensivamente em camundongos, análises tem recentemente expandido para maiores mamíferos1,2. Pecuária/grandes animais linfonodos podem ser usados para caracterizar respostas do hospedeiro a uma infecção, não só para a sua utilização em estudos de susceptibilidade genética ou vacina e para a identificação de alvos para o desenvolvimento de drogas, mas também como sistemas modelo para estudos em humanos em doenças zoonóticas. Por exemplo, no caso da brucelose (uma doença bacteriana zoonótica que impactos meio milhão de pessoas ao redor do mundo cada ano), apesar do aumento significativamente os custos, estudos em ovinos ou caprinos são mais relevantes para a infecção humana e vacina humana desenvolvimento de modelos animais de laboratório. Modelos de infecção mouse recapitular a infecção do sistema reticuloendotelial, mas não os sinais clínicos característicos3.
Em experimentos com animais grandes em comparação com estudos de animais de laboratório, o processo de tecido colheita necessariamente envolve um atraso maior entre a eutanásia e a coleção de tecido, que apresenta um potencial desafio para a preservação da RNA de alta qualidade. O RNA intacto é essencial para a geração de dados transcriptomic biologicamente relevantes. A geração do RNA de alta qualidade das amostras de tecido é particularmente crítico para estudos de grande patógeno animal realizado em instalações de contenção. Tais estudos são inerentemente mais difíceis de executar, como eles não só exigem aprovado instalações e pessoal altamente treinado, mas também carregam custos financeiros significativos, que, dependendo do trabalho, que podem variar de dezenas a centenas de milhares de dólares. Estes tipos de estudos envolvem também uma colaboração interdisciplinar e o conhecimento interdisciplinar para a sua conclusão, adicionando à sua complexidade. Portanto, formação sobre, desenvolvimento e adesão a um sistema simplificado para a coleta de amostra e preservação fornece benefícios significativos para a jusante estudos moleculares de tecidos de animais infectados.
A coleção de linfonodos maiores apresenta desafios adicionais para a coleta de tecido, em comparação com a amostragem semelhante de murino linfonodos. A preparação para a excisão de amostra exige um conhecimento básico da anatomia do nó de linfa, incluindo as estruturas internas relevantes. A estrutura de um nó de linfa é composta por lóbulos linfoides rodeados por seios cheios de linfa. Estas estruturas são colocadas dentro de uma cápsula fibrosa, resistente. 4 um lóbulo linfoide é “anatômica e funcional unidade básica do nó de linfa” e é composto de folículos, uma unidade profunda cortical e cordões medulares e seios4 (figura 1A). Linfócitos B e T são home aos folículos e profundo corticais unidades, respectivamente. Essas estruturas fornecem um andaime 3D e facilitam a interação entre os linfócitos e antígeno ou células apresentadoras.
Grosseiramente, folículos e unidades profundo corticais podem ser identificadas na superfície de corte como eles contêm uma malha reticular mais densa e aparecem mais escuros do que os seios, que são compreendidos por uma malha reticular mais delicada e aparecem mais leves (figura 1B). Por convenção, os patologistas referem-se às regiões dos gânglios linfáticos como o córtex superficial (folículos), o paracórtex (fundo corticais unidades) e a medula (cordões medulares e seios). Um exame adequado das três regiões tem sido considerado como a melhor prática em diretrizes de exame patológico de rotina para linfonodos5. Observe que há uma variação considerável a consistência, tamanho e cor dos gânglios linfáticos, mesmo dentro de um único animal. Como idade de animais, os gânglios linfáticos tendem a diminuir de tamanho e tornam-se mais firme do que aqueles de animais mais jovens, normalmente devido a um aumento em seu tecido conjuntivo e uma redução do linfoide normal estrutura6,7.
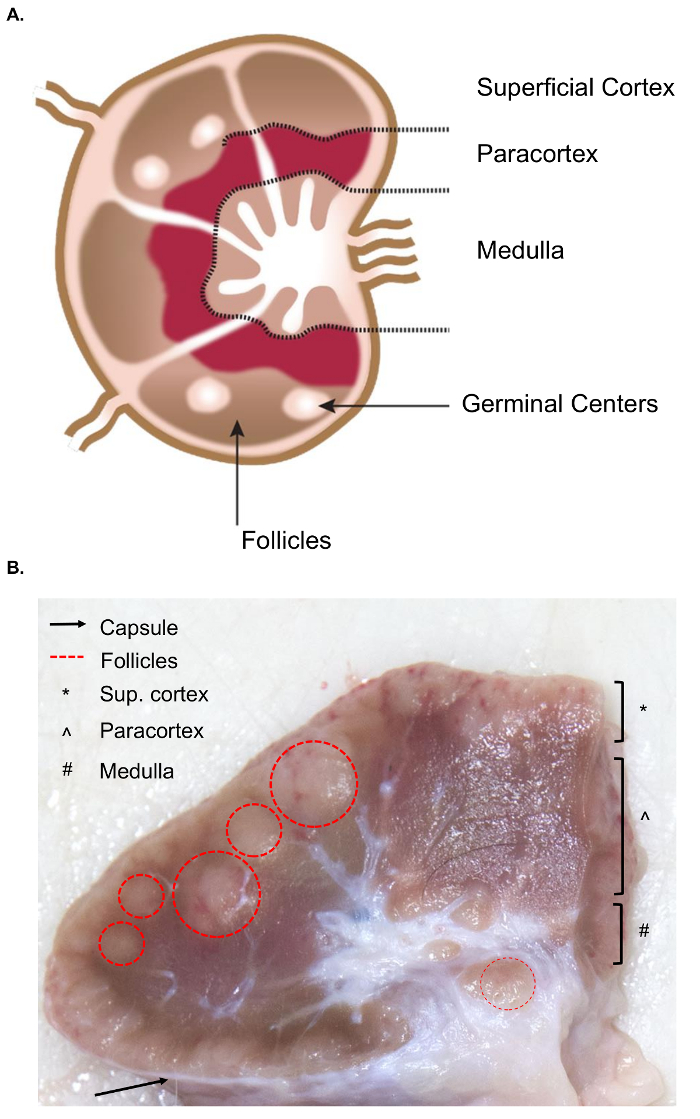
Figura 1. Anatomia do nó de linfa. (A), esta imagem mostra a anatomia do nó de linfa, representando estruturas chaves. (B) ainda esta imagem mostra um nó de linfa bovina cortado em secção transversal. Destacam-se as estruturas relevantes/camadas que são visíveis a olho nu. Clique aqui para ver uma versão maior desta figura.
Dependendo a questão experimental, linfonodos diferentes será de interesse para a recolha e análise. Linfonodos periféricos são aqueles localizados profundamente no tecido subcutâneo. Nos bovinos, linfonodos periféricos ou superficiais frequentemente utilizados na prática clínica e experimental incluem parótida, submandibular, retrofaríngeos, pré-escapulares, pré-femurais (precrural) e superficial inguinal (supramamários nas fêmeas, escrotal nos machos) ( A Figura 2). Na tabela 1, as propriedades de chaves linfáticos superficiais, conforme descrito no gado sistema8, são descritas. Abaixo, alguns planos de recolha de linfonodo potenciais para doenças infecciosas bacterianas, de gado são apresentados como ponto de partida para a investigação.
Brucella abortus/Brucella melitensis: padrão necropsies por b. abortus-infectados de gado e b. melitensis-cabras infectadas no centro nacional de doença Animal recuperar supramamários das, prescapular e tecido de linfonodo parotídeo , tanto para a moagem para a enumeração de bactérias e para a preparação de RNA para o perfil de expressão acolhimento do RNA. B. abortus regularmente podem ser recuperados em cada um destes gânglios linfáticos em bovinos experimentalmente infectados9. A presença de bactérias em cada um desses tipos de nó de linfa pode ser detectada em b. melitensis-infectados cabras até pós-infecção pelo menos nove meses, utilizando as metodologias baseadas em RNA de nossos estudos (Boggiatto et al, inédito). Salmonela SP.: O prescapular, subiliac (pré-femurais), e linfonodos mesentéricos foram úteis durante a criação de perfil de carcaças de gado para uma prevalência de Salmonella 10,11,12 e seria de interesse potencial para estudos de transcriptomic. Escherichia coli O157: H7: linfonodos mesentéricos (no intestino médio e intestino delgado distal locais) podem ser os sites de uma recuperação ocasional das bactérias em bezerros infectados (mas não em bovinos adultos infectados)13. Leptospirose (Leptospira SP.): uma crônica persistência das bactérias tem sido observada em linfonodos drenam a glândula mamária14. Mycobacterium bovis : Nos bovinos, as bactérias foram recuperada infecção pós-experimental de gânglios linfáticos do mediastino e traqueobrônquica de bezerros15. Além disso, o linfonodo RNA tem sido utilizado para examinar as respostas de grande animal hospedeiro de vírus, tais como a síndrome respiratória e reprodutiva suína vírus2. A Figura 2 mostra a localização de um subconjunto desses principais linfonodos do corpo de bovinos.
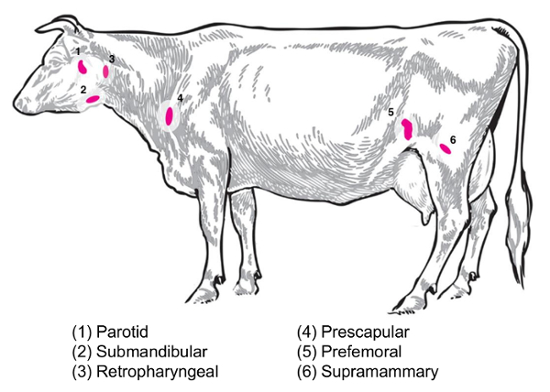
Figura 2: Cartoon retratando locais selecionados do nó de linfa no Bos taurus . Os linfonodos numerados são anotados. Clique aqui para ver uma versão maior desta figura.
Este documento e o vídeo associado, apresentamos um protocolo para o isolamento de grandes linfonodos animais para estudos de RNA, projetados para ser informativo para biólogos moleculares envolvidos em estudos de transcriptomic de infecções de animais grandes. Primeiro, nós fornecemos uma visão geral do procedimento de isolamento para os gânglios linfáticos, usando amostragem de tecidos bovinos e bison como exemplos. Emparelhado com esta demonstração, conforme exibido no vídeo, é um fluxo de trabalho para uma amostra de tecido pode ser reproduzido para isolamento de RNA. Em seguida, descrevemos considerações importantes para o processamento de um linfonodo infectado, com foco na segurança, consistência e qualidade do RNA.
A preparação do RNA do tecido com um reagente de isotiocianato de guanidina-fenol acidificado é baseada no método original de Chomczynski e Sacchi16,17, com uma purificação sobre colunas de rotação silicone-baseado na presença de agentes de caotrópicas baseados no trabalho de Vogelstein e Gillespie18original. Também examinamos o potencial para a recuperação do RNA para transcriptomics de gado gânglios linfáticos preservados por métodos alternativos. Finalmente, podemos explorar o impacto da variável tempo sobre a qualidade do RNA em necropsias de animais grandes, incluindo uma experiência representativa, retratando o efeito de um aumento no tempo entre a eutanásia e a amostragem no perfil de RNA recuperado de bison e linfonodos de bovinos. Este artigo será útil não apenas para biólogos moleculares, mas também ao veterinários pesquisadores iniciando estudos transcriptomic.
Protocol
Representative Results
Discussion
A maioria dos estudos de transcriptomic e os protocolos associados concentrar em amostras humanas, rato, rato ou post-mortem. No entanto, investigações em animais e animais selvagens fornecem uma ampla gama de oportunidades para a caracterização da resposta imune à doença, tanto como aplicáveis à medicina veterinária e, no que diz respeito a doenças zoonóticas, a saúde pública. Este protocolo fornecido um resumo das considerações-chave para extração de RNA de alta integridade de tecidos de animais de gra…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Os autores gostaria de agradecer James Fosse pelo seu excelente trabalho em todos os Videografia e processamento de vídeo; Michael Marti pelo seu excelente trabalho na geração de imagens digitalizadas de gado; Lilia Walther por sua ajuda com a extração do RNA e Bioanalyzer é executado; Mitch Palmer e Carly Kanipe para sua revisão útil e feedback sobre imagens de linfonodo; e os cuidados com animais e pessoal veterinário no centro nacional de doença Animal para todos de seu trabalho árduo e assistência com a pecuária e a preparação para necropsias.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
References
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

