Recogida y tratamiento de los ganglios linfáticos de animales grandes para el análisis de ARN: preparación para estudios de nodo de linfa transcriptómicos de grandes especies animales
Summary
Este protocolo proporciona un resumen de los procedimientos para el aislamiento de ARN para los perfiles transcriptómicos de nodo de linfa tejidos de animales grandes, incluyendo pasos en la identificación y extirpación de los ganglios linfáticos de ganado y la fauna, métodos de muestreo para proporcionar consistencia en varios animales y consideraciones más resultados representativos para la conservación de la colección y procesamiento de análisis de ARN.
Abstract
Animales grandes (ganado y vida silvestre) sirven como importantes reservorios de patógenos, incluyendo Brucella, Salmonella, Mycobacterium bovisy e. coliy son útiles para el estudio de la patogenia y diseminación de las bacterias en hospederos naturales. Con la función clave de la respuesta inmune del huésped de los ganglios linfáticos, ganglios linfáticos tejidos sirven como una fuente potencial de RNA para análisis transcriptómico aguas abajo, para evaluar los cambios temporales en la expresión génica en las células en el transcurso de una infección. Este artículo presenta un resumen del proceso de la colección de nodos de linfa, tejido muestreo y posterior procesamiento del ARN en ganadería, con ganado bovino (Bos taurus) como un modelo, ejemplos adicionales de bisonte americano (bisonte del bisonte de ). El protocolo incluye información sobre la ubicación, identificación y extirpación de ganglios linfáticos de múltiples sitios claves en el cuerpo. Además, se presenta una metodología de toma de muestras de biopsia que permite una consistencia de muestreo a través de múltiples animales. Se discuten varias consideraciones para la preservación de la muestra, incluyendo la generación de ARN adecuado para aguas abajo metodologías como la secuencia de RNA y RT-PCR. Debido a los retrasos inherentes en grandes animales vs ratón tiempo estudia, se presentan los resultados representativos de bisonte y los tejidos bovinos del nodo de linfa para describir el curso temporal de la degradación en este tipo de tejido, en el contexto de una revisión de anterior trabajo metodológico en la degradación del RNA en otros tejidos. En general, este protocolo servirá a ambos investigadores veterinarios comenzando proyectos de transcriptoma en muestras de animales grandes y a biólogos moleculares interesados en aprender técnicas para el muestreo de tejido en vivo y en vitro procesamiento.
Introduction
Secuenciación del RNA análisis del transcriptoma de los ganglios linfáticos proporciona la oportunidad de caracterizar la respuesta inmune de los animales a una variedad de patógenos. Si bien esta metodología se ha utilizado extensivamente en ratones, análisis han ampliado recientemente en grandes mamíferos1,2. Ganadería y grandes ganglios animal puede utilizarse para caracterizar las respuestas de host a una infección, no sólo para su uso en vacunas o estudios de susceptibilidad genética y para la identificación de dianas para el desarrollo de fármacos, sino también como sistemas modelo para estudios en seres humanos en las enfermedades zoonóticas. Por ejemplo, en el caso de brucelosis (una enfermedad bacteriana zoonótica que impactos de medio millón personas alrededor del mundo cada año), a pesar de aumentó significativamente los costos de estudios en ovejas o cabras son más relevantes a la infección humana y vacunas humanas desarrollo de modelos animales de laboratorio. Modelos de infección del ratón recapitulan la infección del sistema reticuloendotelial, pero no los signos clínicos característicos3.
En experimentos con animales grandes en comparación con los estudios animales de laboratorio, el proceso de tejido recolección necesariamente implica un retraso largo entre la eutanasia y la colección de tejidos, que presenta un desafío potencial para la conservación de ARN de alta calidad. ARN intacto es esencial para la generación de datos transcriptómicos biológicamente relevantes. La generación de ARN de alta calidad de las muestras de tejido es particularmente crítica para estudios de patógeno animal grande llevó a cabo en las instalaciones de contención. Tales estudios son inherentemente más difíciles de realizar ya que no sólo requieren aprobado instalaciones y con personal altamente capacitado, sino también llevan costos financieros significativos, que, dependiendo de la obra, pueden variar desde decenas hasta cientos de miles de dólares. Este tipo de estudios también implica una colaboración interdisciplinaria y conocimiento interdisciplinario para su terminación, añadiendo a su complejidad. Por lo tanto, entrenamiento, desarrollo de y la adherencia a un sistema simplificado para la recogida de muestras y conservación proporciona ventajas significativas para posteriores estudios moleculares de los tejidos de animales infectados.
La colección más grandes de nodos de linfa presenta retos adicionales para la colección de tejido en comparación con la toma de muestras similar de ganglios murinos. La preparación para la supresión de la muestra requiere un entendimiento básico de la anatomía de los ganglios linfáticos, incluyendo las estructuras internas pertinentes. La estructura de un nodo de linfa se compone de lobulillos linfoides rodeados por senos llenados de linfa. Estas estructuras están encerradas dentro de una cápsula resistente, fibrosa. 4 un lóbulo linfoide es la “base anatómica y funcional unidad del ganglio linfático” y se compone de folículos, una unidad profunda cortical y cordones medulares y senos paranasales4 (figura 1A). Los linfocitos B y T son el hogar de los folículos y profundidad corticales unidades, respectivamente. Estas estructuras proporcionan un andamio 3D y facilitan la interacción entre los linfocitos y el antígeno o células presentadoras de antígeno.
Grueso, folículos y unidades de profundidad corticales pueden ser identificadas en superficie del corte ya que contienen una red reticular densa y aparecen más oscuros que los senos paranasales, que están compuestos por una red reticular más delicada y aparecen más ligeros (figura 1B). Por Convención, patólogos se refieren a las regiones de los ganglios linfáticos como la corteza superficial (folículos), el paracortex (unidades de profundidad corticales) y la médula (cordones medulares y los senos paranasales). Un examen adecuado de las tres regiones se ha considerado como la mejor práctica en pautas de rutina examinación patológica de ganglios5. Tenga en cuenta que existe una considerable variación en la consistencia, tamaño y color de los ganglios linfáticos, incluso dentro de un solo animal. Edad de los animales, sus nodos de linfa tenderá a disminuir en tamaño y ser más firme que los de animales más jóvenes, generalmente debido a un aumento en su tejido conectivo y una reducción del linfoide normal estructura6,7.
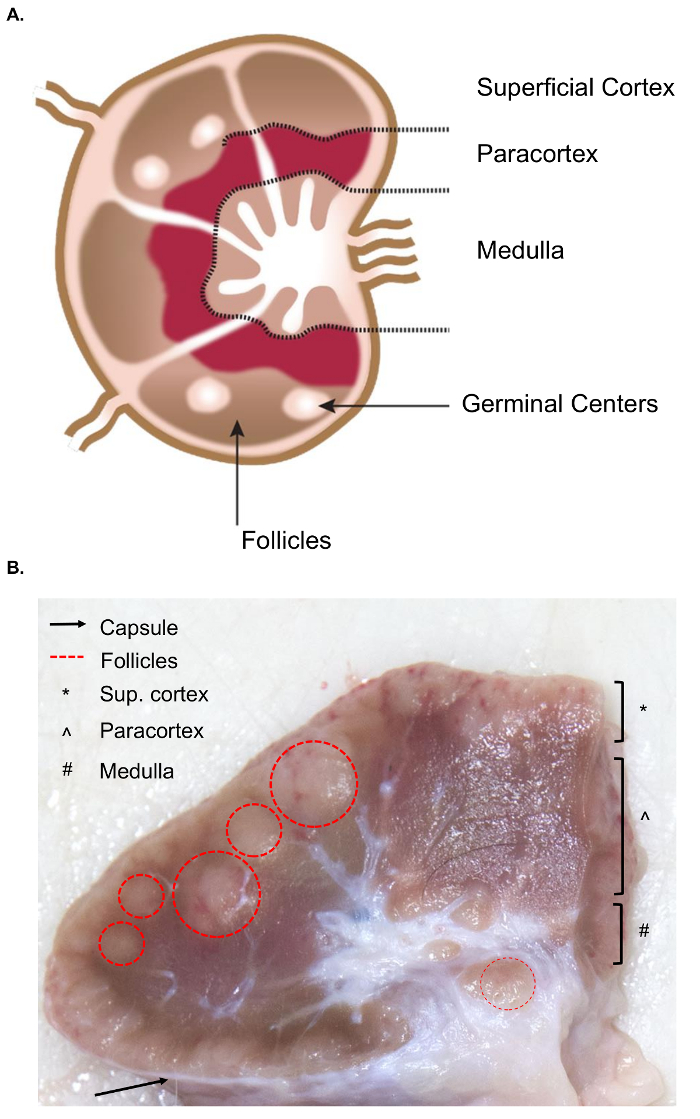
Figura 1. Anatomía de los ganglios linfáticos. (A) esta caricatura muestra la anatomía de los ganglios linfáticos, estructuras claves. (B) todavía la imagen muestra un nodo de linfa bovino cortado transversalmente. Se destacan las estructuras pertinentes/capas que son visibles a simple vista. Haga clic aquí para ver una versión más grande de esta figura.
Dependiendo de la pregunta experimental, diversos ganglios linfáticos será de interés para la recolección y análisis. Los ganglios linfáticos periféricos son los situados profundamente en el tejido subcutáneo. En el ganado, los ganglios linfáticos periféricos o superficiales de uso frecuente en la práctica clínica y experimental incluyen parótida, submaxilar, retrofaríngeo, preescapular, prefemoral (precrural) y superficial inguinal (supramamaria en escrotal en los machos de las hembras) () Figura 2). En la tabla 1, las propiedades de los principales ganglios linfáticos superficiales, como se describe en el sistema de ganados8, se describen. A continuación, se presentan algunas posibles planes de colección de nodo de linfa para enfermedades infecciosas bacterianas del ganado como punto de partida para la investigación.
Brucella abortus/Brucella melitensis: estándar de necropsias para B. abortus-infectado ganados y B. melitensis-cabras infectadas en el Centro Nacional de enfermedad Animal recuperan supramamaria prescapular y tejido ganglionar parotidea , tanto para la molienda para la enumeración bacteriana y para la preparación de RNA para los perfiles de expresión de RNA del anfitrión. B. abortus puede recuperarse regularmente en cada uno de estos nodos de linfa en bovinos experimentalmente infectados9. La presencia de bacterias en cada uno de estos tipos de nodo de linfa puede detectarse en B. melitensis-infectados cabras hasta infección posterior por lo menos nueve meses usando las metodologías basadas en RNA de nuestros estudios (Boggiatto et al., inédito). Salmonella SP.: prescapular, subiliac (prefemoral), y nodos de linfa mesentéricos han sido útiles durante la elaboración de perfiles de las canales de ganado para una prevalencia de Salmonella 10,11,12 y sería de interés potencial para estudios transcriptómico. E. coli O157: H7: nodos de linfa mesentéricos (en el intestino medio e intestino delgado distal) pueden ser los sitios de una recuperación ocasional de la bacterias en terneros infectados (pero no en ganado adulto infectado)13. Leptospirosis (Leptospira SP.): una crónica persistencia de la bacteria se ha observado en los ganglios linfáticos que drenan la glándula mamaria14. Mycobacterium bovis : En el ganado, las bacterias han sido recuperada post experimental infección de los ganglios linfáticos mediastínicos y traqueobronquiales de becerros15. Además, RNA del nodo de linfa se ha utilizado para examinar las respuestas de gran anfitrión animal al virus, como la del virus del síndrome reproductor y respiratorio porcino2. Figura 2 muestra la ubicación de un subconjunto de estos grandes nodos de linfa en el cuerpo del ganado.
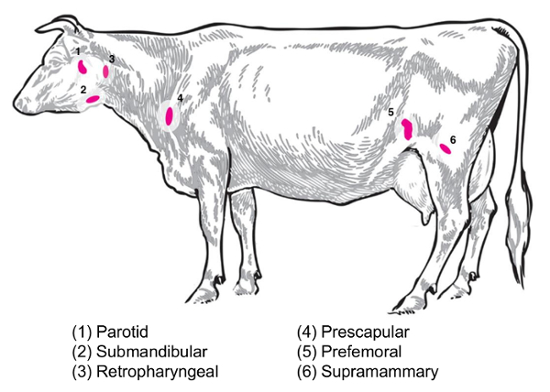
Figura 2: Dibujos que representan lugares seleccionados del nodo de linfa en Bos taurus . Los nodos de linfa números anotados. Haga clic aquí para ver una versión más grande de esta figura.
En este artículo y el video asociado, se presenta un protocolo para el aislamiento de grandes ganglios animal para estudios de RNA, diseñados para ser informativa para biólogos moleculares involucrados en estudios transcriptómicos de infecciones animales grandes. En primer lugar, ofrecemos un resumen del procedimiento de aislamiento para los nodos de linfa, usando el muestreo de tejidos bovinos y Bisonte como ejemplos. Junto con esta demostración, como se muestra en el video, es un flujo de trabajo para una muestra de tejido reproductivo para el aislamiento de RNA. A continuación, se describen consideraciones importantes para el procesamiento de un ganglio infectado, con un enfoque en calidad de RNA, consistencia y seguridad.
La preparación del ARN del tejido con un reactivo de isotiocianato de guanidina de fenol acidificado se basa en el original método de Chomczynski y Sacchi16,17, con una purificación sobre columnas spin basados en sílice en presencia de caotrópico agentes basados en la obra original de Vogelstein y Gillespie18. También examinamos el potencial para la recuperación del ARN para transcriptómica de los ganglios linfáticos de vacas conservada por métodos alternativos. Finalmente, exploramos el impacto de la variable tiempo en la calidad del RNA en necropsias de animales grandes, incluyendo un experimento representativo que representa el efecto de un aumento en el tiempo entre la eutanasia y el muestreo del perfil de RNA recuperado de bisonte y bovina de los nodos de linfa. Este artículo será útil no sólo para biólogos moleculares, pero también para veterinarios investigadores comenzar estudios transcriptómico.
Protocol
Representative Results
Discussion
La mayoría de estudios transcriptómico y los protocolos asociados se centran en ratón, rata o post-mortem de muestras humanas. Sin embargo, investigaciones en ganadería y fauna proporcionan una amplia gama de oportunidades para la caracterización de la respuesta inmune a la enfermedad, como aplicable a la medicina veterinaria y, en relación con las enfermedades zoonóticas, de salud pública. Este protocolo proporciona un resumen de las más importantes consideraciones para la extracción de RNA de alta integridad …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Los autores desean agradecer a James Fosse por su excelente trabajo sobre todo videografía y procesamiento de vídeo; Michael Marti por su excelente trabajo en la generación de imágenes digitalizadas de ganado; Lilia Walther por su ayuda con la extracción de RNA y Bioanalyzer funciona; Mitch Palmer y Carly Kanipe por su útil informe y comentarios en imágenes de nodo de linfa; y el cuidado de los animales y el personal veterinario del Centro Nacional de enfermedades animales por todo su trabajo duro y ayuda con la cría de animales y la preparación para necropsias.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
References
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

