Insamling och bearbetning av lymfkörtlar från stora djur för RNA analys: förbereda för lymfkörtel Transcriptomic studier av stora djurarter
Summary
Detta protokoll ger en översikt över förfarandena för isolering av RNA för transcriptomic profilering av lymfkörtel vävnader från stora djur, inklusive steg i identifiering och excision av lymfkörtlar från boskap och vilda djur, provtagning metoder att tillhandahålla konsekvens över flera djur, och överväganden plus representativa resultat för efter samling bevarande och bearbetning för RNA analys.
Abstract
Stora djur (både boskap och vilda djur) fungera som viktiga reservoarer av zoonotiska patogener, inklusive Brucella, Mycobacterium bovis, Salmonellaoch e.coli, och är användbara för att studera patogenes och/eller spridningen av bakterier i naturliga värdar. Med viktiga funktionen av lymfkörtlar i värd immunsvaret fungera lymfkörtel vävnader som en potentiell källa till RNA för nedströms transcriptomic analyser, för att bedöma de tidsmässiga förändringarna i genuttryck i celler under loppet av en infektion. I denna artikel presenteras en översikt över processen för lymfkörtel insamling, vävnad provtagning och nedströms RNA bearbetning i boskap, med nötkreatur (Bos taurus) som förebild, med ytterligare exempel från den amerikanska bison (Bison bison ). Protokollet innehåller information om plats, identifiering och avlägsnande av lymfkörtlar från flera viktiga platser i kroppen. Dessutom presenteras en biopsi provtagning-metodik som möjliggör en konsistens av provtagning över flera djur. Flera överväganden för provet bevarande diskuteras, med framtagning av RNA passar nedströms metoder som RNA-sekvensering och RT-PCR. På grund av de långa förseningarna inneboende i stora djur vs mus tid kursen studier presenteras representativa resultat från bison och bovin lymfkörtel vävnader för att beskriva tidsförloppet för nedbrytning i denna vävnadstyp, inom ramen för en översyn av tidigare metodarbete på RNA nedbrytning i andra vävnader. Sammantaget kommer detta protokoll vara användbart till både veterinärmedicinska forskare börjar transkriptom projekt på stora djur prover och molekylärbiologer som är intresserade av att lära sig tekniker för i vivo vävnad provtagning och in vitro- bearbetning.
Introduction
RNA-sekvensering analys av transkriptom lymfkörtlar ger möjlighet att karakterisera immunsvaret hos djur till en mängd olika patogener. Medan denna metod har använts flitigt i möss, har analyser nyligen expanderat in i större däggdjur1,2. Boskap/stora djur lymfkörtlar kan användas för att karaktärisera värd Svaren till en infektion, inte bara för deras användning i vaccin eller genetisk känslighet studier och identifiering av mål för läkemedelsutveckling, men också som modellsystem för studier på människa på zoonotiska sjukdomar. Till exempel i fråga om brucellos (en zoonotiska bakteriell sjukdom som konsekvenser en halv miljon människor runt om i världen varje år), trots kraftigt ökade kostnader, studier hos får eller getter är mer relevanta för mänskliga infektion och vaccin mot humant utveckling än laboratoriet djurmodeller. Infektion musmodeller recapitulate retikuloendoteliala systemet infektionen men inte de karakteristiska kliniska tecken3.
I stora djurförsök jämfört med laboratorium djurstudier omfattar processen för vävnad skörd nödvändigtvis en längre fördröjning mellan dödshjälpen och vävnad samlingen, som en potentiell utmaning för bevarandet av hög kvalitet RNA. Intakt RNA är viktigt för generering av biologiskt relevanta transcriptomic data. Generering av hög kvalitet RNA från vävnadsprover är särskilt kritiskt för stora djur patogen studier utfördes i inneslutning faciliteter. Sådana studier är till sin natur mer svårt att genomföra eftersom de inte bara kräver godkänd faciliteter och högt utbildad personal men utför också betydande finansiella kostnader, som, beroende på arbetet, kan variera från tiotals till hundratals tusen dollar. Dessa typer av studier också innebära en tvärvetenskaplig samverkan och tvärvetenskapliga kunskap för deras slutförande, lägga till deras komplexitet. Därför, utbildning, utveckling av och adherencen till ett förenklat system för provtagning och bevarande ger betydande fördelar för nedströms molekylära studier av vävnader från smittade djur.
Insamling av större lymfkörtlar presenterar ytterligare utmaningar för samlingen vävnad jämfört med liknande provtagning av murina lymfkörtlar. Förberedelserna för det provet excision nödvändiggör en grundläggande förståelse av anatomin i lymfkörteln, inklusive de relevanta interna strukturerna. En lymfkörtel struktur består av lymfoida lobules omgiven av bihålor fyllda med lymfan. Dessa strukturer är innesluten i en tuff, fibrösa kapsel. 4 en lymfoida lobule är ”grundläggande anatomiska och funktionella enhet lymfkörtel” och består av folliklar, en djupt kortikala enhet, och medullär sladdar och bihålorna4 (figur 1A). T och B-lymfocyter är hem till hårsäckarna och djupt kortikala enheter, respektive. Dessa strukturer ger en 3D klätterställning och underlätta interaktionen mellan lymfocyter och antigen eller antigenpresenterande celler.
Grovt, folliklar och djupt kortikala enheter kan identifieras på snittytan som de innehåller en tätare retikulära meshwork och blir mörkare än bihålorna, som består av en mer delikat retikulära meshwork och ljusare (figur 1B). Konventionen hänvisa patologer till regioner i lymfkörtlarna som ytliga cortex (folliklar), paracortex (djupt kortikala enheter) och medulla (medullär sladdar och bihålorna). En ordentlig undersökning av alla tre regionerna har bedömts som bästa praxis i patologiska rutinundersökning riktlinjer för lymfkörtlar5. Observera att det finns en betydande variation i konsistens, storlek och färg av lymfkörtlar, även inom ett enda djur. Som djur ålder, deras lymfkörtlar tenderar att minska i storlek och blir fastare än hos yngre djur, vanligtvis på grund av en ökning av deras bindväv och en minskning av normalt lymfoida struktur6,7.
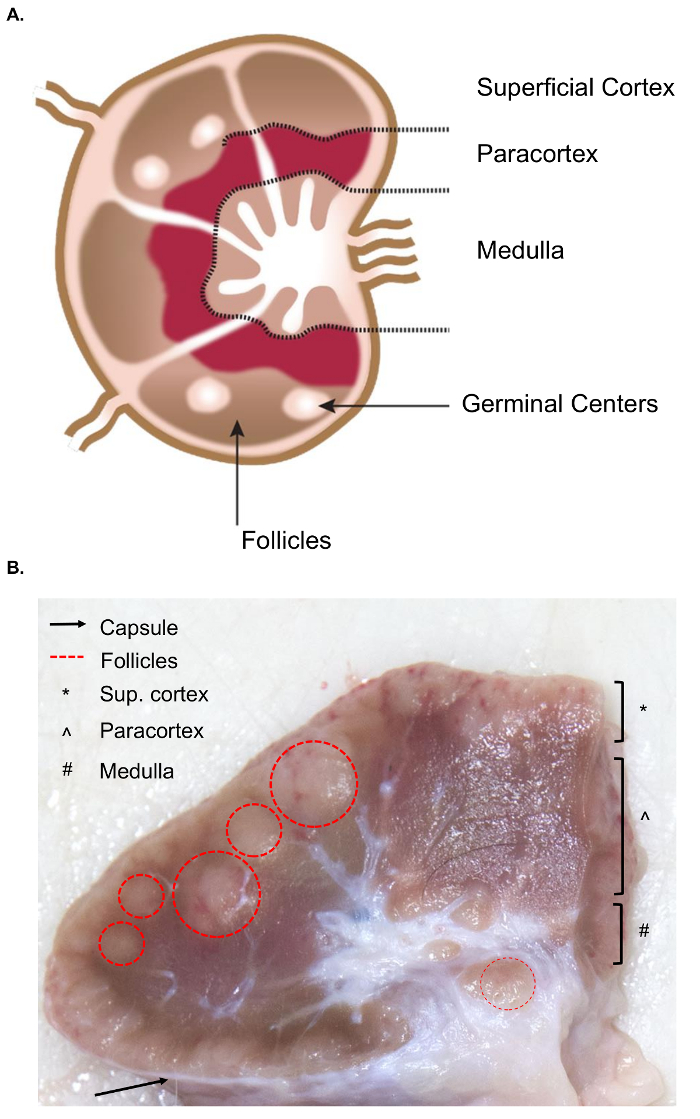
Figur 1. Anatomi av lymfkörteln. (A) den här tecknade bilden visar anatomin av lymfkörteln, som skildrar viktiga strukturer. (B) denna fortfarande bild visar en nötkreatur lymfkörtel som skär i tvärsnitt. De relevanta strukturer/lager som är synliga för blotta ögat markeras. Klicka här för att se en större version av denna siffra.
Beroende på den experimentella frågan, kommer att olika lymfkörtlar vara av intresse för insamling och analys. Perifera lymfkörtlar är de som ligger djupt i den subkutana vävnaden. Hos nötkreatur, perifer eller ytliga lymfkörtlar som ofta används i klinisk och experimentell praktiken inkluderar parotis, submandibular, retrofaryngeal, lymfknutor, prefemorala (precrural) och ytliga ljumsk (juvret hos kvinnor, testikelcancer hos hanar) () (Se figur 2). I tabell 1beskrivs egenskaperna för nyckel ytliga lymfkörtlar, som beskrivs i den boskap system8. Nedan presenteras några potentiella lymfkörtel samling planer för bakteriella infektionssjukdomar av nötkreatur som utgångspunkt för utredningen.
Brucella abortus/Brucella melitensis: Standard necropsies för B. abortus-smittade nötkreatur och B. melitensis-infekterade getter vid National Animal sjukdom Center återställa juvret, prescapular och parotis lymfkörteln vävnad , både för malning för bakteriell uppräkningen och för RNA beredning för värd RNA uttryck profilering. B. abortus kan återvinnas regelbundet i varje av dessa lymfkörtlar i experimentellt infekterade nötkreatur9. Förekomsten av bakterier i varje av dessa lymfkörtel typer kan upptäckas i B. melitensis-infekterade getter upp till minst nio månader efter infektion med RNA-baserade metoderna från våra studier (Boggiatto et al., opublicerad). Salmonella sp.: prescapular, subiliac (prefemorala), och mesenteriala lymfkörtlar har varit användbart under profilering av slaktkroppar av nötkreatur för en Salmonella prevalensen10,11,12 och skulle vara potentiellt intresse för transcriptomic studier. E. coli O157: H7: mesenteriala lymfkörtlar (vid mellersta tunntarmen och distala tunntarmen platser) kan vara en tillfällig återhämtning av bakterier i infekterade kalvar (men inte i infekterade vuxna nötkreatur)13webbplatser. Leptospiros (Leptospira sp.): en kronisk persistens av bakterier har observerats i lymfkörtlarna dränering bröstkörteln14. Mycobacterium bovis : Hos nötkreatur varit bakterierna återställda efter experimentell infektion från lymfkörtlarna mediastinum och tracheobronchial kalvar15. Dessutom har lymfkörtel RNA använts för att undersöka stora djurens värd Svaren till virus, till exempel de svin porcine respiratory syndrome virus2. Figur 2 visar platsen för en delmängd av dessa stora lymfkörtlar i nötkreatur kroppen.
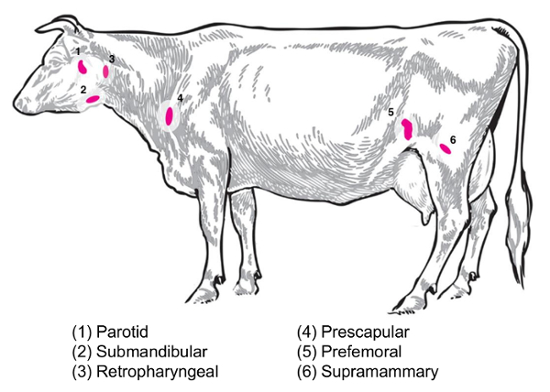
Figur 2: Tecknad som skildrar valda lymfkörtel platser i Bos taurus . Numrerade lymfkörtlarna är kommenterad. Klicka här för att se en större version av denna siffra.
I detta papper och tillhörande videon presenterar vi ett protokoll för isolering av stora djur lymfkörtlar för RNA studier, utformad för att vara informativa för molekylärbiologer inblandade i transcriptomic studier av stora djur infektioner. Först, vi ger en översikt över förfarandet isolering för lymfkörtlarna, med provtagning från nötkreatur och bison vävnader som exempel. Parat med denna demonstration, som visas i videon, är ett arbetsflöde för en reproducerbar vävnad provtagning för RNA isolering. Därefter beskriver vi viktiga överväganden för bearbetning av en infekterad lymfkörteln, med fokus på säkerhet, konsekvens och RNA kvalitet.
Beredning av RNA från vävnaden med en surgjord fenol-guanidin isotiocyanat reagens är baserad på den ursprungliga metoden Chomczynski och Sacchi16,17, med en rening över kiselbaserade spin kolumner i närvaro av chaotropic agenter baserat på det ursprungliga arbetet Vogelstein och Gillespie18. Vi också undersöka potentialen för återvinning av RNA för transkriptomik från nötkreatur lymfkörtlar konserverade med alternativa metoder. Slutligen, vi utforskar effekten av variabeln tid på RNA kvaliteten i stora djur necropsies, inklusive ett representativt experiment som skildrar effekten av en ökning av tid mellan dödshjälpen och provtagning på återvunna RNA profil från bison och bovin lymfkörtlar. Denna artikel kommer att vara användbart inte bara molekylärbiologer utan också till veterinärmedicinska forskare påbörjas transcriptomic studier.
Protocol
Representative Results
Discussion
Majoriteten av transcriptomic studier och tillhörande protokoll fokuserar på mus, råtta eller postmortala mänskliga prover. Utredningar i boskap och vilda djur är dock ett brett utbud av möjligheter för karakterisering av immunsvaret mot sjukdom, både som veterinärmedicin och, när det gäller zoonotiska sjukdomar, att människors hälsa. Detta protokoll föreskrivs en disposition av huvudprioriteringar för hög integritet RNA-extraktion från vävnader från stora djur, som nötkreatur, bison, getter och får….
Divulgations
The authors have nothing to disclose.
Acknowledgements
Författarna vill tacka James Fosse för hans utmärkta arbete på alla videography och video bearbetning; Michael Marti för hans utmärkta arbete i generation av digitaliserade nötkreatur bilder; Lilia Walther för hennes hjälp med RNA-extraktion och Bioanalyzer körningar; Mitch Palmer och Carly Kanipe för deras hjälpsamma granskning och återkoppling på lymfkörtel bilder; och djurvård och veterinära personalen vid National Animal sjukdom Center för alla deras hårda arbete och hjälp med djurhållning och förberedelserna för obduktion.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
References
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

