Gel-seq: En metode til samtidige sekventering bibliotek forberedelse af DNA og RNA bruger Hydrogel matricer
Summary
Gel-seq gør det muligt for forskere at samtidig forberede biblioteker til både DNA – og RNA-seq ubetydelige ekstra omkostninger fra 100-1000 celler ved hjælp af en simpel hydrogel enhed. Dette paper præsenterer en detaljeret tilgang til fabrikation af enheden samt den biologiske protokol til at generere parrede biblioteker.
Abstract
Evnen til at forstærke og enten DNA eller RNA-sekvens fra små start prøver er kun opnået i de sidste fem år. Desværre standardprotokoller til generering af genomisk eller transkriptom biblioteker er inkompatibel og forskere skal vælge om du vil DNA eller RNA-sekvens for en særlig prøve. Gel-seq løser dette problem ved at aktivere forskere at samtidig forberede biblioteker til både DNA og RNA starter med 100-1000 celler ved hjælp af en simpel hydrogel enhed. Dette paper præsenterer en detaljeret tilgang til fabrikation af enheden samt den biologiske protokol til at generere parrede biblioteker. Vi designet Gel-FF., så det let kunne gennemføres af andre forskere; mange genetik labs har allerede det nødvendige udstyr til at reproducere Gel-seq enhed fabrikation. Vores protokollen beskæftiger almindeligt anvendte kits til både hele-udskrift forstærkning (WTA) og bibliotek forberedelse, som også vil kunne være bekendt at forskere allerede bevandret i generering af genomisk og transkriptom biblioteker. Vores tilgang gør det muligt for forskerne at lægge både DNA og RNA sekventering magt på en enkelt prøve uden opdeling og med ubetydelige ekstra omkostninger.
Introduction
Næste generation sequencing (NGS) har haft en dybtgående indvirkning på den måde genetik forskes. Hvor forskere en gang fokuseret på sekvensering genomet af et hele arter, er det nu muligt at sekvens genomet af et enkelt tumor eller endda en enkelt celle i et eksperiment. 1 NGS har også gjort det omkostningseffektivt at sekvens RNA afskrifter findes i en celle, en samling af data er kendt som transkriptom. Evnen til at forstærke og enten DNA eller RNA-sekvens fra små start prøver er kun opnået i de sidste fem år. 2 , 3 , 4 desværre standardprotokoller er inkompatibel og forskere skal vælge om du vil DNA eller RNA-sekvens for en given prøve. Når en start prøve er tilstrækkelig stor, kan det være delt i halve. I mindre målestok, men tab af materiale på grund af opdelingen prøver kan påvirke bibliotek kvalitet, og pooling af prøver kan gennemsnit ud interessante variationer mellem celler. 5 endvidere forskere er mere og mere interesseret i at undersøge prøver, der ikke kan opdeles, som enkelt celler eller små heterogene tumor biopsier. 6
For at løse dette problem, tre protokoller har for nylig blevet udviklet sekvens både DNA og RNA fra det samme udgangspunkt prøve: Gel-FF.7, G & T-FF.8og DR-seq9. Denne artikel præsenterer en detaljeret protokol for Gel-FF., hvilke kan bruges hen til samtidig generere DNA og RNA biblioteker fra så lidt som 100 celler på ubetydelige ekstra omkostninger. Det nye aspekt af Gel-seq er evnen til at adskille DNA og RNA baseret udelukkende på størrelse ved hjælp af lave omkostninger hydrogel matricer. Core innovation af Gel-Seq-protokollen er den fysiske adskillelse af DNA fra RNA. Denne adskillelse er opnået electrophoretically ved hjælp af en kombination af polyacrylamid membraner, at udnytte size forskelle mellem disse molekyler. For at sætte disse størrelse forskelle i sammenhæng, overveje hvordan DNA og RNA er afbildet: mens DNA findes på mikro-skalaen og kan ses ved hjælp af traditionelle mikroskoper, RNA findes på nanometer skala og må være afbildet ved hjælp af komplekse teknikker såsom cryo-elektron mikroskopi. 10
Tilgang til adskiller DNA og RNA i denne protokol er vist i figur 1. Panelet til venstre viser DNA og RNA gratis flyder i løsning i nærheden af en membran. Når et elektrisk felt anvendes, som vist i højre panel, opleve DNA og RNA en elektroforese kraft, der inducerer migration gennem membranen. Ved tuning membran egenskaber, har vi lavet en semipermeabel membran, der adskiller DNA fra RNA. DNA-molekyler er skubbet mod membranen, men bliver viklet ind i kanten på grund af deres store størrelse. Små RNA molekyler, på den anden side kan omkonfigurere og væver deres vej gennem membranen. Denne proces, som kaldes reptation, er lig den måde en slange bevæger sig gennem græsset. I sidste ende disse RNA molekyler er stoppet af en anden, high-density membran, der er for svært for selv mindre polymerer (> 200 basepar) at slippe gennem. Når fysisk adskilt, kan DNA og RNA inddrives og behandles for at generere oplysninger om både genom og transkriptom. Mens vi kan adskille DNA og RNA, har vi fundet der opnås bedre resultater, hvis RNA er omvendt transskriberet til cDNA før adskillelse. CDNA/RNA hybrider er mere stabile end RNA alene og kan stadig passere gennem low-density membranen.
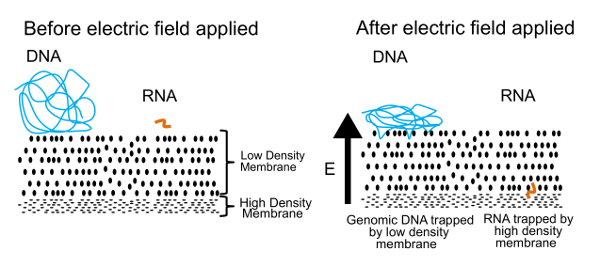
Figur 1 . Gel-seq opererer princippet. Det underliggende princip bruges til fysisk separat DNA og RNA. I en anvendt elektrisk felt, små RNA molekyler vandrer gennem den low-density membran men store DNA molekyler er fanget på overfladen. Dette tal blev gengivet fra Ref. 7 med tilladelse fra Royal Society of Chemistry. Venligst klik her for at se en større version af dette tal.
Dette papir beskriver i detaljer både fabrikation af Gel-seq-enheden og den biologiske protokol til at generere parret DNA og RNA biblioteker. En oversigt over både er vist i figur 2. Enheden er fremstillet af lagdeling tre forskellige tæthed polyacrylamidgeler oven på hinanden i en proces svarende til at skabe standard stabling geler. 11 den biologiske protokol starter med 100-1000 celler suspenderet i PBS. Cellerne er mængden og RNA er omdannet til cDNA, før enheden bruges til at adskille genomisk DNA fra cDNA/RNA hybrider. Efter adskillelse og genvinding, genomisk og transkriptom er biblioteker udarbejdet ved hjælp af en proces, der nøje følger standard hele-genom bibliotek forberedelse kit protokol. Yderligere detaljer om udvikling og validering af Gel-seq kan læses i laboratoriet på en Chip publikation “Gel-seq: hele-genom og transkriptom sekventering af samtidige pesticidanvendelse DNA og RNA bibliotek forberedelse ved hjælp af semi-gennemtrængelige hydrogel barrierer .” 7
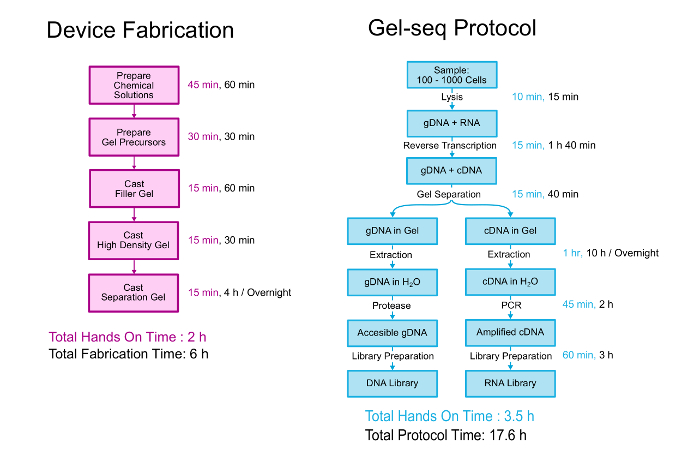
Figur 2 . Gel-seq protokol. En oversigt over trinene til at fabrikere Gel-seq enhed og protokollen til genereret parret DNA og RNA biblioteker. Dele af dette tal var gengivet fra Ref. 7 med tilladelse fra Royal Society of Chemistry. Venligst klik her for at se en større version af dette tal.
For at generere DNA og RNA biblioteker fra enkelt celler, forskere bør overveje at bruge enten G & T-seq eller DR-FF. G & T-FF., ligesom Gel-FF., er baseret på en fysisk adskillelse af RNA fra genomisk DNA. Denne tilgang er baseret på messenger RNA (mRNA) 3′ polyadenylated hale som en pull-down mål. MRNA er fanget på en magnetisk bead ved hjælp af en biotinylated oligo-dT primer. Når mRNA er blevet fanget perlerne er holdt på plads med en magnet, og supernatanten indeholdende genomisk DNA kan fjernet og overført til et andet rør. Efter denne fysiske adskillelse er komplet, kan separate biblioteker genereres fra mRNA og DNA. 8 denne fremgangsmåde fungerer godt, hvis RNA af interesse er polyadenylated, men det kan ikke bruges til at studere ikke-polyadenylated udskrifter, såsom ribosomale RNA, tRNA, eller RNA fra prokaryoter.
DR-seq bygger på et før forstærkning skridt hvor både DNA og cDNA afledt af RNA forstærkes i det samme rør. Prøven er derefter delt i to og behandles sideløbende at forberede DNA – og RNA-seq biblioteker. For at skelne mellem genomisk DNA og cDNA afledt af RNA, tager DR-seq en beregningsmæssige tilgang. Sekvenser, hvor kun exons er til stede er beregningsmæssigt undertrykt i genomisk DNA-data, som de kunne stamme fra enten DNA eller RNA. 9 en fordel ved denne tilgang er at DNA og cDNA/RNA ikke behøver være fysisk adskilt som er gjort i Gel-FF. og G & T-FF. Ulempen, er imidlertid at DR-seq kræver en forudgående viden om genomet og transkriptom (dvs., exons versus introner), og kan ikke være ideel til applikationer såsom sekventering af kerner, hvor mange udskrifter ikke er endnu fuldt splejset og stadig indeholde introns. 12
Det nye aspekt af Gel-seq er evnen til at adskille DNA og RNA i hundredvis af celler baseret udelukkende på størrelse. Denne metode kræver ingen apriori viden om genomet eller transkriptom, er robuste mod ufuldstændige splejsning, og er ikke begrænset til poly-adenylated udskrifter. Til applikationer, hvor en forsker kan begynde med mindst 100 celler, giver Gel-seq en ligetil tilgang ved hjælp af billige og bredt tilgængelige materialer.
Protocol
Representative Results
Discussion
Der er flere kritiske trin forbundet med Gel-seq enhed fabrikation samt selve protokollen. Under fabrikation anbefaler vi at starte med de foreskrevne lag tykkelser for de forskellige regioner af gelen. Vi brugte betydelig tid teste forskellige fabrikation indstillinger og protokollen beskrevet her producerer de bedste enheder til kassetter, der er anført i tabel af materialer og reagenser. Hvis forskere bruger en alternativ kassettesystem, kan de finde det nødvendigt at justere de mængder, der anvend…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Finansiering af dette arbejde blev forudsat ved University of San Diego, National Science Foundation Graduate Research Fellowship Program, NIH give R01-HG007836, og af koreansk Ministeriet for videnskab, IKT og fremtidige planlægning.
Tidligere versioner af en række tal blev først offentliggjort i “Hoople, G. D. et al. Gel-seq: hele-genom og transkriptom sekventering af samtidige pesticidanvendelse DNA og RNA bibliotek forberedelse ved hjælp af semi-gennemtrængelige hydrogel barrierer. Lab på en Chip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). ” Lab på en Chip har sanktioneret genbrug af tal i denne publikation.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
References
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

