細胞間の接触でタンパク質間相互作用の分析計蛍光の変動
Summary
このプロトコルでは、直接細胞内の細胞接合における局在蛋白質すなわち細胞間相互作用を仲介する蛋白質間の相互作用を調査する変動蛍光分光法に基づくアプローチについて説明します。測定器の校正、データ集録および解析、修正可能なアーチファクトのソースを含む詳細なガイドラインを提供します。
Abstract
さまざまな生物学的プロセスには、通常隣接セル界面における相互作用するタンパク質を介した細胞間相互作用が含まれます。関心のだけほとんど試金は、具体的には徹底的な細胞に直接そのような相互作用が可能 します。細胞間の接触で、隣接する細胞の表面に発現するタンパク質の結合を測定するアッセイを紹介します。この試金は 2 つの手順で構成されています: 共焦点レーザー走査型顕微鏡を用いた細胞間の接触で蛍光揺らぎ分光測定が続く異なる蛍光タンパク質を融合した興味の蛋白質を発現する細胞の混合します。我々 は細胞間でアミロイドの前駆物質のような蛋白質 1 (APLP1) の相互作用を測定することによって生物学的文脈でこのアッセイの可能性を実証します。(走査蛍光相互相関分光法、相関数と明るさ解析) 蛍光ベースの技術と計測器の校正を使用してデータ集録の詳細なプロトコルを提供します。さらに、データ分析および識別して退色や細胞の動きのためのそれらなどの外部のスプリアス信号のバリエーションを修正する方法で重要な手順をについて説明します。
一般に、提示されたアッセイはホモ – または同一または異なる種類の細胞間に細胞間の接触、有性タンパク質間相互作用に適用され、商業共焦点レーザー走査型顕微鏡に実装することができます。重要な要件は、いくつかの分以上興味の蛋白質の拡散の動力学を調査するための十分なする必要がありますシステムの安定性です。
Introduction
多くの生物学的プロセスは、細胞間相互作用、例えば細胞間接着1,の2,3細胞融合4細胞認識5のサイトで発生します。このようなイベントは、多細胞生物の細胞間コミュニケーション、例えば、免疫反応時の開発時に特に重要です。これらのプロセスは通常表面、すなわち、隣接する細胞の膜 (PM) にローカライズされてで規制された空間と時間が正確に特定細胞間接触の相互作用を受ける蛋白質によって仲介されます。多くの場合、これらの相互作用は直接ホモ – または血液タンパク質トランス相互作用がイオンまたは細胞外リンカー1として配位子を伴うことがあります。基本的な重要性のプロービング生きている細胞のネイティブ環境で直接これらの特定タンパク質の相互作用アッセイの欠如があります。多くのメソッドいずれか必要セル中断 (co 免疫沈降6など生化学的アッセイなど)、固定 (例えば、いくつかの超解像光学顕微鏡検査の技術や細胞の電子顕微鏡観察7の連絡先)、または、非特定の例えば、集約接着アッセイ8,9/。この問題を克服するためには、蛍光共鳴エネルギー移動 (FRET)10または蛍光補完11に基づく蛍光技術を実施しています。ただし、fluorophores が付いて十分に小さい距離を達成するためにこれらのメソッド、蛋白質10、トランス相互作用と干渉する可能性がある細胞側の蛍光ラベルが必要です。
細胞間の接触でタンパク質間相互作用の代替蛍光を用いた測定を紹介します。このアプローチを組み合わせた蛍光相互相関方法 (スキャン蛍光相互相関分光法 (sFCCS)、相関数と明るさ (ccN と B)) との蛋白質の融合コンストラクトを発現する細胞の混合例えば、接着受容体、関心。2 つの相互作用する細胞の調査受容体が細胞内から 2 つのスペクトル分離蛍光タンパク質 (FPs) が付いています (図 1 aを参照)。
採用方法は、共焦点レーザー走査型顕微鏡の焦点音量を介し蛍光融合タンパク質の拡散運動による蛍光揺らぎの統計分析に基づいています。詳しくは、アッセイは、細胞間の接触の両方の近隣の PMs の興味の蛋白質の共同の拡散をプローブします。タンパク質は、トランス相互作用を受ける、これらトランス錯体は蛍光蛋白質両方のスペクトル チャネルに発光、両エミッタの蛍光相関変動の原因を運ぶ。その一方で、バインディングが発生しない PMs が直面しているタンパク質の数値変動は独立して相関の変動を引き起こしてないになります。買収は、2 つの方法で実行できます: 1) sFCCS 細胞間接触の間でライン状のスキャンに基づいており、プローブ接触領域にあるスポットでの相互作用を効果的に。蛍光変動の時空間分析を通じて sFCCS 情報を提供もダイナミクスすなわち、蛋白質複合体; の拡散係数2) ccN & B は、細胞間の接触領域で取得されるイメージのシーケンスの画素単位の分析に基づいています。プローブする能力を持ってする、ダイナミクスに関する情報は提供されません全体に沿ってマップ相互作用領域 (1 つの焦点面を使用) にお問い合わせください。両方の方法は、分子の明るさ、すなわち、平均蛍光信号は時間単位で単一拡散蛋白質複合体によって放出され、したがって、タンパク質複合体の化学量論の見積もりを提供の分析と組み合わせること細胞間の接触。
この記事では、試料調製、測定器の校正、データ集録および解析商業共焦点レーザー走査型顕微鏡の提示の分析を実行するための詳細なプロトコルを提供します。実験は、フォトンカウンティングまたはアナログ検出器、高開口数と目的を備えてあらゆる楽器で実行できます。さらに、プロトコルの重要な手順について説明し、artefactual 信号の変動、例えば、検出器雑音、退色や細胞の動きを引き起こしているいくつかのプロセスの補正方式を用意しています。もともと付着性のセル間の相互作用をプローブを開発し、アッセイ懸濁細胞の変更可能性がありますまたは適応モデル膜システム、例えば、巨大リポソーム (Guv) または巨大なプラズマ膜小胞 (GPMVs) できるように、異なる脂質環境や組織の骨格12,13の不在には相互作用の定量化。
走査蛍光相互相関分光蛍光相互相関分光法14の修正版は、脂質膜15の遅い拡散ダイナミクスを調べるために設計されました。それは興味の蛍光蛋白質を含んでいる午後に垂直なライン スキャン取得に基づいています。2 つの異なる分類された蛋白質種の相互作用を調査するには、買収はスペクトル分離 fluorophores のレーザーの 2 の行と 2 つの検出ウィンドウを使用して 2 つのスペクトル チャネルで実行されます。PM の蛋白質の遅い拡散ダイナミクスによる (D≤ ~ 1 μ m2/s)、15の行に行から励起方式を交互にクロス話無料測定を実行できます。分析から始まります: block-wise ~ 1000 行、2) 最大蛍光信号、すなわち、 PM 位置各ブロック内の位置の測定の平均と 3) シフトに基づく水平細胞運動のためのアルゴリズムの修正を配置 1)ための共通の起源12,15、各チャンネルで個別にすべてのブロック。その後、PM に対応するピクセルの自動選択は、すべて整列ライン (すなわちセンター ± 2.5σ) の合計のガウス近似から中央の領域を選択するによって実行されます。各ラインの信号の統合により、各チャンネルの膜の蛍光時間シリーズF(t) (g = グリーン チャンネル、r = 赤のチャネル)。ピクセル サイズが十分に小さくなど注意してください < 200 nm のポイントの形状広がり関数と PM の位置に対応する、その中心を見つけます。実質的な退色の存在下で各チャンネルの蛍光時間シリーズが二重指数関数でモデル化し、し、次の数式を修正:16
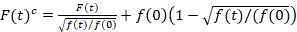 . (1)
. (1)
この数式は、振幅とF(t)c、裸眼F(t)から得られるパラメーターの推定値と比較しての相関分析から得られた拡散時の両方に効果的に補正に注意してくださいすることが重要です。自動・相互相関関数、(各種/艦砲射撃) 蛍光性の信号が計算されます。
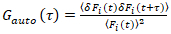 、(2)
、(2)
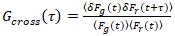 、(3)
、(3)
どこ δFは私私F(t) = 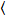 Fは私(t)
Fは私(t)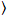 と私= g、r。
と私= g、r。
二次元拡散モデルは、すべての相関関数 (CFs) を装着されています。
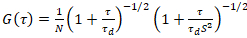 . (4)
. (4)
ここで、 Nは、各チャネルの拡散時間観測量とτd蛍光タンパク質の数を表します。このモデルは、記述されている実験の設定で PM におけるタンパク質の拡散は x-z 平面で発生した蛍光相関の一般的に使用される構成とは対照的分光法 (FCS) 実験膜プロービング考慮共焦点容積17の x y 平面に拡散。腰 w0と構造因子 S、伸長を記述する wz z、S の焦点の容積の wz/w0を =、スペクトルと同様の染料と同じ光設定実行ポイント FCS 校正測定から得られる拡散係数の既に使用可能な値を使用してD染料:
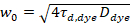 、(5)。
、(5)。
τd、染料がデータを 3次元拡散モデルをフィッティングから得られる色素の測定平均拡散時間を考慮する分数すべてN分子のTのアカウント切り替え、時定数と三重項状態ττ:
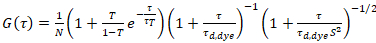 . (6)
. (6)
最後に、拡散係数 (D)、分子の明るさの値 (ε) と sFCCS データ (rel.cc.) の相対的な相互相関は次のように計算されます。
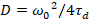 、(7)
、(7)
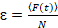 、(8)
、(8)
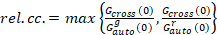 、(9)
、(9)
相互相関関数の振幅Gクロス(0) と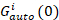 は、 i番目のチャネル チャネルの自己相関関数の振幅。
は、 i番目のチャネル チャネルの自己相関関数の振幅。
この定義の相対の相関、すなわち方程式 9 で意味の代わりにmaxを使用して、異なる濃度で現在 2 つの蛋白質種の複合体の最大数が制限されている考慮、種は低い数値で提示します。
相関数と明るさは、瞬間の分析に基づいてサンプルでは、固定位置で時間をかけて得た画像のスタックの各ピクセルの蛍光強度通常 100 〜 200 から成るフレーム、スペクトルの 2 つのチャンネル (g = グリーン チャンネル、r = 赤のチャネル)。時間平均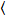 私
私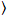 私と分散
私と分散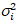 、各ピクセルのスペクトル チャネルで分子明るさ εiと番号の私n の計算 (私= g、 r)18。
、各ピクセルのスペクトル チャネルで分子明るさ εiと番号の私n の計算 (私= g、 r)18。
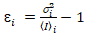 、(10)
、(10)
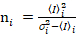 .(11)
.(11)
真フォトンカウンティング検出器の理想的な場合に与えられた方程式が適用されることに注意してくださいすることが重要です。アナログ検出システムでは、次の方程式は19,20を適用されます。
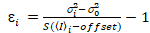 、(12)
、(12)
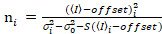 . (13)
. (13)
ここで、S は検出された光子と記録されたデジタル カウントの変換係数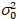 読み出しノイズは、オフセット検出器強度オフセットを参照します。一般に、これらの量は、安定した照明19、例えば、反射金属表面または乾燥色素溶液の強度の関数として検出器分散を測定に基づいて、任意の検出器型の校正する必要があります。オフセットは、励起光のないサンプルの計数率を測定することによって決定できます。検出器関連する差異の線形回帰を実行することによって
読み出しノイズは、オフセット検出器強度オフセットを参照します。一般に、これらの量は、安定した照明19、例えば、反射金属表面または乾燥色素溶液の強度の関数として検出器分散を測定に基づいて、任意の検出器型の校正する必要があります。オフセットは、励起光のないサンプルの計数率を測定することによって決定できます。検出器関連する差異の線形回帰を実行することによって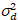 対強度 (私) プロット、S と
対強度 (私) プロット、S と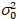 19決定をすることができます。
19決定をすることができます。
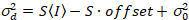 . (14)
. (14)
最後に、相互相関明るさは、各ピクセルの計算し、21として一般的に定義されます。
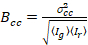 、(15)
、(15)
どこ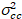 クロス分散です
クロス分散です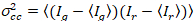 。
。
長寿命の変動をフィルターするためにすべての ccN & B 計算次のフィルタ リングされていない、独立して各ピクセル22有蓋車されます。簡単に言えば、 niε は私(私= g、 r) Bccは 8-15 フレームなどのセグメントをスライド式で計算されます。こうして得られた値を平均して最終的なピクセルの数と明るさの値を取得できます。
化学量論の分析
細胞間の接触でタンパク質複合体の化学量論組成を推定するために、sFCCS や ccN & B データの各スペクトル チャネルで分子の明るさを別々 に分析できます。SFCCS、各チャネルの測定あたり 1 つの輝度値を取得します。CcN & B は、細胞間の接触に対応するすべてのピクセルの輝度のヒストグラムが得られるし、測定の代表的な明るさとして平均 (または中央値) の値を使用できます。単量体の参照について同じ分析を実行する直接検出された蛋白質複合体の平均のオリゴマーの状態を取得するすべての明るさの値を正規化することができます。この時点で、オリゴマーの状態を過小評価可能性があります非蛍光性 FPs の存在を修正することが重要です。これは通常 1 色 sFCS または番号を使用して参照のホモ二量体蛋白質23,24の明るさと明るさ (N & B) を測定することによって実行されます。
Protocol
Representative Results
Discussion
ここで説明した実験手順により、蛋白質蛋白質の調査は細胞間の接触、蛍光揺らぎ分光技術、すなわち sFCCS、ccN & B でトランス相互作用これらのメソッドは、それぞれ 1 つまたは他の融合蛋白質を表現する 2 つの隣接セルとの接触で興味の蛋白融合した 2 つのスペクトル分離 FPs によって放射される蛍光変動の統計的分析を含みます。トランス錯体の存在は、近隣の PMs のタンパ?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
この作品は、ドイツ研究振興協会 (DFG) によってを部分的に支持された 254850309 を付与します。著者は、論文の批判的な読みの Madlen リュクネールをありがとうございます。
Materials
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
References
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -. M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, 273 (2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Biologie du développement. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -. Y., Mok, L. -. P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634 (2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- . Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010)
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494 (2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073 (2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487 (2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412 (2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545 (2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).

