En fluorescens svingninger spektroskopi analysen av Protein-Protein interaksjoner i celle-celle kontakter
Summary
Denne protokollen beskriver en fluorescens svingninger spektroskopi tilnærming for å undersøke interaksjon mellom proteiner formidling celle-celle interaksjoner, dvs proteiner lokalisert i cellen veikryss, direkte i levende celler. Vi gir detaljerte veiledninger på instrumentet kalibrering, datainnsamling og analyse, inkludert rettelser til mulig gjenstanden kilder.
Abstract
En rekke biologiske prosesser innebærer celle-celle interaksjoner, vanligvis formidlet av proteiner som samhandler på grensesnittet mellom nabokommunene cellene. Rundt er bare få analyser dugelig av spesielt sondering slik interaksjon direkte i levende celler. Her presenterer vi en analyse for å måle binding av proteiner uttrykt på overflater av nabokommunene cellene på celle-celle kontakter. Denne analysen består av to trinn: blanding av celler som uttrykker proteiner rundt smeltet forskjellige fluorescerende proteiner, etterfulgt av fluorescens svingninger spektroskopi målinger på celle-celle kontakter ved hjelp av en AC confocal laserskanning mikroskop. Vi viser muligheten for denne analysen i en biologisk relevante kontekst ved å måle samspillet av amyloid forløper som protein 1 (APLP1) over celle-celle veikryss. Vi gir detaljert protokoller på datainnsamling bruker fluorescens-baserte teknikker (skanning fluorescens kryss-korrelasjon spektroskopi, cross-korrelasjon tall og lysstyrke analyse) og nødvendige instrument kalibreringer. Videre, vi diskutere avgjørende skritt i dataanalyse og hvordan å identifisere og korrigere eksterne, falske signal variasjoner, for eksempel på grunn av photobleaching eller celle bevegelse.
Vanligvis presentert analysen gjelder homo – eller heterotypic protein-protein interaksjon på celle-celle kontakter mellom celler av samme eller forskjellige typer og kan implementeres på en kommersiell AC confocal laserskanning mikroskop. En viktig forutsetning er stabiliteten i systemet, som må være tilstrekkelig å undersøke diffusive dynamikken i proteiner rundt over flere minutter.
Introduction
Mange biologiske prosesser oppstå ved celle-celle interaksjoner, f.eks celle-celle adhesjon1,2,3, celle-celle fusion4 og mobilnettet anerkjennelse5. Slike hendelser er spesielt viktig i utviklingen av multicellular organismer og for celle-celle kommunikasjon, f.eks under immunreaksjoner. Disse prosessene er vanligvis formidlet av proteiner som er lokalisert på overflaten, dvs. i plasma membranen (PM) til nabokommunene cellene og gjennomgå spesifikke interaksjoner ved celle-celle kontakt som er nøyaktig regulert i rom og tid. I mange tilfeller kan disse interaksjoner er direkte homo- eller heterotypic protein-protein trans interaksjoner, men kan også innebære ioner eller ligander som ekstracellulære linkers1. Selv om du er av grunnleggende betydning, er det mangel på analyser sondering disse bestemt protein-protein interaksjoner direkte i sine opprinnelige omgivelser av levende celler. Mange metoder krever enten celle avbrudd (f.eks biokjemiske analyser som co-immunoprecipitation6), fiksering (f.eks noen super-oppløsning mikroskopi optisk teknikker og elektronmikroskop av celle-celle kontakter7), eller er ikke-spesifikk, f.eks aggregering / vedheft søk8,9. For å overvinne problemet, er fluorescens teknikker gjennomført basert på fluorescens resonans energi overføring (bånd)10 eller fluorescens complementation11. Men for å oppnå tilstrekkelig små avstander mellom fluorophores, krever disse metodene fluorescerende etiketter på ekstracellulære side av proteiner10, potensielt forstyrre trans interaksjoner.
Her presenterer vi en alternativ fluorescens-baserte analysen for protein-protein interaksjoner i celle-celle kontakter. Denne tilnærmingen kombinerer fluorescens kryss-korrelasjon tilnærminger (skanning fluorescens kryss-korrelasjon spektroskopi (sFCCS), cross-korrelasjon nummer og lysstyrke (ccN og B)) og blanding av celler som uttrykker en fusion konstruksjon av protein av interesse, f.eks en vedheft reseptor. Undersøkte receptors i de to samspill cellene er merket med to spectrally atskilt fluorescerende proteiner (FPs), fra den intracellulær side (se figur 1A).
De næringsdrivende metodene er basert på den statistiske analysen fluorescens svingninger forårsaket av diffusive bevegelse fluorescerende fusion proteiner gjennom fokal volumet av en AC confocal laserskanning mikroskop. Mer i detalj sonder analysen co spredningen av proteiner interesse både nabolandet PMs på celle-celle kontakter. Hvis proteiner gjennomgå trans interaksjoner, vil disse trans komplekser bære fluorescerende proteiner emitting i begge spectral kanalene, forårsaker korrelert fluorescens svingninger av både emittere. Derimot, hvis ingen binding, vil antall svingninger proteiner i overfor PMs være uavhengig, forårsaker ingen samsvarende svingninger. Oppkjøpet kan utføres på to måter: 1) sFCCS er basert på en linje-formet skanning over celle-celle kontakten og effektivt sonder samhandlinger i et sted i regionen kontakt. Gjennom en timelig analyse av fluorescens svingninger gir sFCCS også dynamics informasjon, dvs. diffusjon koeffisientene av protein komplekser; 2) ccN & B er basert på en pixel-wise analyse av en sekvens av bilder kjøpt til celle-celle kontakt regionene. Den har evnen å undersøke og kart interaksjoner langs hele kontakt regionen (i en fokalplanet), men gir ikke informasjon på dynamics. Begge metodene kan kombineres med en analyse av molekylære lysstyrken, dvs. gjennomsnittlig fluorescens signalet slippes ut i tidsenheten av enkelt spre protein komplekser, og dermed gir anslag over støkiometri av protein komplekser på celle-celle kontakter.
I denne artikkelen gir vi detaljert protokoller for eksempel forberedelse, instrument kalibrering, datainnsamling og analyse å utføre presentert analysen på en kommersiell AC confocal laserskanning mikroskop. Eksperimenter kan utføres på instrument Foton teller eller analoge detektorer og et mål med høy numeriske blenderåpning. Vi videre diskutere avgjørende skritt av protokollen og gi korreksjon ordninger for flere prosesser forårsaker artefactual signal svingninger, f.eks detektor støy, photobleaching eller celle bevegelse. Opprinnelig utviklet for å undersøke interaksjoner mellom tilhenger celler, analysen kan endres for suspensjon celler eller tilpasset modell membran systemer, f.eks gigantiske unilamellar blemmer (GUVs) eller gigantiske plasma membran blemmer (GPMVs), slik at den kvantifisering av interaksjoner i forskjellige lipid miljøer eller i fravær av en organisert cytoskjelett12,13.
Skanning fluorescens kryss-korrelasjon spektroskopi er en modifisert versjon av fluorescens kryss-korrelasjon spektroskopi14 og var spesielt beregnet på sonde langsom diffusive dynamikk i lipid membraner15. Den er basert på en linje skanning oppkjøpet vinkelrett til PM med fluorescerende proteiner av interesse. For å undersøke interaksjoner av to annerledes merket protein arter, utføres oppkjøpet i to spectral kanaler med to laser linjer og to oppdagelsen Vinduer for spectrally atskilt fluorophores. På grunn av treg diffusjon dynamikken i proteiner i PM (D≤ ~ 1 µm2/s), en cross-talk-fri måling kan utføres av vekslende eksitasjon ordningen fra linje til linje15. Analysen starter med: 1) en justering algoritme korrigere for sideveis celle bevegelse basert på block-wise gjennomsnitt ~ 1000 linjer, 2) fastsettelse av posisjon med maksimal fluorescens signaldvs. PM posisjon, i hver blokk og 3) skiftende Alle blokkene til en felles opprinnelse12,15, separat i hver enkelt kanal. Deretter utføres en automatisk valg av bildepunkter som tilsvarer PM ved å velge den sentrale regionen et Gaussian anfall av summen av alle justerte linjer (dvs. center ± 2.5σ). Integrering av signalet i hver linje gir membran fluorescens tiden serien f (t) i hver enkelt kanal (g = grønn kanal, r = rød sone). Merk at pikselstørrelsen må være små nok, f.eks < 200 nm, å rekonstruere form av punkt spredning funksjon og finne midten, tilsvarer plasseringen av PM. I nærvær av betydelig photobleaching, fluorescens tiden serien i hver kanal kan være modellert med en dobbel-eksponentiell funksjon og deretter korrigert med følgende formel:16
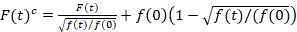 . (1)
. (1)
Det er viktig å merke seg at denne formelen effektivt korrigerer både amplitudes og diffusion times fra korrelasjon analyse av f (t)c, sammenlignet med parameteren anslår at skulle hentes fra det ukorrigert f (t). Deretter auto – og kryss-korrelasjon funksjonene (ACFs / CCFs) av fluorescensen signaler beregnes:
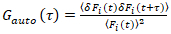 , (2).
, (2).
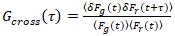 , (3).
, (3).
hvor sesFjeg = Fjeg(t) – 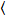 Fjeg(t)
Fjeg(t)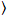 og jeg = g, r.
og jeg = g, r.
En todimensjonal diffusjon modell er da utstyrt til alle korrelasjon funksjoner (CFs):
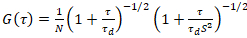 . (4)
. (4)
Her, angir N antall fluorescerende proteiner i observasjon volumet og τd diffusjon tiden for hver kanal. Denne modellen tar hensyn at beskrevet eksperimentelle innstillingen, spredning av proteiner i PM oppstår i x-z flyet, i motsetning til vanlige konfigurasjonen fluorescens korrelasjon spektroskopi (FCS) eksperimenter på membraner undersøkelser diffusjon i x-y flyet AC confocal volum17. Livet w0 og struktur faktoren S, som beskriver forlengelse wz fokal volumroten i z, S = wz/w0, hentes fra et punkt FCS kalibrering mål med spectrally lignende fargestoffer og optisk innstillingene bruke allerede tilgjengelig for diffusion rthe Dfargestoff:
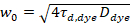 , (5).
, (5).
hvor τd, fargestoff er målt gjennomsnittlig diffusjon tiden av fargestoff molekyler, fra montering en modell for tredimensjonale diffusjon til dataene, tar konto overganger av en brøkdel T alle N molekyler til en trilling stat med en gang konstant ττ:
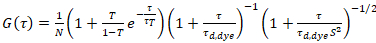 . (6)
. (6)
Til slutt, diffusjon koeffisientene (D), molekylær lysstyrkeverdier (ε) og relativ kryss-korrelasjonen sFCCS data (rel.cc.) er beregnet som følger:
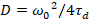 , (7).
, (7).
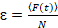 , (8).
, (8).
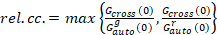 , (9).
, (9).
hvor Gcross(0) er amplituden til funksjonen kryss-korrelasjon og 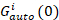 er amplituden til funksjonen autokorrelasjon i jeg-th kanalen.
er amplituden til funksjonen autokorrelasjon i jeg-th kanalen.
Denne definisjonen av relativ kryss-korrelasjon, dvs med max i stedet for betyr i ligningen 9, tar hensyn til at maksimalt antall komplekser av to protein arter til stede i ulike konsentrasjoner er begrenset av den arter til stede i et lavere tall.
Cross-korrelasjon tall og lysstyrke er basert på litt analyse av fluorescens intensiteten for hvert bildepunkt i en bildestakk kjøpt over tid til en fast stilling i utvalget, vanligvis bestående av ~ 100-200 rammene, med to spectral kanaler () g = grønn kanal, r = rød sone). Fra timelige middelverdien 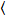 jeg
jeg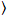 jeg og varians
jeg og varians 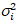 , den molekylære lysstyrke εjeg og nummer njeg beregnes i hver piksel og spectral kanal (jeg = g, r)18:
, den molekylære lysstyrke εjeg og nummer njeg beregnes i hver piksel og spectral kanal (jeg = g, r)18:
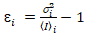 , (10).
, (10).
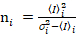 . (11)
. (11)
Det er viktig å merke seg at gitt ligningene gjelder det ideelle tilfellet av en sann Foton-telling detektor. For analoge oppdagingssystemer gjelder følgende ligningene19,20:
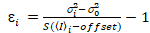 , (12).
, (12).
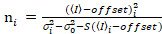 . (13)
. (13)
Her, S er omregningsfaktoren mellom oppdaget fotoner og innspilte digitalt teller, 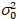 er avlesning støy og forskyvning refererer til detektor intensitet forskyvningen. Vanligvis bør disse antallene kalibreres, for alle merker, basert på måling detektor avviket som en funksjon av intensitet for jevn belysning19, f.eks en reflekterende metalloverflate eller tørket fargestoff løsning. Forskyvning kan bestemmes ved å måle antall prisen for et utvalg uten eksitasjon lys. Ved å utføre en lineær regresjon av detektor-assosiert variansen
er avlesning støy og forskyvning refererer til detektor intensitet forskyvningen. Vanligvis bør disse antallene kalibreres, for alle merker, basert på måling detektor avviket som en funksjon av intensitet for jevn belysning19, f.eks en reflekterende metalloverflate eller tørket fargestoff løsning. Forskyvning kan bestemmes ved å måle antall prisen for et utvalg uten eksitasjon lys. Ved å utføre en lineær regresjon av detektor-assosiert variansen 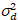 versus intensitet (jeg) plot, S og
versus intensitet (jeg) plot, S og 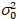 kan være bestemt19:
kan være bestemt19:
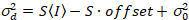 . (14)
. (14)
Til slutt, cross-korrelasjon lysstyrken beregnes i hver piksel og defineres generelt som21
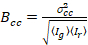 , (15).
, (15).
hvor 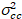 er kryss-variansen
er kryss-variansen 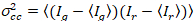 .
.
For å filtrere varige svingninger, utføres alle ccN & B beregninger etter en boxcar filtrering, uavhengig for hver piksel22. Kort, njeg, εjeg (jeg = g, r) og Bcc beregnes i skyve segmenter av f.eks 8-15 rammer. Verdiene anskaffes kan være så gjennomsnitt for å få den endelige pixel tall og lysstyrke.
Støkiometri analyse
For å beregne støkiometri av protein komplekser på celle-celle kontakter, kan molekylær lysstyrken bli separat analysert i hver spectral kanal for sFCCS eller ccN & B data. I sFCCS hentes en lysstyrkeverdi per måling i hver enkelt kanal. I ccN & B, et lysstyrke histogram for alle bildepunkter ved celle-celle kontakten er innhentet og gjennomsnittlig (eller median) verdien kan brukes som representant lysstyrke for målingen. Ved å utføre den samme analysen på en monomerisk referanse, kan alle lysstyrkeverdier normaliseres for å få direkte gjennomsnittlig oligomeric staten oppdaget protein komplekser. På dette punktet, er det viktig å korrigere for tilstedeværelsen av ikke-fluorescerende FPs som kan resultere i en undervurdering av oligomeric. Dette er vanligvis utføres av måle lysstyrken på en homo-dimeric referanse protein23,24 med én farge sFCS eller tall og lysstyrke (N & B).
Protocol
Representative Results
Discussion
Den eksperimentelle prosedyren beskrevet her kan etterforskningen av protein-protein trans samhandlinger på celle-celle kontakter, ansette fluorescens svingninger spektroskopi teknikker, nemlig sFCCS og ccN & B. Disse metodene omfatter en statistisk analyse av fluorescens svingninger slippes ut av to spectrally atskilt FPs smeltet sammen til protein(s) rundt på en kontakt med to nabokommunene cellene hver uttrykke en eller andre fusion protein. Tilstedeværelsen av trans komplekser er kvantifisert ved…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Dette arbeidet ble delvis støttet av Deutsche Forschungsgemeinschaft (DFG) gi 254850309. Forfatterne takker Madlen Luckner for kritisk lesning av manuskriptet.
Materials
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
References
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -. M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, 273 (2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Biologie du développement. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -. Y., Mok, L. -. P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634 (2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- . Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010)
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494 (2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073 (2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487 (2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412 (2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545 (2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).

