En fluorescens fluktuation spektroskopi haltbestämning av Protein-Protein interaktioner på Cell-Cell kontakter
Summary
Det här protokollet beskriver ett fluorescens fluktuation spektroskopi-baserat tillvägagångssätt för att undersöka interaktioner mellan proteiner medla cell-cell interaktioner, dvs proteiner lokaliserade i cell korsningar, direkt i levande celler. Vi tillhandahåller detaljerade riktlinjer på instrumentkalibrering, datainsamling och analys, inklusive korrigeringar till möjlig artefakt källor.
Abstract
En mängd biologiska processer innebär cell-cell interaktioner, typiskt medierad av proteiner som samverkar i gränssnittet mellan angränsande celler. Av intresse klarar bara några analyser av specifikt sondera sådana interaktioner direkt i levande celler. Här presenterar vi ett test för att mäta bindningen av proteiner som uttrycks på ytbehandlar av närliggande celler, på cell-cell kontakter. Denna analys består av två steg: blandning av celler som uttrycker proteinerna av intresse smält till olika fluorescerande proteiner, följt av fluorescens fluktuation spektroskopi mätningar vid cell-cell kontakter med hjälp av en confocal laser skanning Mikroskop. Vi demonstrera genomförbarheten av denna analys i ett biologiskt relevanta sammanhang genom att mäta interaktioner av proteinet amyloid prekursor-liknande 1 (APLP1) över cell cell korsningar. Vi tillhandahåller detaljerade protokoll på dataförvärvet med fluorescens-baserade tekniker (Skanna fluorescens cross-korrelation spektroskopi, cross-korrelation nummer och ljusstyrka analys) och krävs instrument kalibreringar. Vidare diskuterar vi kritiska steg i dataanalys och hur att identifiera och korrigera externa, falska signal variationer, exempelvis på grund av fotoblekning eller cell rörelse.
I allmänhet presenterade analysen är tillämplig på alla homo- eller heterotypic protein-protein interaktioner på cell-cell kontakter, mellan celler av samma eller olika typer och kan genomföras på en kommersiell confocal laserscanning mikroskopet. Ett viktigt krav är stabiliteten i systemet som måste vara tillräckligt sond diffus dynamics av proteinerna av intresse under flera minuter.
Introduction
Många biologiska processer inträffar på platser av cell-cell interaktioner, t.ex., cell-cell adhesion1,2,3, cell-cell fusion4 och cellulära erkännande5. Sådana händelser är särskilt viktigt under utvecklingen av flercelliga organismer och för cell-cell kommunikation, exempelvis under immunsvar. Dessa processer är typiskt medierad av proteiner som är lokaliserade på ytan, dvs på plasmamembranet (PM) på angränsande celler och genomgå särskilda interaktioner i cell cell kontakt som är tydligt reglerad i utrymme och tid. I många fall, dessa interaktioner är direkt homo- eller heterotypic protein-protein trans interaktioner, men kan också innebära joner eller ligander egenskap extracellulära linkers1. Även om det är av grundläggande betydelse, finns det brist på analyser sondera dessa specifika protein-protein interaktioner direkt i den ursprungliga miljön i levande celler. Många metoder antingen kräva cell störningar (t.ex. biokemiska analyser såsom co-immunoprecipitation6), fixering (t.ex. vissa super-upplösning optisk mikroskopi tekniker och elektronmikroskopi av cell-cell kontakter7), eller är icke-specifika, t.ex. aggregering / vidhäftning analyser8,9. För att lösa problemet, har fluorescens tekniker genomförts utifrån fluorescens resonans energi överföring (bandet)10 eller fluorescens komplettering11. Men för att uppnå tillräckligt små avstånd mellan fluorophores, kräver dessa metoder fluorescerande etiketterna på den extracellulära sidan av proteiner10, potentiellt störa trans interaktioner.
Här presenterar vi alternativa fluorescens-baserat test för protein-protein interaktioner på cell-cell kontakter. Denna strategi kombinerar fluorescens cross-korrelation strategier (skanning fluorescens cross-korrelation spektroskopi (sFCCS), cross-korrelation nummer och ljusstyrka (ccN och B)) och blandning av celler som uttrycker en fusion konstruktion av proteinet av intresse, t.ex. en vidhäftning-receptorn. De undersökta receptorerna i två samverkande cellerna är märkta med två spektralt separerade fluorescerande proteiner (FPs), från den intracellulära (se figur 1A).
De sysselsatta metoderna baseras på den statistiska analysen av fluorescens fluktuationer inducerad av diffus rörelse av fluorescerande fusionsproteinerna genom fokal volymen av en confocal laser skanning Mikroskop. Mer i detalj sonder analysens samtidig diffusionen av proteinerna av intresse för båda angränsande PMs på cell-cell kontakter. Om proteinerna genomgå trans interaktioner, kommer dessa trans komplex bära fluorescerande proteiner avger i båda spektrala kanaler, orsakar korrelerade fluorescens fluktuationer i både utsläppsländerna. Däremot, om ingen bindning uppstår, blir de antal svängningarna av proteiner i inför PMs oberoende, orsakar inga korrelerade fluktuationer. Förvärv kan utföras på två sätt: 1) sFCCS är baserat på en rad-formade scan hela cell cell kontakten och effektivt sonder samspelet i en plats som ligger i regionen kontakt. Genom en temporal analys av fluorescens svängningar ger sFCCS också dynamics information, dvs Diffusionskoefficienterna av proteinkomplex; (2) ccN & B bygger på en pixel-wise analys av en sekvens av bilder som förvärvats på cell-cell kontakt regionerna. Den har förmåga att söka och karta interaktioner längs hela kontakta region (i ett fokalplan), men ger inte information om dynamics. Båda metoderna kan kombineras med en analys av molekylär ljusstyrka, dvs genomsnittliga fluorescens signal som avges i tidsenheten av enda diffuserande proteinkomplex och, således ge uppskattningar av stökiometri av proteinkomplex på cell-cell kontakter.
I denna artikel tillhandahåller vi detaljerade protokoll för provberedning, instrumentkalibrering, datainsamling och analys att utföra presenterade analysen på en kommersiell confocal laserscanning mikroskopet. Experimenten kan utföras på något instrument utrustat med photon counting eller analoga detektorer och ett mål med hög numeriska bländaröppningen. Vi har ytterligare diskutera kritiska steg i protokollet och ger korrigering system för flera processer som orsakar tingsliga signal fluktuationer, exempelvis detektor buller, fotoblekning eller cell rörelse. Utvecklades ursprungligen för att undersöka samspelet mellan vidhäftande celler, analysen kan ändras för suspension celler eller anpassad till modellen membransystem, t.ex. giant unilamellar blåsor (GUVs) eller giant plasma membranet blåsor (GPMVs), så att den kvantifiering av interaktioner i olika lipid miljöer eller i avsaknad av en organiserad cytoskelettet12,13.
Scanning fluorescens cross-korrelation spektroskopi är en modifierad version av fluorescens cross-korrelation spektroskopi14 och utformades sond långsam diffus dynamics i lipid membran15. Den är baserad på en linje scan förvärv vinkelrätt till PM som innehåller de fluorescerande proteinerna av intresse. För att undersöka samspelet mellan två olika märkta proteinet arter, utförs förvärvet i två spektrala kanaler med två laserlinjer och två upptäckt windows för spektralt separerade fluorophores. På grund av långsam diffusion dynamiken av proteiner i PM (D≤ ~ 1 µm2per sekund), en cross-talk-gratis mätning kan utföras av omväxlande excitation systemet från linje till linje15. Analysen börjar med: 1) en justering algoritm korrigera för laterala cell rörelse utifrån block-wise genomsnitt ~ 1000 rader, 2) bestämning av placerar med maximal fluorescens signaldvs PM position, i varje block och 3) skiftande av alla block till ett gemensamt ursprung12,15, separat i varje kanal. Ett automatiskt val av pixlar motsvarar PM utförs sedan genom att välja regionen centrala från en Gaussisk passform av summan av alla anpassade linjer (dvs center ± 2.5σ). Integration av signalera i varje rad ger membran fluorescens tidsserierna F(t) i varje kanal (g = grön kanal, r = röda kanalen). Observera att pixelstorlek måste vara liten nog, t.ex. < 200 nm, att rekonstruera formen på punkten sprida funktion och hitta dess centrerar, motsvarar positionen för PM. I närvaro av betydande fotoblekning, fluorescens tidsserierna i varje kanal kan modelleras med en dubbel-exponentialfunktionen och sedan korrigeras med följande formel:16
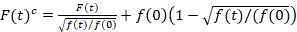 . (1)
. (1)
Det är viktigt att notera att denna formel effektivt korrigerar både amplituder och diffusion gånger erhållits från korrelation analys av F(t)c, jämfört med parametern uppskattningar som skulle erhållas från den okorrigerade F(t). Sedan funktionerna auto – och cross-korrelation (ACFs / CCFs) av fluorescensen signaler beräknas:
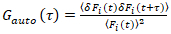 , (2).
, (2).
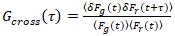 , (3).
, (3).
där δFjag = Fjag(t) – 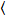 Fjag(t)
Fjag(t)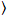 och jag = g, r.
och jag = g, r.
En tvådimensionell diffusion modell monteras sedan alla korrelation funktioner (CFs):
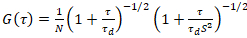 . (4)
. (4)
Här, betecknar N antalet fluorescerande proteiner i observation volym och τd diffusion tiden för varje kanal. Denna modell tar hänsyn att beskrivs i experimentell inställningen diffusion av proteiner i PM uppstår i x-z planet, i motsats till den vanliga konfigurationen av fluorescens korrelation spektroskopi (FCS) experiment på membran sondering diffusion i x-y-planet av confocal volym17. Midjan w0 och struktur faktor S, som beskriver töjning wz av fokal volymen i z, S = wz/w0, erhålls från en punkt FCS kalibrering mätning utförs med spektralt liknande färgämnen och samma optiska inställningar använder redan tillgängliga värden för den diffusionskoefficienten Dfärgämne:
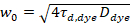 , (5).
, (5).
där τd, färgämne är den uppmätta genomsnittliga diffusion tid av färgämne molekylerna, erhållits från passande modell för tredimensionellt diffusion till data, med hänsyn till konto övergångar av en bråkdel T alla N molekyler till en triplett stat med en tidskonstant ττ:
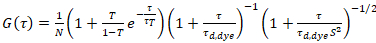 . (6)
. (6)
Slutligen beräknas Diffusionskoefficienterna (D), molekylär ljusstyrkevärden (ε) och den relativa cross-korrelationen av sFCCS data (rel.cc.) enligt följande:
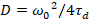 , (7).
, (7).
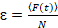 , (8).
, (8).
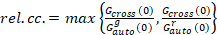 , (9).
, (9).
där Gcross(0) är amplituden av funktionen cross-korrelation och 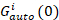 är amplituden av autokorrelation funktion i jag-th kanal.
är amplituden av autokorrelation funktion i jag-th kanal.
Denna definition av den relativa cross-korrelationen, dvs med max istället för menar i ekvation 9, tar hänsyn till att det maximala antalet komplex av två protein arter vid olika koncentrationer begränsas av den arter som finns i ett lägre nummer.
Cross-korrelation nummer och ljusstyrka är baserad på en stund analys av fluorescensintensiteten för varje pixel i en bildstapel förvärvade med tiden till en fast position i urvalet, vanligtvis bestående av ~ 100-200 ramar, med två spektrala kanaler () g = grön kanal, r = röda kanalen). Från temporal medelvärdet 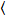 jag
jag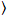 jag och variansen
jag och variansen 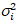 , de molekylära ljusstyrka εi och nummer njag beräknas i varje pixel och spektrala kanal (jag = g, r)18:
, de molekylära ljusstyrka εi och nummer njag beräknas i varje pixel och spektrala kanal (jag = g, r)18:
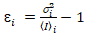 , (10).
, (10).
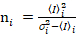 . (11)
. (11)
Det är viktigt att Observera att de givna ekvationerna gäller för det ideala fallet av en sann fotonräknande detektor. För analoga detektionssystem tillämpas följande ekvationer19,20:
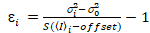 , (12).
, (12).
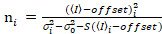 . (13)
. (13)
Här, S är konverteringsfaktorn mellan upptäckta fotoner och inspelade digitala räkningarna, 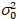 är utslaget buller och offset hänvisar till detektorn intensitet offset. Generellt, dessa kvantiteter bör kalibreras, för någon Detektortyp, baserat på mätning detektor variansen som en funktion av intensitet för jämn belysning19, t.ex. en reflekterande metallyta eller torkade dye lösning. Förskjutning kan bestämmas genom mätning räkna räntan för ett prov utan excitation ljus. Genom att utföra en linjär regression av detektor-associerade variansen
är utslaget buller och offset hänvisar till detektorn intensitet offset. Generellt, dessa kvantiteter bör kalibreras, för någon Detektortyp, baserat på mätning detektor variansen som en funktion av intensitet för jämn belysning19, t.ex. en reflekterande metallyta eller torkade dye lösning. Förskjutning kan bestämmas genom mätning räkna räntan för ett prov utan excitation ljus. Genom att utföra en linjär regression av detektor-associerade variansen 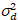 kontra intensitet (jag) tomt, S och
kontra intensitet (jag) tomt, S och 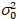 kan vara beslutsamt19:
kan vara beslutsamt19:
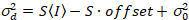 . (14)
. (14)
Slutligen, cross-korrelation ljusstyrkan beräknas i varje pixel och definieras i allmänhet som21
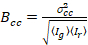 , (15).
, (15).
där 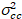 är cross-variansen
är cross-variansen 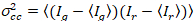 .
.
För att filtrera långlivat fluktuationer, utförs alla ccN & B beräkningar efter en boxcar filtrering, självständigt för varje pixel22. Kort, ni, εjag (jag = g, r) och Bcc beräknas i glidande segment av t.ex., 8-15 bildrutor. Erhållits på detta sätt kan vara då medelvärdet för att få den slutliga pixeln nummer och ljusstyrka värden.
Stökiometri analys
För att uppskatta stökiometri av proteinkomplex på cell-cell kontakter, kan molekylär ljusstyrkan analyseras separat för varje spektrala kanal för sFCCS eller ccN & B data. I sFCCS erhålls ett intensitetsvärde per mätning i varje kanal. I ccN & B, ett ljusstyrka histogram över alla pixlar motsvarar den cell-cell kontakten erhålls och värdet genomsnitt (eller median) kan användas som representativa ljusstyrka för mätning. Genom att utföra samma analys på en monomer referens, kan alla ljusstyrkevärden normaliseras för att direkt få den genomsnittliga Oligomera delstaten de upptäckta proteinkomplex. Vid denna punkt, är det viktigt att korrigera för förekomst av icke-fluorescerande FPs som kan leda till en underskattning av Oligomera staten. Detta görs vanligtvis genom att mäta ljusstyrka en homo-dimeriskt referens protein23,24 använder en färg sFCS eller nummer och ljusstyrka (N & B).
Protocol
Representative Results
Discussion
Den experimentella metod som beskrivs här kan utredningen av protein-protein trans interaktioner på cell-cell kontakter, anställa fluorescens fluktuation spektroskopi tekniker, nämligen sFCCS och ccN & B. Dessa metoder innebär en statistisk analys av fluorescens fluktuationer som avges av två spektralt separerade FPs smält till proteinernas sevärdheter på en kontakt med två angränsande celler, var och en uttrycker en eller den andra fusionsprotein. Förekomsten av trans komplex kvantifieras i…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Detta arbete var delvis stöds av Deutsche Forschungsgemeinschafts (DFG) bevilja 254850309. Författarna vill tacka Madlen Luckner för kritisk läsning av manuskriptet.
Materials
| DMEM growth medium | PAN-Biotech | P04-01548 | |
| DPBS w/o: Ca2+ and Mg2+ | PAN-Biotech | P04-36500 | |
| DPBS w: Ca2+ and Mg2+ | PAN-Biotech | P04-35500 | |
| Trypsin EDTA | PAN-Biotech | P10-023100 | |
| TurboFect Transfection Reagent | Thermo Fisher Scientific | R0531 | |
| HEK 293T cells | DSMZ | ACC 635 | |
| Alexa Fluor 488 NHS Ester | Thermo Fisher Scientific | A20000 | |
| Rhodamine B | Sigma-Alderich | 83689-1G | |
| Plasmid DNA | Addgene | NA | See reference 12 (Dunsing et. al., MBoC 2017),for a detailed description of all plasmids |
| 6-well plate | Starlab | CC7672-7506 | |
| 35-mm glass bottom dishes | CellVis | D35-14-1.5-N | |
| Zeiss LSM780 confocal | Carl Zeiss | NA | |
| MATLAB software package | MathWorks | 2015b | |
| Neubauer cell counting chamber | Marienfeld | 640110 |
References
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (2002).
- Tepass, U., Truong, K., Godt, D., Ikura, M., Peifer, M. Cadherins in embryonic and neural morphogenesis. Nature Reviews Molecular Cell Biology. 1 (2), 91-100 (2000).
- Harris, T. J. C., Tepass, U. Adherens junctions: from molecules to morphogenesis. Nature Reviews Molecular Cell Biology. 11 (7), 502-514 (2010).
- Hernández, J. M., Podbilewicz, B. The hallmarks of cell-cell fusion. Development. 144 (24), 4481-4495 (2017).
- Huppa, J. B., Davis, M. M. T-cell-antigen recognition and the immunological synapse. Nature Reviews Immunology. 3 (12), 973-983 (2003).
- Kaden, D., Voigt, P., Munter, L. -. M., Bobowski, K. D., Schaefer, M., Multhaup, G. Subcellular localization and dimerization of APLP1 are strikingly different from APP and APLP2. Journal of cell science. 122, 368-377 (2009).
- Yap, A. S., Michael, M., Parton, R. G. Seeing and believing: recent advances in imaging cell-cell interactions. F1000Research. 4, 273 (2015).
- Kashef, J., Franz, C. M. Quantitative methods for analyzing cell-cell adhesion in development. Biologie du développement. 401 (1), 165-174 (2015).
- Soba, P., et al. Homo- and heterodimerization of APP family members promotes intercellular adhesion. The EMBO Journal. 24 (20), 3624-3634 (2005).
- Kim, S. A., Tai, C. -. Y., Mok, L. -. P., Mosser, E. A., Schuman, E. M. Calcium-dependent dynamics of cadherin interactions at cell-cell junctions. Proceedings of the National Academy of Sciences of the United States of America. 108 (24), 9857-9862 (2011).
- Feinberg, E. H., et al. GFP Reconstitution Across Synaptic Partners (GRASP) Defines Cell Contacts and Synapses in Living Nervous Systems. Neuron. 57 (3), 353-363 (2008).
- Dunsing, V., Mayer, M., Liebsch, F., Multhaup, G., Chiantia, S. Direct evidence of amyloid precursor-like protein 1 trans interactions in cell-cell adhesion platforms investigated via fluorescence fluctuation spectroscopy. Molecular biology of the cell. 28 (25), 3609-3620 (2017).
- Schneider, F., et al. Diffusion of lipids and GPI-anchored proteins in actin-free plasma membrane vesicles measured by STED-FCS. Molecular Biology of the Cell. 28 (11), 1507-1518 (2017).
- Bacia, K., Kim, S. A., Schwille, P. Fluorescence cross-correlation spectroscopy in living cells. Nature methods. 3 (2), 83-89 (2006).
- Ries, J., Schwille, P. Studying Slow Membrane Dynamics with Continuous Wave Scanning Fluorescence Correlation Spectroscopy. Biophysical Journal. 91 (5), 1915-1924 (2006).
- Ries, J., Chiantia, S., Schwille, P. Accurate Determination of Membrane Dynamics with Line-Scan FCS. Biophysical Journal. 96 (5), 1999-2008 (2009).
- Chiantia, S., Ries, J., Schwille, P. Fluorescence correlation spectroscopy in membrane structure elucidation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1788 (1), 225-233 (2009).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical journal. 94 (6), 2320-2332 (2008).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy research and technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of Molecular Concentration and Brightness from Fluorescence Fluctuation Data with an Electron Multiplied CCD Camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Digman, M. A., Wiseman, P. W., Choi, C., Horwitz, A. R., Gratton, E. Stoichiometry of molecular complexes at adhesions in living cells. Proceedings of the National Academy of Sciences of the United States of America. 106 (7), 2170-2175 (2009).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. The FASEB journal official publication of the Federation of American Societies for Experimental Biology. 25 (9), 2883-2897 (2011).
- Dunsing, V., Luckner, M., Zühlke, B., Petazzi, R. A., Herrmann, A., Chiantia, S. Optimal fluorescent protein tags for quantifying protein oligomerization in living cells. Scientific Reports. 8 (1), 10634 (2018).
- Chen, Y., Johnson, J., Macdonald, P., Wu, B., Mueller, J. D. Observing Protein Interactions and Their Stoichiometry in Living Cells by Brightness Analysis of Fluorescence Fluctuation Experiments. Methods in enzymology. 472, 345-363 (2010).
- . Absolute Diffusion Coefficients: Compilation of Reference Data for FCS Calibration Available from: https://www.picoquant.com/images/uploads/page/files/7353/appnote_diffusioncoeffients.pdf (2010)
- Foo, Y. H., Naredi-Rainer, N., Lamb, D. C., Ahmed, S., Wohland, T. Factors affecting the quantification of biomolecular interactions by fluorescence cross-correlation spectroscopy. Biophysical journal. 102 (5), 1174-1183 (2012).
- Baum, M., Erdel, F., Wachsmuth, M., Rippe, K. Retrieving the intracellular topology from multi-scale protein mobility mapping in living cells. Nature Communications. 5, 4494 (2014).
- Wohland, T., Rigler, R., Vogel, H. The standard deviation in fluorescence correlation spectroscopy. Biophysical journal. 80 (6), 2987-2999 (2001).
- Ries, J., et al. Automated suppression of sample-related artifacts in Fluorescence Correlation Spectroscopy. Optics Express. 18 (11), 11073 (2010).
- Ries, J., Schwille, P. New concepts for fluorescence correlation spectroscopy on membranes. Physical Chemistry Chemical Physics. 10 (24), 3487 (2008).
- Mayer, M. C., et al. Amyloid precursor-like protein 1 (APLP1) exhibits stronger zinc-dependent neuronal adhesion than amyloid precursor protein and APLP2. Journal of Neurochemistry. 137 (2), 266-276 (2016).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. 33 (21), 3508-3510 (2017).
- Hammond, G. R. V., Sim, Y., Lagnado, L., Irvine, R. F. Reversible binding and rapid diffusion of proteins in complex with inositol lipids serves to coordinate free movement with spatial information. The Journal of cell biology. 184 (2), 297-308 (2009).
- Hendrix, J., Dekens, T., Schrimpf, W., Lamb, D. C. Arbitrary-Region Raster Image Correlation Spectroscopy. Biophysical journal. 111 (8), 1785-1796 (2016).
- Hendrix, J., et al. Live-cell observation of cytosolic HIV-1 assembly onset reveals RNA-interacting Gag oligomers. The Journal of cell biology. 210 (4), 629-646 (2015).
- Hendrix, J., Schrimpf, W., Höller, M., Lamb, D. C. Pulsed Interleaved Excitation Fluctuation Imaging. Biophysical Journal. 105 (4), 848-861 (2013).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5 (1), 5412 (2014).
- Chojnacki, J., et al. Envelope glycoprotein mobility on HIV-1 particles depends on the virus maturation state. Nature Communications. 8 (1), 545 (2017).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Müller, J. D., Chen, Y., Gratton, E. Resolving Heterogeneity on the Single Molecular Level with the Photon-Counting Histogram. Biophysical Journal. 78 (1), 474-486 (2000).
- Kim, S. A., Heinze, K. G., Bacia, K., Waxham, M. N., Schwille, P. Two-Photon Cross-Correlation Analysis of Intracellular Reactions with Variable Stoichiometry. Biophysical Journal. 88 (6), 4319-4336 (2005).
- Jenkins, E., et al. Reconstitution of immune cell interactions in free-standing membranes. Journal of cell science. 132 (4), (2018).

