एक अणु स्वस्थानी संकरण (एस-फिश) में फ्लोरोसेंट का उपयोग करना और मुरीन ओओसाइट्स में mRNAs का स्थानीयकरण करना ।
Summary
व्यक्तिगत अंडकोशिका में एमआरएनए की संख्या की गणना करने के लिए, स्वस्थानी संकरण (आरएनए-मछली) में एकल अणु आरएनए प्रतिदीप्ति गैर-आसंन कोशिकाओं के लिए अनुकूलित किया गया था । Oocytes एकत्र थे, प्रतिलिपि विशिष्ट जांच के साथ संकरण, और एक छवि परिमाणीकरण सॉफ्टवेयर का उपयोग कर मात्रा निर्धारित ।
Abstract
वर्तमान तरीकों में नियमित रूप से oocytes और भ्रूण में mrna मात्रा करने के लिए इस्तेमाल किया डिजिटल रिवर्स-ट्रांसक्रिप्शन पोलीमरेज़ चेन रिएक्शन (dpcr), मात्रात्मक, वास्तविक समय आरटी पीसीआर (आरटी-qpcr) और आरएनए अनुक्रमण शामिल हैं । जब इन तकनीकों एक एकल अंडक या भ्रूण का उपयोग किया जाता है, कम कॉपी mrnas मज़बूती से पता नहीं कर रहे हैं । इस समस्या से उबरने के लिए, oocytes या भ्रूण विश्लेषण के लिए एक साथ एकत्र किया जा सकता है; हालांकि, यह अक्सर नमूनों के बीच उच्च परिवर्तनशीलता की ओर जाता है । इस प्रोटोकॉल में हम शाखायुक्त डीएनए रसायन का उपयोग करते हुए स्वस्थानी संकरण (मछली) में प्रतिदीप्ति के उपयोग का वर्णन करते हैं । यह तकनीक व्यक्तिगत कोशिकाओं में एमआरएनए के स्थानिक पैटर्न को पहचानती है । जब तकनीक हाजिर खोज और ट्रैकिंग कंप्यूटर सॉफ्टवेयर के साथ युग्मित है, सेल में mRNAs की बहुतायत भी मात्रा निर्धारित किया जा सकता है । इस तकनीक का प्रयोग, वहां एक प्रयोगात्मक समूह के भीतर परिवर्तनशीलता कम है और कम oocytes और भ्रूण के लिए प्रयोगात्मक समूहों के बीच महत्वपूर्ण मतभेदों का पता लगाने की आवश्यकता है । व्यावसायिक रूप से उपलब्ध शाखित-डीएनए एसएम-फिश किटों को अनुकूलित किया गया है ताकि वे sectioned ऊतकों या स्लाइड पर आसंन कोशिकाओं में mRNAs का पता लगा सकें । हालांकि, oocytes प्रभावी ढंग से स्लाइड्स और किट में कुछ अभिकर्मकों का पालन नहीं कर रहे थे बहुत कठोर oocytes lysis में जिसके परिणामस्वरूप । इस लघटन को रोकने के लिए फिश किट में कई संशोधन किए गए । विशेष रूप से, अंडक पारभविलन और धोने अंडक और भ्रूण की इम्यूनोफ्लोरेसेंस के लिए डिजाइन बफ़र्स मालिकाना बफ़र्स की जगह । पारभासी, washes, और जांच और एम्पलीफायर के साथ इन्क्यूबिशन्स 6-कूप प्लेटों में प्रदर्शन किया और oocytes बढ़ते मीडिया का उपयोग प्रोटोकॉल के अंत में स्लाइड पर रखा गया था. इन संशोधनों को व्यावसायिक रूप से उपलब्ध किट की सीमाओं को दूर करने में सक्षम थे, विशेष रूप से, अंडक lysis । करने के लिए सही और प्रतिलिपि बनाने के लिए व्यक्तिगत oocytes में mRNAs की संख्या गिनती, कंप्यूटर सॉफ्टवेयर का इस्तेमाल किया गया था । एक साथ, यह प्रोटोकॉल पीसीआर और अनुक्रमण के लिए एक विकल्प का प्रतिनिधित्व करता है एकल कक्षों में विशिष्ट टेप की अभिव्यक्ति की तुलना करने के लिए ।
Introduction
रिवर्स-ट्रांसक्रिप्टेस पॉलीमरेज चेन रिएक्शन (पीसीआर) एमआरएनए क्वांटेशन के लिए गोल्ड स्टैंडर्ड रहा है । दो assays हैं, डिजिटल पीसीआर (डीपीपीसीआर)1 और मात्रात्मक, रीयल टाइम पीसीआर (qpcr)2 वर्तमान में उपयोग किया जाता है । दो पीसीआर तकनीकों में से, डीपीसीआर का यह सुझाव है कि यह एकल कोशिकाओं में एमआरएनए बहुतायत को मापने के लिए इस्तेमाल किया जा सकता है qPCR की तुलना में अधिक संवेदनशीलता है । हालांकि, हमारे हाथ में, प्रत्येक प्रायोगिक नमूना प्रति 5 से 10 oocytes के पूल में कम बहुतायत mRNAs के dPCR विश्लेषण कम reproducibility और उच्च रूपांतर3के साथ डेटा का उत्पादन किया है । यह आरएनए निष्कर्षण और रिवर्स ट्रांसक्रिप्शन दक्षता से संबंधित प्रयोगात्मक त्रुटि के कारण होने की संभावना है । आरएनए अनुक्रमण भी एक माउस और मानव oocytes4,5का उपयोग किया गया है । इस तकनीक की आवश्यकता है cDNA प्रवर्धन कदम पुस्तकालय पीढ़ी है जो एक प्रयोगात्मक समूह के भीतर परिवर्तनशीलता बढ़ जाने की संभावना के लिए आवश्यक है । इसके अलावा, कम बहुतायत टेप detectable नहीं हो सकता है । हालांकि अनुक्रमण कीमतों में पिछले कुछ वर्षों के नीचे चला गया है, यह अभी भी bioसूचनाविज्ञान विश्लेषण की उच्च लागत के कारण अत्यधिक लागत हो सकती है । अंत में, mRNA स्थानीयकरण प्रोटीन समारोह6के लिए योगदान स्थानिक परिवर्तन के साथ एक गतिशील प्रक्रिया है । इसलिए, हम एक तकनीक है कि सटीक और प्रतिलिपि प्रस्तुत करने योग्य मात्रात्मक उपायों और एकल oocytes में व्यक्तिगत mRNAs के स्थानीयकरण का उत्पादन होगा अपनाने के लिए निर्धारित किया है ।
शाखित डीएनए,स्वस्थाने संकरण में प्रतिदीप्ति के साथ मिलकर प्रतिदीप्ति संकेत के बजाय आरएनए/ परख संकरण की एक श्रृंखला के माध्यम से किया जाता है, प्रवर्धन (branched डीएनए का उपयोग करके), और प्रतिदीप्ति लेबलिंग चरणों के लिए प्रतिदीप्ति संकेत बढ़ाना7। यह तकनीक 18 से 25 आधार वाले ओलिनोन्यूक्लियोटाइड जांच युग्मों की बाइंडिंग से शुरू होती है जो एक विशिष्ट एमआरएनए3,8,10के पूरक होते हैं । पंद्रह से बीस जांच जोड़े लक्ष्य प्रतिलिपि के लिए विशिष्टता सुनिश्चित करने के प्रत्येक प्रतिलिपि के लिए डिजाइन किए हैं । Mrna-विशिष्ट संकरण पूर्व प्रवर्धक और प्रवर्धक जांच कि एक branched विन्यास फार्म का पालन किया जाता है । लगभग, ४०० लेबल fluorophores प्रत्येक एम्पलीफायर से बाइंड, प्रतिदीप्ति में एक ८००० गुना वृद्धि में जिसके परिणामस्वरूप व्यक्ति mRNAs का पता लगाने की अनुमति (चित्रा 1)11.
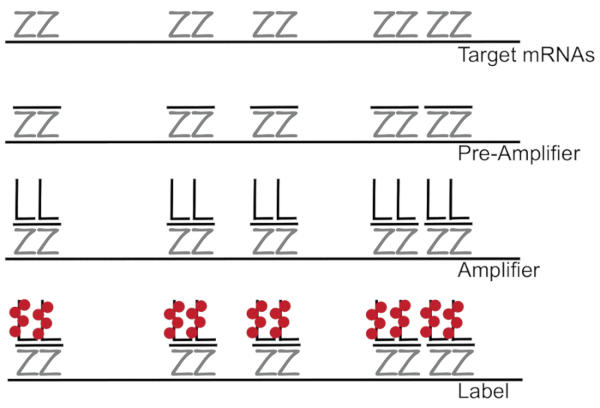
चित्रा 1: SM-मछली प्रोटोकॉल की योजनाबद्ध । ट्रांसक्रिप्ट विशिष्ट प्रोब, शाखायुक्त डीएनए प्रवर्धक तथा फ्लुओरफोर को लक्ष्य एमआरएनए के अनुक्रमिक संकरण के रूप में दर्शाया गया है । इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।
पिछले अध्ययनों में स्वस्थानी संकरण में एकल अणु प्रतिदीप्ति (SM-FISH) स्थानीयकृत β-actin mrnas में व्यक्तिगत न्यूरॉन्स12 और मानव पेपिलोमा वायरस डीएनए में सर्वाइकल कैंसर सेल लाइनों का उपयोग7. कंप्यूटर सॉफ्टवेयर स्पॉट ढूँढना और ट्रैकिंग प्रोग्राम व्यक्तिगत कबरा फ्लोरोसेंट संकेत की पहचान करता है और प्रत्येक सेल3,13में mrnas की संख्या को निर्धारित करने के लिए सफलतापूर्वक उपयोग किया गया है ।
12ंयूरॉंस में mrna का पता लगाने के परिणामों के आधार पर, हम hypothesized कि एस. एम. मछली एक उपयोगी murine oocytes और कम बहुतायत mrna सहित भ्रूण में मात्रा प्रतिलेख के लिए उपकरण साबित होता है । हालांकि, तकनीक आसंन स्थिर कोशिकाओं और formaldehyde के साथ उपयोग के लिए अनुकूलित है पैराफिन एंबेडेड (FFPE) ऊतक वर्गों । Oocytes एक स्लाइड का पालन नहीं कर सकते, तब भी जब वे पाली-एल-lysine के साथ लेपित हैं । इसके अलावा, वे कायिक कोशिकाओं और ऊतक वर्गों की तुलना में अधिक नाजुक होते हैं, जब व्यावसायिक रूप से उपलब्ध किट3में मालिकाना बफ़र्स के कुछ के अधीन सेल lysis में जिसके परिणामस्वरूप । इन चुनौतियों से उबरने के लिए, oocytes तय की और मैंयुअल रूप से buffers की बूंदों के बीच स्थानांतरित किया गया । इसके अलावा, permeabilization और किट में धोने बफ़र्स सेल lysis को कम करने के लिए प्रतिस्थापित किया गया । Predesigned जांच मछली किट या विशिष्ट टेप के साथ खरीद रहे है अनुरोध किया जा सकता है । प्रत्येक मालिकाना जांच सेट multiplexing के लिए अनुमति देने के लिए तीन प्रतिदीप्ति चैनल (C1, C2 और C3) में से एक में उपलब्ध है । वर्तमान प्रयोग में, murine oocytes दोहरे दाग रहे थे और एक C2 nanog जांच और एक C3 Pou5f1 जांच का उपयोग कर मात्रा निर्धारित. इन जांच Nanog और oocytes और भ्रूण में Pou5f1 की रिपोर्ट अभिव्यक्ति के आधार पर चयन किया गया । संकरण कदम के समापन पर, oocytes विरोधी की बूंदों में रखा गया-फ़ेड बढ़ते मीडिया ऊतकीय स्लाइड के लिए आवेदन के लिए । Confocal छवियों कबरा फ्लोरोसेंट संकेतों जो व्यक्तिगत mRNAs का प्रतिनिधित्व की संख्या को निर्धारित करने के लिए इस्तेमाल किया गया । एमएनए के परिमाण के अतिरिक्त, इमेजिंग में कोशिका में विशिष्ट मृणा के स्थानिक वितरण को भी दर्शाया गया है, जो अन्य आरएनए प्रमात्रीकरण पद्धतियों को प्राप्त करने में असमर्थ है । इस तकनीक के लिए एक प्रयोगात्मक समूह में oocytes की छोटी संख्या के उपयोग की अनुमति एक प्रायोगिक समूह के भीतर कम परिवर्तनशीलता है साबित करने के लिए प्रयोगात्मक3समूहों के बीच महत्वपूर्ण अंतर की पहचान ।
Protocol
Representative Results
Discussion
प्रोटोकॉल के दौरान छोटे कदमों की एक श्रृंखला mRNAs के सफल प्रतिदीप्ति और सटीक गिनती सुनिश्चित करेगा । पहले, प्रोटोकॉल संग्रह और oocytes का निर्धारण करने के बाद तुरंत किया जाना चाहिए । ध्यान दें कि PVP के लिए जोड़…
Divulgations
The authors have nothing to disclose.
Acknowledgements
हम स्थापना और स्थान खोजने और ट्रैकिंग 13 कार्यक्रम और संनाभि माइक्रोस्कोपी इमेजिंग के लिए नेब्रास्का लिंकन माइक्रोस्कोपी कोर के विश्वविद्यालय के तकनीकी समर्थन के उपयोग के साथ अपने उदार मदद के लिए डॉ डैनियल आर larson धंयवाद । यह अध्ययन नेब्रास्का कृषि अनुसंधान प्रभाग, लिंकन, नेब्रास्का के विश्वविद्यालय के योगदान का प्रतिनिधित्व करता है और यूएनएल हैच फंडों द्वारा समर्थित था (NEB-26-206/परिग्रहण संख्या-२३२४३५ और एनईबी-26-231/परिग्रहण संख्या-१०१३५११) ।
Materials
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
References
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).

