Användning av enda molekyl fluorescerande i Situ hybridisering (SM-fisk) för att kvantifiera och lokalisera mRNA i murina oocyter
Summary
Försäljningsbonus reproducibly numrerar av mRNA i enskilda oocyter, enda molekyl RNA fluorescens in situ var hybridisering (RNA-fisk) optimerad för icke-anhängare celler. Oocyter samlades, hybridiseras med de avskrift särskilda sonderna och kvantifieras med hjälp av ett bildbehandlingsprogram för kvantifiering.
Abstract
Nuvarande metoder som rutinmässigt används för att kvantifiera mRNA i oocyter och embryon inkluderar digital omvänd-transkription polymeras-kedjereaktion (dPCR), kvantitativa, real-time RT-PCR (RT-qPCR) och RNA-sekvensering. När dessa tekniker utförs med en enda äggcell eller embryo, upptäcks låg-kopia mRNA inte tillförlitligt. För att lösa detta problem, kan oocyter eller embryon poolas tillsammans för analys. Detta leder dock ofta till hög variation bland prover. I detta protokoll beskriver vi användningen av fluorescens in situ hybridisering (FISH) med hjälp av förgrenade DNA kemi. Denna teknik identifierar det rumsliga mönstret av mRNA i enskilda celler. När tekniken är tillsammans med plats att hitta och spårningsprogramvara, kan mängden av mRNA i cellen också kvantifieras. Med denna teknik, finns det nedsatt variabilitet inom en experimentell grupp och färre oocyter och embryon krävs för att identifiera betydande skillnader mellan experimentella grupper. Kommersiellt tillgängliga Grenade-DNA SM-fisk Kit har optimerats för att påvisa mRNA i sektionerad vävnader eller fastsittande celler på bilderna. Men oocyter effektivt följer inte bilder och vissa reagenser i kit var för hård vilket resulterar i äggcellen lysis. För att förhindra detta lysis, har flera ändringar gjorts i fisk-kit. Specifikt, ersatte äggcell permeabilisering och tvätta buffertar för immunofluorescens av oocyter och embryon de egenutvecklade buffertarna. Den permeabilisering, tvättar och inkubationer med sonder och förstärkare utfördes i 6-bra plattor och oocyter placerades på bilder i slutet av protokollet med montering media. Dessa ändringar skulle kunna övervinna begränsningarna av kommersiellt tillgängliga kit, i synnerhet äggcellen Lys. För att korrekt och reproducibly räkna antalet mRNA i enskilda oocyter, användes datorprogram. Tillsammans, utgör detta protokoll ett alternativ till PCR och sekvensering för att jämföra uttrycket av specifika avskrifter i enstaka celler.
Introduction
Transkriptas polymeras-kedjereaktion (PCR) har varit den gyllene standarden för mRNA kvantifieringsmetoden. Två analyser, digital PCR (dPCR)1 och kvantitativa, real time PCR (qPCR)2 används för närvarande. Av de två PCR-teknikerna har dPCR större känslighet än qPCR tyder på att det kunde användas att mäta mRNA överflöd i enstaka celler. Dock i våra händer producerat dPCR analys av låg överflöd mRNA i pooler av 5 till 10 ägg per varje experimental prov data med låg reproducerbarhet och hög variation3. Detta beror sannolikt på den experimentella fel i samband med RNA-extraktion och omvänd transkription effektivitet. RNA-sekvensering har också utförts med ett enda musklick och mänskliga äggceller4,5. Denna teknik kräver cDNA förstärkning steg krävs för bibliotek produktion vilket sannolikt ökar variationen inom en experimentell grupp. Låg överflöd avskrifter kan dessutom inte upptäckas. Även om sekvensering priserna har gått ner de senaste åren, kan det fortfarande vara kostnadseffektivt oöverkomliga på grund av de höga kostnaderna för bioinformatik analyser. Slutligen, mRNA lokalisering är en dynamisk process med rumsliga förändringar bidrar till protein funktion6. Därför har vi ställt ut för att anta en teknik som skulle ge exakta och reproducerbara kvantitativa åtgärder och lokalisering av enskilda mRNA i enda oocyter.
Grenad DNA kopplat till fluorescens in situ hybridisering förstärker fluorescens signal i stället för ljudförstärkande RNA/cDNA möjliggör detektering av enda mRNA i enskilda celler 7,8,9. Analysen utförs genom en serie av hybridisering, amplifiering (med förgrenade DNA) och fluorescens märkning steg för att förstärka fluorescens signal7. Tekniken börjar med bindningen av 18 – till 25-base oligonukleotiden sonden par som kompletterar en specifik mRNA3,8,10. Femton till tjugo sonden par är utformade för varje avskrift säkerställa specificitet för målet avskriften. MRNA-specifika hybridisering följs av förförstärkare och slutsteg sonder som bildar en förgrenad konfiguration. Ungefärligt binder 400 etikett fluorophores till varje förstärkare, vilket resulterar i en 8000-fold ökning fluorescens som möjliggör identifiering av enskilda mRNA (figur 1)11.
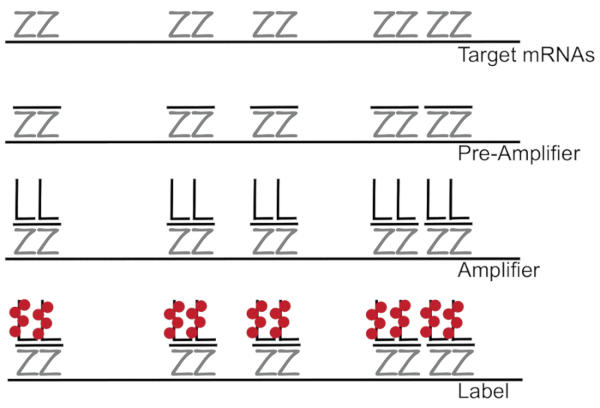
Figur 1: Schematisk av protokollet SM-fisk. Sekventiell hybridisering av avskrift specifika sond, grenad DNA förstärkare och fluorophore till ett mål som mRNA visas. Klicka här för att se en större version av denna siffra.
Tidigare studier med enda molekyl fluorescens i situ hybridisering (SM-fisk) lokaliserade β-aktin mRNA i enskilda nervceller12 och humant papillomvirus DNA i livmoderhalscancer cell linjer7. Det datorprogram som plats att hitta och spåra programmet identifierar enskilda punktuell fluorescerande signalen och har framgångsrikt använts för att kvantifiera antalet mRNA i varje cell3,13.
Baserat på resultaten av mRNA detection i nervceller12, hypotesen vi att SM-fisk skulle visa sig vara ett användbart verktyg att kvantifiera avskrift nivåer i murina oocyter och embryon inklusive låg överflöd mRNA. Men tekniken är optimerad för användning med vidhäftande fast celler och formaldehyd fast paraffin inbäddade (FFPE) vävnadssnitt. Oocyter kan inte följa en bild, även när de är belagda med Poly-L-Lysin. Dessutom är de mer ömtålig än somatiska celler och vävnadssnitt resulterar i cellen lysis när de utsätts för vissa av de egenutvecklade buffertarna i kommersiellt tillgängliga kit3. För att övervinna dessa utmaningar, var oocyter fast och manuellt överförs mellan droppar av buffertar. Dessutom byttes permeabilisering och tvätta buffertar i kits för att minska cellys. Fördesignade sonder köps tillsammans med fisk kit eller specifika avskrifter kan begäras. Varje egenutvecklade probe set finns i en av tre fluorescens kanaler (C1, C2 och C3) för att låta för multiplexering. I det nuvarande experimentet var murina oocyter dual-färgade och kvantifieras med hjälp av en C2 Nanog sond och en C3 Pou5f1 sond. Dessa sonder valdes baserat på rapporterade uttryck för Nanog och Pou5f1 i oocyter och embryon. Vid avslutningen av hybridisering steg placerades oocyter i droppar anti fade montering media för ansökan till histologiska bilder. Confocal bilder har använts för att kvantifiera antalet punktuell fluorescerande signaler som företräder enskilda mRNA. Förutom att kvantifiera mRNA, imaging visade också den rumsliga fördelningen av specifika mRNA i cellen, är som andra RNA kvantifiering metoder oförmögen att uppnå. Denna teknik visade sig ha låg variabilitet inom en experimentell grupp som medger användning av mindre antal ägg i varje experimentell grupp att identifiera betydande skillnader mellan experimentella grupper3.
Protocol
Representative Results
Discussion
En serie smärre steg under protokollet kommer att säkerställa framgångsrika fluorescens och korrekt räkningarna av mRNA. Det första måste protokollet utföras omedelbart efter insamling och fixering av oocyterna. Observera att PVP är 4% paraformaldehyd fixering bufferten att förhindra oocyter fastnar till varandra. Vi hittade att det är nödvändigt att utföra experimentet omedelbart efter insamling och fixering av oocyterna. Varje försening resulterar i en mycket lägre fluorescens signal som skulle leda til…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Vi tackar Dr. Daniel R. Larson för hans generösa hjälp med installation och användning av plats att hitta och spåra Program 13 och tekniskt stöd från University of Nebraska Lincoln mikroskopi kärnan för konfokalmikroskopi bildtagning. Denna studie utgör ett bidrag av universitetar av Nebraska jordbruks forskningsavdelning, Lincoln, Nebraska och stöddes av UNL Hatch medel (NEB-26-206/anslutningen nummer-232435 och NEB-26-231/anslutningen nummer-1013511).
Materials
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
References
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).

