Oligomerisering dynamik cellytan receptorer i levande celler av total inre reflektion fluorescens mikroskopi kombinerat med antal och ljusstyrka analys
Summary
Vi beskriver en avbildning metod för bestämning av den genomsnittliga oligomeriska tillstånd av mEGFP-märkta-receptor oligomerer induceras av ligand bindning i plasmamembranet av levande celler. Protokollet är baserat på den totala inre reflektions fluorescens (TIRF) mikroskopi kombinerat med antal och ljusstyrka (N & B) analys.
Abstract
Trots betydelsen och ubiquity av receptor oligomerization, är få metoder som gäller för att upptäcka klusterhändelser och mäta graden av klustring. Här beskriver vi en avbildning metod för att bestämma den genomsnittliga oligomeriska tillstånd av mEGFP-märkta-receptorn homokomplex i membranet av levande celler. Protokollet är baserat på den totala inre reflektions fluorescens (TIRF) mikroskopi kombinerat med antal och ljusstyrka (N & B) analys. N & B är en metod som liknar fluorescens-korrelationsspektroskopi (FCS) och Photon Counting histogram (PCH), som är baserade på den statistiska analysen av fluktuationerna i fluorescensintensiteten hos fluoroforer som sprider sig in och ut ur en belysnings volym under en observationstid. I synnerhet är N & B en förenkling av PCH för att få information om det genomsnittliga antalet proteiner i oligomeriska blandningar. Intensiteten fluktuation amplituder beskrivs av den molekylära ljusstyrkan hos fluorophore och det genomsnittliga antalet fluorofoder inom belysnings volymen. Således anser N & B endast den första och andra ögonblicken av amplitudfördelningen, nämligen den genomsnittliga intensiteten och variansen. Detta är, på samma gång, styrkan och svagheten i metoden. Eftersom endast två moment anses, N & B kan inte bestämma molar fraktion av okända oligomerer i en blandning, men det bara uppskattar den genomsnittliga oligomerisering tillstånd av blandningen. Det kan dock tillämpas på relativt små tidsserier (jämfört med andra moment metoder) av bilder av levande celler på en pixel-för-pixel-basis, helt enkelt genom att övervaka tids variationer av fluorescensintensiteten. Det minskar den effektiva tiden per pixel till några mikrosekunder, vilket gör att förvärv inom tidsintervallet sekunder till millisekunder, vilket är nödvändigt för snabb oligomerisering kinetik. Slutligen kan stora cellområden och subcellulära fack utforskas.
Introduction
Vi beskriver en total intern reflektion fluorescens-antal och ljusstyrka (TIRF-N & B) avbildning metod för att bestämma den genomsnittliga oligomeriska tillstånd av receptor molekyler vid plasmamembranet i levande celler, som syftar till att länka receptorn församlingen dynamiken till proteiners biologiska funktion (figur 1).
Vid extracellulära ligand bindning, receptorer initiera intracellulära signaltransduktion beroende på deras konformation, oligomerisering, potentiella Co-receptorer och membran sammansättning. Trots betydelsen och ubiquity av receptor oligomerization, erkänd som en viktig händelse i cellulära signalering1,2,3,4,5,6, 7, få metoder kan upptäcka klusterhändelser och mäta graden av kluster experimentellt8,9. Den konfokalvolym (x, y ≈ 300 nm, z ≈ 900 nm) är otillräckligt löst för att bevisa molekylär interaktion och stökiometri, även efter optimering av bildrestaurering algoritmer10. Den sub-Unit sammansättningen av protein oligomerer kan inte lösas på en rent rumslig grund även av super-upplösning metoder på x, y upplösning på 20-70 nm som PALM11, Storm12, och sted13. Dessutom kan deras temporala upplösning (i storleksordningen minuter per bild) inte följa kinetik i intervallet sekunder. Single molekyl Step-blekning löser stoichiometri av protein oligomerer endast om de är orörbara14.
En av de mest mångsidiga metoderna för att mäta densitet och oligomerisering av fluorescerande märkta proteiner inom enstaka bilder är den rumsliga intensiteten fördelning analys (SpIDA), som förlitar sig på rumsliga provtagning. Den är tillämplig på både kemiskt fasta och levande celler, och gör det möjligt att analysera flera regioner av intresse i cellen samtidigt med hjälp av standardfluorescens mikroskopi15. Alternativt moment metoder, såsom fluorescens-korrelationsspektroskopi (FCS)16, Photon Counting histogram (PCH)17, och antal och ljusstyrka (N & B)18,19, är lämpliga för kvantitativa Oligomer Mätningar. Dessa metoder analyserar variationer i fluorescensintensiteten som kan observeras i tid när fluoroforerna sprids in och ut ur en belysnings volym. Amplituderna i intensiteten kan beskrivas unikt genom den molekylära ljusstyrkan hos fluorophore (ε) och det genomsnittliga antalet fluoroforer (n) inom belysnings volymen17 (figur 2). Normalt kan diffusions koefficienten för fluoroforer och det genomsnittliga antalet molekyler (omvänt besläktade med G (0)-värdet) inom belysnings volymen erhållas genom FCS20. Men eftersom diffusions tiden bara skalas med kubisk roten av massan, är FCS inte tillräckligt känslig för att upptäcka förändringar i molekylmassa21. I praktiken kan enfärgade FCS inte detektera Dimerization av membran receptorer. PCH löser blandningar av olika oligomerer noggrant. Med mer än två moment av amplitudfördelningen upptäcker den molekyler med olika ljusstyrka som upptar samma belysnings volym. Scanning FCS22 och utveckling, såsom intressant par-korrelation av molekylär ljusstyrka (pcomb) Approach23, infördes för att utvidga räckvidden för tillämpligheten av fluorescenskorrelations metoder i biologiska system24 , förbli Single Point-metoder som saknar förmåga att snabba mätningar i ett stort område av en cell, som kräver många på varandra följande observationer vid varje pixel och datainsamling i storleksordningen sekunder.
N & B är en förenklad version av pch som endast beaktar den första och andra ögonblicken av amplituden för fluorescensfördelningen, nämligen Medelintensiteten, och varians, σ2 (figur 2)18,19 och på grund av detta, kan den inte bestämma molar fraktion av okända oligomerer i en blandning, men bara uppskattar den genomsnittliga oligomerisering tillstånd av blandningen. N & B har dock fördelen av att arbeta med relativt mindre tidsserier av bilder av levande celler än PCH på en pixel-för-pixel-basis, helt enkelt genom att övervaka fluktuationerna i tiden för fluorescensintensiteten. Eftersom N & B minskar tiden per pixel till några mikrosekunder, kan det följa snabbt oligomerisering kinetik över stora cellområden, vilket gör att bild förvärv på en tidsskala av sekunder i raster skanning mikroskopi (t. ex., konfokal, 2-Photon) och millisekunder i kamerabasbaserad mikroskopi (t. ex. TIRFM).
Flera rapporter har visat förmåga N & B att kvantifiera antalet subenheter i protein kluster genom att avbilda utökade cell regioner. Paxillin-EGFP-kluster upptäcktes vid vidhäftnings platserna i CHO-K1-cellerna25, och den intracellulära aggregeringen av patogenen Httex1p peptid BESKREVS i cos-7-celler26. N & B applicerades för att följa den ligand-drivna oligomeriseringen av ErbB-receptorn27, och effekten av LIGAND FGF21 på Klothob (KLB) och FGFR1c i hela cells28. Kombinationen av tirf Imaging och N & B-analys användes för att visa att dynamin-2 i första hand är tetrameriska genom hela cellmembranet29. Vi tillämpade N & B till både raster skanning och tirf bilder för att bevisa ligand-driven Dimerization av uPAR och FGFR1 cellmembran receptorer30,31.
Fluorescenskorrelations metoder, såsom N & B, FCS och PCH, bygger på föreställningen att ockupationen av partiklar i en öppen volym följer en Poissonfördelning. Eftersom endast fotoner som avger fluoroforer kan upptäckas, är medelvärdet för en uppmätt fluorescensintensitet kontra tid i en bildpunkt i bilden  , produkten av det genomsnittliga antalet fluorofomedel i belysnings volymen, n, och deras Molekylär ljusstyrka, ε17:
, produkten av det genomsnittliga antalet fluorofomedel i belysnings volymen, n, och deras Molekylär ljusstyrka, ε17:

där ε uttrycks som det antal fotoner som avges per tidsenhet (konventionellt per sekund) per molekyl när molekylen står i centrum för belysnings volymen.
Ljusstyrka är en egenskap hos varje fluorophore i en given förvärvs uppsättning, medan intensiteten är summan av alla bidrag från alla fluoroforer. I biologiska tävlingar kommer ljusstyrkan att öka med ökningen av antalet fluoroforer som fluktuerar tillsammans, vilket ger information om oligomeriseringstillståndet hos det fluorescerande-märkta proteinet. Fluktuationsamplituderna vid en given pixel mäts från variansen för fluorescenssignalen, σ2:

Om medelvärdet av kvadraten på intensitet,  och kvadraten på medelvärdet av intensiteten,
och kvadraten på medelvärdet av intensiteten,  beräknas utifrån de individuella intensitetsvärdena i varje pixel i varje ram:
beräknas utifrån de individuella intensitetsvärdena i varje pixel i varje ram:
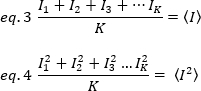
där K är antalet totala bildrutor i tidsserierna. Experimentellt är det nödvändigt att beräkna för hela bildserien variansen som beskriver scattern för de enskilda intensitetsvärdena vid varje pixel i en enskild bild runt medelintensitetsvärdet. Avvikelsen inkluderar alla variationer av olika ursprung. I en första närhet, variancen som de diffusa partiklarna i belysnings volymen, σ20, kan AVS kil ras från variancen tack vare som avkännaren sköt stojar, σ2d. De två varianserna är oberoende; således ges total avvikelsen av summan:

Variationen, på grund av molekylära variationer in och ut ur detekterings volymen, är linjärt beroende av den molekylära ljusstyrkan och intensiteten:

Omdisponering av EQ. 6 enligt EQ. 1:

Enligt det typiska konceptet i fluorescenskorrelationspektroskopi, anger ekvation 7 att variationen på grund av antalet variationer beror på kvadraten på partikel ljusstyrkan.
Därefter är variansen på grund av detektor svängningar en linjär funktion av den upptäckta intensiteten, under antagandet att detektorn drivs under dess mättnadsgräns19:

I fallet av fotonen som räknar avkännare a= 1 och c= 0, således är detektor avvikelsen jämbördig till den genomsnittliga styrkan:

För att tillämpa dessa begrepp på verkliga mätningar i Live-celler, Gratton och kollegor18 definiera skenbar ljusstyrka, B, för varje pixel som förhållandet mellan variansen över den genomsnittliga intensiteten:

B är den parameter som mäts experimentellt. I detta arbete, tidsserie bilder av FGFR1-receptorer vid plasmamembranet av HeLa celler fångas upp av TIRF mikroskopi och den genomsnittliga skenbara ljusstyrkan, B, bestäms av N & B-analys. Sedan, efter tillsats av FGF2, på varandra följande tidsserier fångas för att följa förändringarna i själv-församlingen av receptorn molekyler i membranet ytan efter stimulering av receptorn med den kanoniska ligand.
Men eftersom detektorn i TIRF-mikroskopet är en EMCCD-kamera måste uttrycket för den skenbara ljusstyrkan ändras till19:

där offset är intensitetsförskjutningen av detekterings elektroniken som är kännetecknande för detektor inställningarna. Varians och medelintensitet för en analog detektor ges respektive av:
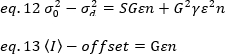
där G är den analoga förstärkningen i digitala nivåer (DL/fotoner), S, de digitala nivåerna per foton19, ges av lutningen på en intensitet kontra var Ians tomt för en ljuskälla med konstant intensitet (inga temporala fluktuationer). Γ-faktorn är relaterad till pixel detekterings volymens form. Enligt Hassler et al.32, den γ faktorn är lika med 0,3 för tirf Imaging arbetar vid maximal vinst på detekterings kameran19. Offset-, S-och G-parametrarna är kamerans och mikroskopens egenskaper. Den skenbara ljusstyrkan, B, erhålls genom omdisponering av EQ. 11 enligt EQ. 12 och 13:

Experimentellt är ε en komplex funktion av laserintensitet och detekterings effektiviteten i systemet. Eftersom B/S är linjärt beroende av ε är det dock endast viktigt att fastställa det relativa värdet av ε för ett givet detektions läge:

där ε står i proportion till ε. En kalibrering utförs fortfarande med en intern referens.
Protocol
Representative Results
Discussion
N & B kräver flera försiktighetsåtgärder vid valet av cell modell och märknings strategi. Den kan endast tillämpas på levande celler som förblir stabilt under bildfångst tiden. Extra svängningar på grund av hela cellen stel förskjutning kan hanteras med lämpliga bildrestaurering metoder38. Men i allmänhet när en cell rör sig, cellmembranet också deformeras, och struktur deformation, producerar stor extra varians, introducerar allvarlig begränsning till analysen av membranproteine…
Divulgations
The authors have nothing to disclose.
Acknowledgements
CNIC stöds av ministeriet för Ciencia, Innovacion y Universidades och Pro CNIC Foundation, och är en Severo Ochoa Center of Excellence (SEV-2015-0505). Vi stöds också av Europeiska regionala utvecklingsfonden (FEDER) “Una Manera de hacer Europa”. UC erkänner stödet från Associazione Italiana ricerca Sul Cancro, föreningen för internationell cancerforskning (numera känd som global cancer Research), och det italienska hälsoministeriet. At erkänner “Fondazione Banca del Monte di Lombardia” för att delvis stödja hans arbete med PV Fellowship “Progetto Professionalità Ivano Becchi” 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
References
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochimie. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

