Oversættelse ribosom affinitet Rensning (TRAP) til at undersøge Arabidopsis thaliana Root Development på en celletype-specifik skala
Summary
Oversættelse ribosom affinitet rensning (TRAP) giver mulighed for at dissekere udviklingsprogrammer med minimal behandling af organer og væv. Protokollen giver høj kvalitet RNA fra celler målrettet med en grøn fluorescerende protein (GFP)-mærket ribosomal subunit. Downstream analyseværktøjer, såsom qRT-PCR eller RNA-seq, afslører væv og celle type-specifikke udtryk profiler.
Abstract
I denne artikel giver vi praktiske instruktioner for at få oversætte data fra forskellige Arabidopsis thaliana rodcelletyper via den oversættende ribosom affinitetrensning (TRAP) metode og fortløbende optimeret lav-input bibliotek forberedelse.
Som udgangsmateriale anvender vi anlægslinjer, der udtrykker GFP-mærket ribosomprotein RPL18 på en celletypespecifik måde ved hjælp af passende promotorer. Før immunrensning og RNA-ekstraktion er vævet snapfrosset, hvilket bevarer vævsintegriteten og samtidig tillader udførelse af tidsseriestudier med høj tidsmæssig opløsning. Især, cellevæg strukturer forbliver intakt, hvilket er en stor ulempe i alternative procedurer såsom fluorescens-aktiveret celle sortering-baserede tilgange, der er afhængige af væv protoplasting at isolere forskellige cellepopulationer. Derudover er ingen vævsfiksering nødvendig som i lasercapture mikrodissektionsbaserede teknikker, som gør det muligt at opnå RNA af høj kvalitet.
Men prøveudtagning fra delpopulationer af celler og kun isolere polysome-associerede RNA alvorligt begrænser RNA udbytter. Det er derfor nødvendigt at anvende tilstrækkeligt følsomme metoder til tilberedning af biblioteker til en vellykket dataindsamling foretaget af RNA-seq.
TRAP er et ideelt værktøj til planteforskning, da mange udviklingsprocesser involverer cellevægrelaterede og mekaniske signalveje. Brugen af initiativtagere til at målrette specifikke cellepopulationer er at bygge bro over kløften mellem organ og encelleniveau, som igen lider under ringe opløsning eller meget høje omkostninger. Her anvender vi TRAP til at studere cellecellekommunikation i lateral roddannelse.
Introduction
Drevet af den stigende anvendelse af næste generations sekventeringsteknikker kan rumlig opløsning i udviklingsbiologi øges. Moderne undersøgelser har til formål at dissekere væv ned til specialiserede celletyper, hvis ikke enkeltcelleniveau1,2,3,4. Med henblik herpå er der i løbet af de sidste 50 år udviklet en overflod af forskellige metoder (se figur 1A)5,6,7,8,9,10,11,12,13,14,15.
Mange værktøjer i plantevidenskab har været tilpasninger af teknikker, der var banebrydende inden for dyreforsøg. Det er ikke tilfældet for den metode, vi indfører i detaljer her. I 2005, udstyret med en stærk baggrund i protein oversættelse, Bailey-Serres Lab satte sig for at ingeniør ribosomale proteiner til efterfølgende affinitet rensning16. Således kunne de undgå tidskrævende og arbejdskrævende polysome profilering, som er baseret på ultracentrifugering med en saccharose gradient og blev brugt til at vurdere oversætte ribosomer siden 1960’erne17,18. Metoden er siden blevet omtalt som translationel ribosom affinitetrensning (TRAP)16. Efter vellykkede oversættelsesundersøgelser i planter, Heiman et al. tilpasset TRAP for dyr19 og andre udvidet sin ansøgning til gær20, Drosophila21, Xenopus22 og zebrafisk23,24.
Selv om genetisk modifikation af modelsystemet er en forudsætning for TRAP, som begrænser dets anvendelse til arter, der kan underkastes genetisk transformation, kan man samtidig udnytte denne indvending til at målrette delmængder af celler, der er af særlig interesse og ellers yderst vanskeligt at isolere fra det intakte væv/organ25 (f.eks. stærkt forgrenede dendritiske celler i en musehjerne eller svampehyphae i inficeret plantevæv). I planter holdes alle celler på plads via cellevægge, der danner grundlag for det hydrostatiske skelet26. For at frigøre en plantecelle fra denne matrix har forskerne enten fysisk skåret cellen ud af det omgivende væv gennem lasercapture microdissection (LCM)27 eller udført enzymatisk fordøjelse af cellevæggene28. Blandt de sidstnævnte celler, såkaldte protoplasts, er populationen af interesse fluorescerende mærket og kan adskilles via fluorescens-aktiveret cellesortering (FACS)7. LCM kræver normalt en prøve, der skal fastsættes og indlejres i voks, hvilket i sidste ende forringer kvaliteten af sin RNA29. FACS-baserede metoder giver høj kvalitet RNA, men processen med protoplasting selv introducerer forskelle i genekspression30 og væv med modificerede og tykke sekundære cellevægge er notorisk vanskelige at behandle. Desuden antages mange udviklingsprocesser i planter at være afhængige af mekanisk transmitterede signaler, og derfor er cellevæggens integritet af afgørende betydning31. To metoder, der bruger en genvej til at omgå celleisolation ved at operere på niveauet af kerner, er fluorescensaktiveret nuklear sortering (FANS) og isolering af kerner mærket i bestemte celletyper (INTAKT). Som i TRAP, de bruger celletype-specifikke initiativtagere til at markere kerner, der efterfølgende bliver beriget via sortering eller trække ned, henholdsvis8,15. En stor udfordring for alle disse tilgange er at få tilstrækkeligt RNA-materiale fra delmængder af celler i et væv. Da TRAP kun fanger en brøkdel af de cellulære RNA’er, er prøveindsamling en betydelig flaskehals. Derfor er der behov for særligt følsomme biblioteksforberedelsesprotokoller for at producere data af høj kvalitet fra lave inputmængder.
Siden oprettelsen er TRAP enten blevet anvendt i kombination med DNA-mikroarrays eller, da sekventeringsomkostningerne er faldet betydeligt i de senere år, RNA-seq10,32,33. En lang række forskningsspørgsmål er allerede blevet belyst som gennemgået i Sablok et al.34. Vi er overbeviste om, at flere rapporter vil følge i de kommende år, da teknikken er meget alsidig, når man kombinerer forskellige initiativtagere til at målrette specifikke celletyper. Til sidst, Dette vil ske selv i en ukuelig måde, og kan kombineres med sondering plantens reaktion på mange biotiske og abiotiske stressfaktorer. Derudover, hvor stabile transgene linjer ikke er tilgængelige, behårede rod udtryksystemer er også blevet anvendt med succes til at udføre TRAP i tomat og medicago35,36.
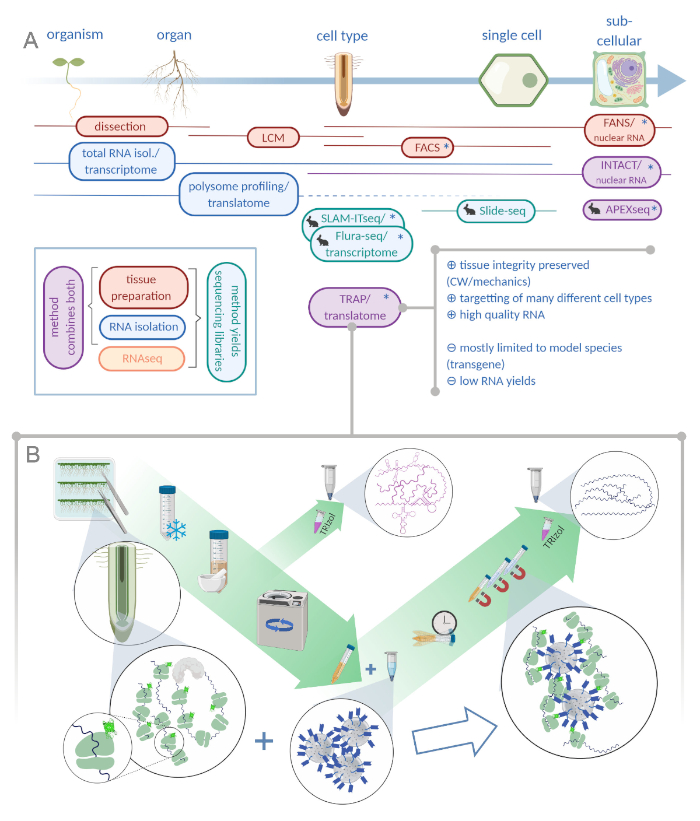
Figur 1: Oversættelse af ribosom affinitetsrensning (TRAP) supplerer analyseporteføljen “omics”. A. Stigende niveauer af analytisk præcision, ned til encellede eller endda subcellulære opløsning kan opnås ved et væld af metoder eller kombinationer heraf. Ordningen giver et overblik over de værktøjer, der i øjeblikket er til rådighed på plante- og dyreområdet. Vævssamling ved cellulær opløsning kan opnås ved protokoller som LCM eller FACS, som derefter kobles til standard transkription eller polysome profileing/translatome analyse. TRAP og INTAKT integrerer både vævsindfangning og RNA-isolation, da de er baseret på epitop-tagging. Intakt e-prøver imidlertid kun cellekerner og udgør derfor et særligt tilfælde af transskriptionsanalyse. En lille kanin ikon markerer nyudviklede metoder på dyreområdet: Mens SLAM-ITseq og Flura-seq stole på metaboliske målretning af spirende RNAs med modificerede uracil baser i celler, der udtrykker eftergivende enzym, Slide-seq gør brug af en belagt glas dias med DNA stregkoder, der giver positionelle oplysninger i cellulære område. Der anvendes en nærhedsmærkningsmetode i APEX-seq for at udtage prøver af RNA’er i specifikke subcellulære rum. Navnlig kræver øget opløsning ofte generering af transgent materiale (stjerner), og disse metoder anvendes således overvejende til modelarter. TRAP er specielt velegnet til plantevidenskabelige undersøgelser, der involverer cellevæg (CW) eller mekaniker signalering samt cellearter, der er vanskelige at frigive fra deres CW matrix. B. Detaljerede vådlaboratorietrin i TRAP-proceduren: Kimplanter, der udtrykker GFP-mærket ribosomprotein i forskellige celletyper (f.eks. rodendodermis), dyrkes på petriskåle i syv dage og rodmateriale høstet ved snapfrysning. En total RNA-kontrolprøve indsamles fra det homogeniserede rå ekstrakt, før det pelleteres i snavset via centrifugering. Magnetiske anti-GFP perler tilsættes til det klare ekstrakt for at udføre immunudfældning. Efter inkubation og tre vasketrin opnås det polysomeassocierede RNA (TRAP/polysome RNA) direkte via fenol-chloroform ekstraktion. LCM: mikrodissektion af laserfanger, FACS/FANS: fluorescensaktiveret celle/nuklear sortering APEX-seq: metode baseret på manipuleret ascorbatperoxidase, INTAKT: isolering af kerner mærket i specifikke celletyper, SLAM-ITseq: thiol(SH)-linked alkylering for metabolisk sekventering af RNA i væv, Flura-seq: fluorouracil-mærket RNA sekventering (Oprettet med Biorender.com) Klik her for at se en større version af denne figur.
Målet med denne artikel er at give en detaljeret beskrivelse af TRAP-metoden, at fremhæve kritiske trin og at give vejledning til en mulig metode til forberedelse af biblioteket.
Et generisk TRAP-eksperiment vil hovedsagelig bestå af følgende trin (se også figur 1B):(1) Fremstilling af plantemateriale, herunder kloning af ribosomt-taggingkonstruktion, transgene linjeproduktion og -udvælgelse, dyrkning og opsamling af frø, sterilisering og plettering samt stressanvendelse/-behandling (valgfri) og vævshøst; 2) immunrensning, herunder vævshomogenisering og rydning af råekstrakt, perlevask og immunrensning samt vasketrin 3) RNA-ekstraktion og kvalitetsvurdering og (4) biblioteksforberedelse.
Arabidopsis-roden har været et modelsystem til undersøgelse af planteudvikling lige siden dens indførelse som modelanlæg37,38. Her er anvendelsen af TRAP fremvist i forbindelse med anlæggetlateral rod udvikling. I planter, opbygningen af hele rodsystemet er afhængig af udførelsen af dette program og er derfor meget vigtigt for overlevelsen af organismen39. I Arabidopsisstammer laterale rødder fra pericyclevæv , der ligger ved siden af xylem-karog derfor kaldes xylempolpericycle (XPP; se figur 2C)40. Nogle XPP-celler, som er placeret dybt inde i roden, erhverve en grundlægger celleidentitet og, på en lokal hormonelle udløse, begynder at formere sig ved hævelse og dividere anticlinally41. Men på grund af tilstedeværelsen af en stiv cellevæg matrix, denne proces udøver mekanisk stress på det omgivende væv. Især er den overliggende endodermis påvirket, da det er i vejen for den laterale rodvækst akse42,43,44. Faktisk vil den nydannende primordium nødt til at vokse gennem den overliggende endodermis celle (Figur 2C2), mens cortex og epidermis celler er bare skubbet til side for primordium til endelig at dukke45,46. Nylige arbejde i vores laboratorium har vist, at endodermis aktivt bidrager til at imødekomme spredning i perihjulet. Målrettet blokering af endodermal hormonel signalering er tilstrækkelig til at hæmme selv den allerførste division i XPP-cellerne47. Derfor pericycle-endodermis kommunikation udgør en meget tidlig checkpoint for lateral rod udvikling i Arabidopsis. Det vides dog ikke, hvordan denne krydstale udføres. For at opklare dette mysterium valgte vi TRAP-seq-tilgangen til at målrette XPP og endodermale celler. At berige for celler i den laterale rod program, vi efterlignede hormonelle udløse ved eksogent anvende en auxin analog (1-naphthaleneeddikesyre, NAA)48, som samtidig lov til at tidsmæssigt løse den indledende fase af lateral roddannelse.
Protocol
Representative Results
Discussion
Verificering af lokaliseringsmønsteret RPL18
Afgørende for at undgå fejlfortolkning af data fra ethvert TRAP-eksperiment er det korrekte udtryksmønster for den mærkede ribosomal-underenhed. Derfor indarbejdes GFP som en epitop tag til RPL18 meget elegant tillader verifikation af det ønskede udtryk mønster og fortløbende, pulldown af polysom fraktion fra samme væv. Mere invasive tilgange til at sikre ordentlig promotor mønstre efterfølges af Jiao og Mayerowitz 2010, som kræver GUS-farvning …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Vi vil gerne takke Jean-Claude Walser fra Det Genetiske Mangfoldighedscenter Zürich for afgørende ekspertrådgivning i den tidlige fase af dette projekt. Arbejdet i Vermeer-laboratoriet blev støttet af et SNF Professorship grant (PP00P3_157524) og et Tilskud til R’EQUIP-udstyr (316030_164086) fra Swiss National Science Foundation (SNSF), der blev tildelt JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

