एक सेल प्रकार विशिष्ट पैमाने पर अरबीdopsis थैलियाना रूट विकास की जांच करने के लिए रिबोसोम एफ़िनिटी शुद्धि (ट्रैप) का अनुवाद
Summary
राइबोसोम आत्मीयता शुद्धिकरण (ट्रैप) का अनुवाद करने से अंगों और ऊतकों के न्यूनतम प्रसंस्करण के साथ विकासात्मक कार्यक्रमों को विच्छेदन करने की संभावना प्रदान होती है। प्रोटोकॉल एक हरे फ्लोरोसेंट प्रोटीन (GFP) लेबल राइबोसोमल उपइकाई के साथ लक्षित कोशिकाओं से उच्च गुणवत्ता वाले आरएनए पैदावार । डाउनस्ट्रीम विश्लेषण उपकरण, जैसे क्यूआरटी-पीसीआर या आरएनए-सीक्यू, ऊतक और सेल प्रकार-विशिष्ट अभिव्यक्ति प्रोफाइल को प्रकट करते हैं।
Abstract
इस लेख में, हम अनुवादराइसोम एफ़िनिटी शुद्धि (ट्रैप) विधि और लगातार अनुकूलित कम इनपुट लाइब्रेरी तैयारी के माध्यम से विभिन्न अरबीडोप्सिस थैलियाना रूट सेल प्रकारों से अनुवाद डेटा प्राप्त करने के लिए हाथ पर निर्देश देते हैं।
प्रारंभिक सामग्री के रूप में, हम पौधे की लाइनों को नियोजित करते हैं जो पर्याप्त प्रमोटरों के उपयोग से सेल प्रकार-विशिष्ट तरीके से जीएफपी-टैग किए गए राइबोसोमल प्रोटीन आरपीएल18 को व्यक्त करते हैं। इम्यूनोशुद्धि और आरएनए निष्कर्षण से पहले, ऊतक स्नैप फ्रोजन है, जो ऊतक अखंडता को बरकरार रखता है और साथ ही उच्च लौकिक संकल्प के साथ समय श्रृंखला अध्ययन के निष्पादन की अनुमति देता है। विशेष रूप से, सेल दीवार संरचनाएं बरकरार रहती हैं, जो वैकल्पिक प्रक्रियाओं में एक प्रमुख खामी है जैसे फ्लोरेसेंस-सक्रिय सेल छंटाई-आधारित दृष्टिकोण जो अलग सेल आबादी को अलग करने के लिए ऊतक प्रोटोस्टॉन्स्तक पर भरोसा करते हैं। इसके अतिरिक्त, लेजर कैप्चर माइक्रोडिसेक्शन-आधारित तकनीकों के रूप में कोई ऊतक निर्धारण आवश्यक नहीं है, जो उच्च गुणवत्ता वाले आरएनए को प्राप्त करने की अनुमति देता है।
हालांकि, कोशिकाओं की उपआबादी से नमूना और केवल पॉलीसम से जुड़े आरएनए को अलग करना आरएनए पैदावार को गंभीर रूप से सीमित करता है। इसलिए आरएनए-सीक्यू द्वारा सफल डेटा अधिग्रहण के लिए पर्याप्त रूप से संवेदनशील पुस्तकालय तैयार करने के तरीकों को लागू करना आवश्यक है।
ट्रैप पौधे अनुसंधान के लिए एक आदर्श उपकरण प्रदान करता है क्योंकि कई विकासात्मक प्रक्रियाओं में सेल दीवार से संबंधित और यांत्रिक सिग्नलिंग रास्ते शामिल हैं। विशिष्ट सेल आबादी को लक्षित करने के लिए प्रमोटरों का उपयोग अंग और एकल-कोशिका स्तर के बीच अंतर को पाट रहा है जो बदले में थोड़ा संकल्प या बहुत अधिक लागत से पीड़ित है। यहां, हम पार्श्व जड़ गठन में सेल-सेल संचार का अध्ययन करने के लिए ट्रैप लागू करते हैं।
Introduction
अगली पीढ़ी की अनुक्रमण तकनीकों के बढ़ते आवेदन से प्रेरित होकर विकासात्मक जीव विज्ञान में स्थानिक संकल्प को बढ़ाया जा सकता है । समकालीन अध्ययनों का उद्देश्य ऊतकों को विशेष कोशिका प्रकारों से विभाजित करना है, यदि एकल-कोशिका स्तर1,,2,,3,44नहीं है। इसके लिए पिछले पचास वर्षों में विभिन्न तरीकों की अधिकता तैयार की गई है (देखें चित्रा 1A)5,6,7,8,9,10,11,12,13,14,15.,
संयंत्र विज्ञान में कई उपकरण तकनीकों के अनुकूलन रहे हैं जिन्हें पशु अनुसंधान में बीड़ा उठाया गया था। यह तरीका हम यहां विस्तार से शुरू कर रहे है के लिए मामला नहीं है । 2005 में, प्रोटीन अनुवाद में एक मजबूत पृष्ठभूमि से लैस, बेली-सेरेस लैब ने बाद में आत्मीयता शुद्धि16के लिए राइबोसोमल प्रोटीन इंजीनियर के लिए तैयार किया। इस प्रकार, वे समय लेने वाली और श्रम-प्रधान पॉलीसम प्रोफाइलिंग से बच सकते हैं, जो एक सुक्रोज ढाल के साथ अल्ट्रासेंट्रोइफेशन पर आधारित है और 1 9 60 के दशक17,,18के बाद से राइबोसोम ्स के अनुवाद का आकलन करने के लिए उपयोग किया गया था। विधि के बाद से अनुवादीय राइबोसोम आत्मीयता शुद्धि (TRAP)16के रूप में संदर्भित किया गया है । पौधों में सफल अनुवाद अध्ययन के बाद, हीमैन एट अल. जानवरों के लिए अनुकूलित ट्रैप19 और अन्य ने खमीर20, ड्रोसोफिला21, ज़ेनोपस22 और जेब्राफिश23,,24तक अपना आवेदन बढ़ाया।
हालांकि मॉडल प्रणाली के आनुवंशिक संशोधन जाल के लिए एक शर्त है, जो आनुवंशिक परिवर्तन के लिए उत्तरदायी प्रजातियों के लिए अपने आवेदन सीमा है, एक साथ25 इस आपत्ति का दोहन करने के लिए कोशिकाओं के सबसेट है कि विशेष रुचि के है और अंयथा बेहद मुश्किल बरकरार ऊतक से अलग करने के लिए कर सकते है/ पौधों में, सभी कोशिकाओं को सेल दीवारों के माध्यम से जगह में रखा जाता है जो हाइड्रोस्टैटिक कंकाल26का आधार बनाते हैं। इस मैट्रिक्स से एक संयंत्र कोशिका को मुक्त करने के लिए, वैज्ञानिकों ने या तो शारीरिक रूप से लेजर कैप्चर माइक्रोडिसेक्शन (एलसीएम)27 के माध्यम से अपने आसपास के ऊतकों से बाहर कोशिका को काट दिया है या सेल दीवारों28के एंजाइमेटिक पाचन प्रदर्शन किया है । बाद की कोशिकाओं में, तथाकथित प्रोटोप्लास्ट, ब्याज की आबादी फ्लोरोसेंटी लेबल है और फ्लोरेसेंस-सक्रिय सेल छंटाई (FACS)7के माध्यम से अलग किया जा सकता है । एलसीएम को आमतौर पर मोम में तय और एम्बेडेड होने के लिए एक नमूना की आवश्यकता होती है, जो अंततः इसके आरएनए29की गुणवत्ता को खराब कर देता है। FACS आधारित तरीकों उच्च गुणवत्ता वाले आरएनए उपज है, लेकिन प्रोटॉपअनेशन की प्रक्रिया ही जीन अभिव्यक्ति30 में मतभेदों का परिचय और संशोधित और मोटी माध्यमिक सेल दीवारों के साथ ऊतकों का इलाज करने के लिए बेहद मुश्किल हैं । इसके अलावा, पौधों में कई विकासप्रक्रियाओं को यांत्रिक रूप से प्रेषित संकेतों पर भरोसा करने के लिए माना जाता है और इसलिए सेल की दीवार की अखंडता31सर्वोपरि महत्व की है। दो तरीके, जो न्यूक्ली के स्तर पर परिचालन करके सेल अलगाव को दरकिनार करने के लिए शॉर्टकट का उपयोग करते हैं, फ्लोरेसेंस-सक्रिय परमाणु छंटाई (प्रशंसक) और विशिष्ट सेल प्रकार (बरकरार) में टैग किए गए नाभिक के अलगाव हैं। ट्रैप के रूप में, वे नाभिक को चिह्नित करने के लिए सेल प्रकार-विशिष्ट प्रमोटरों का उपयोग करते हैं, जो बाद में क्रमशः8,,15को छंटाई या नीचे खींचने के माध्यम से समृद्ध हो जाते हैं। इन सभी दृष्टिकोणों के लिए एक बड़ी चुनौती ऊतक में कोशिकाओं के सबसेट से पर्याप्त आरएनए सामग्री प्राप्त करना है। चूंकि ट्रैप सेलुलर आरएनए के केवल एक अंश को कैप्चर करता है, नमूना संग्रह काफी अड़चन है। इसलिए, कम इनपुट राशि से उच्च गुणवत्ता वाले डेटा का उत्पादन करने के लिए विशेष रूप से संवेदनशील पुस्तकालय तैयार करने के प्रोटोकॉल की आवश्यकता होती है।
अपनी स्थापना के बाद से, ट्रैप का उपयोग या तो डीएनए माइक्रोरे के संयोजन में किया गया है या, क्योंकि अनुक्रमण लागत हाल के वर्षों में काफी गिरा, आरएनए-सीक्यू10,,32,,33। सबलोक एट अल३४में समीक्षा के रूप में अनुसंधान प्रश्नों की एक भीड़ पहले से ही स्पष्ट किया गया है । हम आश्वस्त है कि अधिक रिपोर्ट आने वाले वर्षों में पालन करेंगे के रूप में तकनीक बहुत बहुमुखी है जब विभिंन प्रमोटरों के संयोजन के लिए विशिष्ट सेल प्रकार को लक्षित । अंततः, यह भी एक inducible तरीके से किया जाएगा, और कई बायोटिक और अजैविक तनाव कारकों के लिए संयंत्र की प्रतिक्रिया की जांच के साथ संयुक्त किया जा सकता है । इसके अतिरिक्त, जहां स्थिर ट्रांसजेनिक लाइनें उपलब्ध नहीं हैं, बालों वाली रूट अभिव्यक्ति प्रणालियों का उपयोग टमाटर और मेडिगो35,,36में ट्रैप करने के लिए सफलतापूर्वक किया गया है।
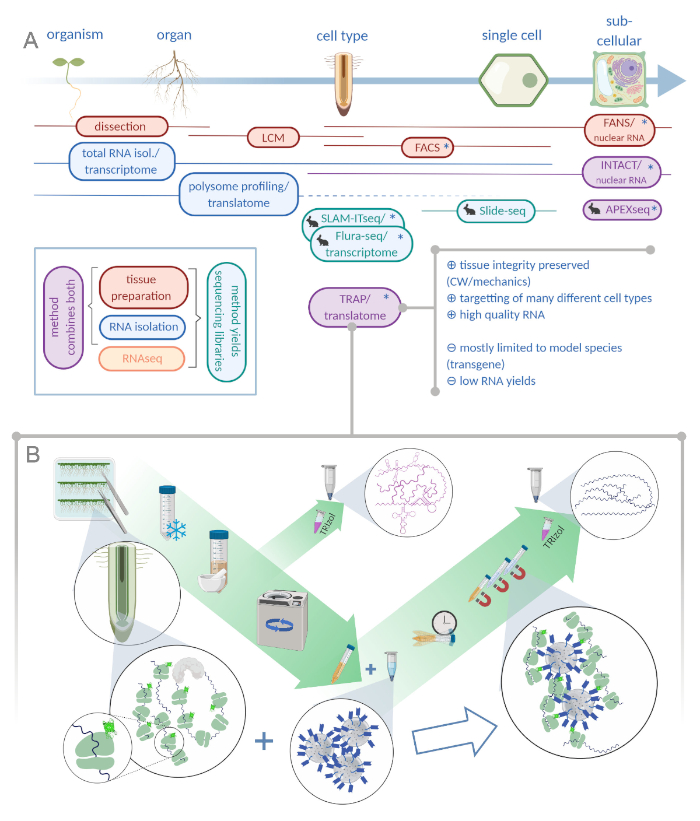
चित्रा 1: राइबोसोम आत्मीयता शुद्धिकरण (ट्रैप) का अनुवाद “ओमिक्स” विश्लेषण पोर्टफोलियो का पूरक है। A. विश्लेषणात्मक परिशुद्धता के बढ़ते स्तर, एकल सेल या यहां तक कि उपकोशिकीय संकल्प के लिए नीचे तरीकों या उसके संयोजन की अधिकता से प्राप्त किया जा सकता है । यह योजना संयंत्र और पशु क्षेत्र में वर्तमान में उपलब्ध उपकरणों का अवलोकन देती है । सेलुलर रिज़ॉल्यूशन पर ऊतक संग्रह एलसीएम या एफएसीएस जैसे प्रोटोकॉल द्वारा प्राप्त किया जा सकता है, जो तब मानक प्रतिलेखन या पॉलीसम प्रोफाइलिंग/अनुवाद विश्लेषण के साथ मिलकर होते हैं। ट्रैप और बरकरार ऊतक कैप्चर और आरएनए अलगाव दोनों को एकीकृत करते हैं क्योंकि वे एपिटोप-टैगिंग पर आधारित होते हैं। हालांकि, बरकरार नमूने केवल सेल न्यूक्लिकी और गठन, इसलिए, ट्रांसक्रिप्टोम विश्लेषण का एक विशेष मामला है । एक छोटा खरगोश आइकन पशु क्षेत्र में नए विकसित तरीकों को चिह्नित करता है: जबकि स्लैम-ITseq और फ्लूरा-सीक्यू स्वतंत्र एंजाइम व्यक्त कोशिकाओं में संशोधित यूसिल ठिकानों के साथ नवजात आरएनए के मेटाबोलिक टारगेटिंग पर भरोसा करते हैं, स्लाइड-सीक्यू डीएनए बारकोड के साथ एक लेपित ग्लास स्लाइड का उपयोग करता है जो सेलुलर रेंज में स्थितीय जानकारी प्रदान करता है। विशिष्ट उपकोशिकीय डिब्बों में आरएनए का नमूना लेने के लिए एपेक्स-सीक्यू में निकटता-लेबलिंग दृष्टिकोण का पालन किया जाता है। विशेष रूप से, बढ़े हुए संकल्प के लिए अक्सर ट्रांसजेनिक सामग्री (तारक) की पीढ़ी की आवश्यकता होती है और इस प्रकार इन तरीकों का उपयोग मुख्य रूप से मॉडल प्रजातियों के लिए किया जाता है। ट्रैप विशेष रूप से पौधे विज्ञान अध्ययनों के लिए अनुकूल है जिसमें सेल वॉल (सीडब्ल्यू) या मैकेनिक सिग्नलिंग के साथ-साथ सेल प्रजातियां शामिल हैं जिन्हें उनके सीडब्ल्यू मैट्रिक्स से छोड़ना मुश्किल है। बी जाल प्रक्रिया के विस्तृत गीले प्रयोगशाला कदम: अलग सेल प्रकार (जैसे रूट एंडोडरमिस) में GFP-टैग राइबोसोमल प्रोटीन व्यक्त रोपण सात दिनों के लिए पेट्री व्यंजन ों पर उगाए जाते हैं और स्नैप फ्रीजिंग द्वारा काटा गया जड़ सामग्री। सेंट्रलाइज्ड के जरिए मलबे को पैलेट करने से पहले एमोजेनाइज्ड क्रूड एक्सट्रैक्ट से कुल आरएनए कंट्रोल सैंपल एकत्र किया जाता है । इम्यूनोप्रिसिप्रिशन करने के लिए क्लीयर किए गए एक्सट्रैक्ट में मैग्नेटिक एंटी-जीएफपी मोतियों को जोड़ा जाता है। इनक्यूबेशन और तीन वॉश स्टेप्स के बाद पॉलीसम से जुड़े आरएनए (ट्रैप/पॉलीसम आरएनए) को सीधे फिनॉल-क्लोरोफॉर्म एक्सट्रैक्शन के जरिए प्राप्त किया जाता है । एलसीएम: लेजर कैप्चर माइक्रोडिसेक्शन, FACS/FANS: फ्लोरेसेंस-सक्रिय सेल/ एपेक्स-सीक्यू: इंजीनियर एकोर्सबेट पेरोक्सिडेस पर आधारित विधि, बरकरार: विशिष्ट सेल प्रकारों में टैग किए गए नाभिक का अलगाव, स्लैम-आईटीएसक्यू: थियोल (एसएच) – ऊतक में आरएनए के मेटाबोलिक अनुक्रमण के लिए अल्बाइलेशन से जुड़ा हुआ है, फ्लूरा-सीक्यू: फ्लोरोरासिल-लेबल आरएनए अनुक्रमण (Biorender.com के साथ बनाया गया) कृपया यहां क्लिक करें
इस लेख का लक्ष्य जाल विधि का विस्तृत विवरण प्रदान करना, महत्वपूर्ण चरणों को उजागर करना और संभावित पुस्तकालय तैयारकरने की विधि के लिए मार्गदर्शन प्रदान करना है।
एक जेनेरिक ट्रैप प्रयोग में अनिवार्य रूप से निम्नलिखित चरण शामिल होंगे (चित्रा 1Bभी): (1) राइबोसोम-टैगिंग निर्माण की क्लोनिंग, ट्रांसजेनिक लाइन उत्पादन और चयन, बीज, नसबंदी और चढ़ाना, और तनाव आवेदन/उपचार (वैकल्पिक) और ऊतक संचयन की क्लोनिंग सहित संयंत्र सामग्री की तैयारी; (2) ऊतक समरूपता और कच्चे तेल के अर्क के समाशोधन, मनका धोने और इम्यूनोशुद्धि, और धोने के कदम सहित इम्यूनोशुद्धि; (3) आरएनए निष्कर्षण और गुणवत्ता मूल्यांकन; और (4) पुस्तकालय की तैयारी।
अरबीडोप्सिस रूट एक मॉडल प्लांट37,38के रूप में शुरू होने के बाद से पौधों के विकास का अध्ययन करने के लिए एक मॉडल प्रणाली रही है . यहां, ट्रैप के आवेदन को पौधे पार्श्व जड़ विकास के संदर्भ में प्रदर्शित किया जाता है। पौधों में, पूरे रूट सिस्टम का बिल्डअप इस कार्यक्रम के निष्पादन पर निर्भर करता है और इसलिए जीव39के अस्तित्व के लिए बहुत महत्वपूर्ण है। अरबीडोप्सिसमें पार्श्व जड़ें पेरिसाइकिल ऊतक से उत्पन्न होती हैं जो दारू के जहाजों के बगल में रहती हैं और इसलिए दारू ध्रुव पेरिसाइकिल (XPP; देखें चित्रा 2 C)40। कुछ एक्सपीपी कोशिकाएं, जो जड़ के अंदर गहरे स्थित हैं, एक संस्थापक सेल पहचान प्राप्त करती हैं और एक स्थानीय हार्मोनल ट्रिगर पर, सूजन और एंटीक्लिनली41को विभाजित करके पैदा होना शुरू कर देती हैं। हालांकि, एक कठोर सेल दीवार मैट्रिक्स की उपस्थिति के कारण, यह प्रक्रिया आसपास के ऊतकों पर यांत्रिक तनाव डालती है। विशेष रूप से, अंतर्निहित एंडोडरमिस प्रभावित होता है, क्योंकि यह पार्श्व जड़ वृद्धि धुरी42,43,44के रास्ते में है। दरअसल, नए बनाने वाले प्राइमोर्डियम को ओवरलाइंग एंडोडरमिस सेल(चित्रा 2C2)के माध्यम से विकसित करना होगा जबकि कॉर्टेक्स और एपिडर्मिस कोशिकाओं को प्राइमोर्डियम के लिए अंत में45,,46उभरने के लिए अलग धकेल दिया जाता है। हमारी प्रयोगशाला में हाल के काम से पता चला है कि एंडोडरमिस सक्रिय रूप से pericycle में प्रसार को समायोजित करने के लिए योगदान दे रहा है । एंडोडरमल हार्मोनल सिग्नलिंग को लक्षित अवरुद्ध करना एक्सपीपी कोशिकाओं47में प्रथम श्रेणी को बाधित करने के लिए पर्याप्त है । इसलिए, पेरिसाइकिल-एंडोडरमिस संचार अरबीडोप्सिसमें पार्श्व जड़ विकास के लिए एक बहुत ही प्रारंभिक चौकी का गठन करता है। हालांकि यह पता नहीं चल पाया है कि यह क्रॉसटॉक कैसे किया जाता है । इस रहस्य को जानने के लिए, हमने एक्सपीपी और एंडोडरमल कोशिकाओं को लक्षित करने के लिए ट्रैप-सीक्यू दृष्टिकोण चुना। पार्श्व रूट कार्यक्रम में कोशिकाओं के लिए समृद्ध करने के लिए, हमने एक ऑक्सिन एनालॉग (1-नेफ्थेलेनेटिक एसिड, एनएए)48को लागू करके हार्मोनल ट्रिगर की नकल की, जिसने साथ ही पार्श्व जड़ गठन के प्रारंभिक चरण को अस्थायी रूप से हल करने की अनुमति दी।
Protocol
Representative Results
Discussion
आरपीएल18 स्थानीयकरण पैटर्न का सत्यापन
किसी भी ट्रैप प्रयोग से डेटा की गलत व्याख्या से बचने के लिए महत्वपूर्ण टैग राइबोसोमल उपइकाई का उचित अभिव्यक्ति पैटर्न है। इसलिए, आरपीएल18 के लिए एक एपिटोप ट…
Divulgations
The authors have nothing to disclose.
Acknowledgements
हम इस परियोजना के प्रारंभिक चरण में महत्वपूर्ण विशेषज्ञ सलाह के लिए आनुवंशिक विविधता केंद्र ज्यूरिख के जीन-क्लाउड वालसर का शुक्रिया अदा करना चाहते हैं। वर्मीयर लैब में काम को जेएमवी को सम्मानित स्विस नेशनल साइंस फाउंडेशन (एसएनएसएफ) से एसएनएफ प्रोफेसरशिप ग्रांट (PP00P3_157524) और एक आर लैस उपकरण अनुदान (316030_164086) द्वारा समर्थित किया गया था।
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

