세포 유형 별 척도에서 애기장대 뿌리 개발을 조사하기 위해 리보솜 친화성 정제 (TRAP) 번역
Summary
리보솜 친화도 정제(TRAP)를 번역하면 장기와 조직의 최소한의 처리로 개발 프로그램을 해부할 수 있습니다. 이 프로토콜은 녹색 형광 단백질(GFP)으로 표적화된 세포로부터 고품질의 RNA를 산출합니다. qRT-PCR 또는 RNA-seq와 같은 다운스트림 분석 도구는 조직 및 세포 유형별 발현 프로파일을 밝힙습니다.
Abstract
이 기사에서는 번역 리보솜 친화도 정제 (TRAP) 방법 및 연속최적화 된 낮은 입력 라이브러리 준비를 통해 상이한 애기장대 탈리아나 루트 세포 유형에서 translatome 데이터를 얻기 위한 실습 지침을 제공합니다.
시작 재료로서, 우리는 적절한 프로모터를 사용하여 세포 유형별 방식으로 GFP 태그리보소말 단백질 RPL18을 발현하는 식물 라인을 사용합니다. 면역 정화 및 RNA 추출 전에 조직은 고정되어 조직 무결성을 보존하고 동시에 높은 시간적 해상도로 타임 시리즈 연구를 실행할 수 있습니다. 특히, 세포벽 구조는 손상되지 않은 상태로 남아 있으며, 이는 뚜렷한 세포 집단을 분리하기 위해 조직 에 의존하는 형광 활성화 세포 선별 기반 접근법과 같은 대체 절차의 주요 단점입니다. 또한 고품질 RNA를 얻을 수 있는 레이저 포획 미세 해부 기반 기술에서와 같이 조직 고정이 필요하지 않습니다.
그러나, 세포의 하위 집단에서 샘플링만 폴리섬 관련 RNA를 격리하는 것은 RNA 수율을 심각하게 제한합니다. 따라서 RNA-seq에 의한 성공적인 데이터 수집을 위해 충분히 민감한 라이브러리 준비 방법을 적용해야 합니다.
TRAP은 세포벽 관련 및 기계적 신호 경로와 관련된 많은 개발 공정으로 플랜트 연구에 이상적인 도구를 제공합니다. 특정 세포 집단을 표적으로 하는 프로모터의 사용은 차례차례로 작은 해결책 또는 아주 높은 비용 때문에 손해를 입는 기관과 단세포 수준 사이 격차를 다리로 입니다. 여기에서, 우리는 측방 루트 형성에 있는 세포 세포 통신을 공부하기 위하여 TRAP를 적용합니다.
Introduction
차세대 염기서열 분석 기술의 응용이 증가함에 따라 개발 생물학의 공간 해상도가 증대될 수 있습니다. 현대 연구는 단세포 수준1,2,2,3,,4가아니라면, 전문화한 세포 모형에 조직을 해부하는 것을 목표로 합니다. 이를 위해, 지난 50년 동안 다양한 방법이 고안되었다(도 1A참조)5,6,6,77,8,,9,,10,,11,12,,13,,14,,15.,
식물 과학의 많은 도구는 동물 연구에서 개척 된 기술의 적응되었습니다. 여기에서 자세히 소개하는 방법은 그렇지 않습니다. 2005년, 단백질 번역에 대한 탄탄한 배경을 갖춘 베일리-세레스 연구소는 후속 친화도 정제16을위해 리보소말 단백질을 설계하기 시작했습니다. 따라서, 그들은 수크로오스 그라데이션을 가진 초원심분리에 기초하고 1960년대,17,18년이후 리보솜 을 번역하는 데 사용된 시간 소모적이고 노동 집약적인 폴리솜 프로파일링을 피할 수 있었다. 상기 방법은 이후 번역리보솜 친화도 정제(TRAP)16으로지칭되었다. 식물에서 성공적인 트랜스플라토메 연구 후, Heiman 등은 동물19에 대한 TRAP을 적용했으며 다른 동물은 효모20, 초파리21, 제노푸스22 및 제브라피시23,,24로적용을 확대했다.
모델 시스템의 유전자 변형은 TRAP의 전제 조건이지만, 이는 유전자 변형에 대한 적용을 제한하는 종에 대한 적용을 제한하지만, 하나는 동시에 특별한 관심과 그렇지 않으면 손상되지 않은 조직 / 기관(예를 들어, 감염된 식물 에서 마우스 뇌 또는 곰팡이 hyphae에서 고도로 분지 수지상 세포)에서 분리하기 매우 어려운 세포의 하위 세트를 대상으로이 반대를 활용할 수 있습니다. 식물에서, 모든 세포는 정수체골격(26)의기초를 형성하는 세포벽을 통해 제자리에 유지된다. 이 매트릭스에서 식물 세포를 해방하기 위하여는, 과학자는 레이저 포획 미세 분부 (LCM)27를 통해 그 주변 조직에서 세포를 물리적으로 잘라내거나 세포벽28의효소 소화를 수행했습니다. 후자의 세포 들 중, 소위 프로토 플라스타체, 관심있는 집단은 형광 표지되고 형광 활성화 세포 선별(FACS)7을통해 분리될 수 있다. LCM은 일반적으로 그것의 RNA29의질을 궁극적으로 악화시키는 왁스에서 고정되고 매립되는 견본을 요구합니다. FACS 기반 방법은 고품질 RNA를 산출하지만, 프로토라스팅 자체의 과정은 유전자 발현30의 차이를 도입하고 변형되고 두꺼운 이차 세포벽을 가진 조직은 치료하기가 악명 이어 있다. 더욱이, 식물의 많은 발달 공정은 기계적으로 전송된 신호에 의존하는 것으로 가정되므로 세포벽의 무결성이 가장 중요하다31. 핵의 수준에서 작동하여 세포 격리를 우회하기 위해 바로 가기를 사용하는 두 가지 방법은 형광 활성화 핵 선별 (FANS) 및 특정 세포 유형 (INTACT)에 태그 된 핵의 분리입니다. TRAP에서와 같이, 그(것)들은 핵을 표시하기 위하여 세포 유형 특정 프로모터를 이용하고, 그 후에 분류를 통해 농축되거나, 각각8,,15. 이 모든 접근을 위한 중요한 도전은 조직에 있는 세포의 부분 집합에서 충분한 RNA 물자를 얻는 것입니다. TRAP이 셀룰러 RNA의 일부만 캡처하므로 샘플 수집은 상당한 병목 현상입니다. 따라서 낮은 입력 량에서 고품질 데이터를 생성하려면 특히 중요한 라이브러리 준비 프로토콜이 필요합니다.
설립 이래, TRAP은 DNA 마이크로어레이와 병용되어 사용되거나, 시퀀싱 비용이 최근 몇 년 동안 현저히 감소함에 따라, RNA-seq10,,32,,33. 연구 질문의 무리는 이미 사블락 등에서 검토로 해명되었습니다34. 우리는 기술이 특정 세포 모형을 표적으로 하기 위하여 다른 프로모터를 결합할 때 아주 다재다능하기 때문에 앞으로 더 많은 보고가 따를 것이라는 점을 확신합니다. 결국, 이것은 유도 할 수있는 방법으로도 수행 될 것이며, 많은 생물학적 및 비생물적 스트레스 요인에 대한 식물의 반응을 조사하는 것으로 결합 될 수 있습니다. 또한, 안정적인 형질전환라인을 사용할 수 없는 곳에서는, 털이 많은 뿌리 발현 시스템도 토마토와메디아고(35,,36)에서TRAP을 수행하는 데 성공적으로 사용되었다.
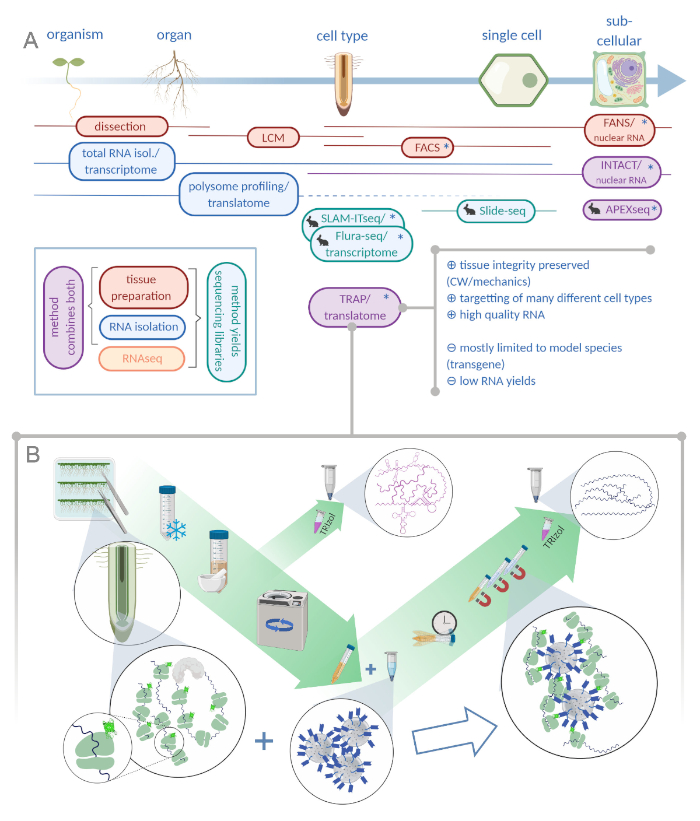
그림 1: 리보솜 친화성 정제(TRAP)를 번역하는 것은 “오믹스” 분석 포트폴리오를 보완합니다. A. 분석 정밀도의 증가 수준, 단일 세포 또는 심지어 세포 이하 의 해상도는 이들의 방법 또는 조합의 과다에 의해 달성 될 수있다. 이 계획은 식물과 동물 분야에서 현재 사용 가능한 도구의 개요를 제공합니다. 세포 결정에서 조직 수집은 LCM 또는 FACS와 같은 프로토콜에 의해 달성 될 수 있으며, 표준 전사체 또는 폴리섬 프로파일링 / 트랜스 라토메 분석과 결합됩니다. TRAP 과 INTACT는 에피토프 태깅을 기반으로 조직 캡처 및 RNA 격리를 모두 통합합니다. 그러나, INTACT 는 세포 핵만을 샘플링하고, 따라서, 전사체 분석의 특별한 경우를 구성한다. 작은 토끼 아이콘은 동물 분야에서 새로 개발 된 방법을 표시 : SLAM-ITseq 및 Flura-seq는 허용 효소를 발현하는 세포에서 수정 된 우라실 염기를 가진 초기 RNAs의 대사 지색에 의존하는 반면, Slide-seq는 세포 범위에서 위치 정보를 제공하는 DNA 바코드가있는 코팅 된 유리 슬라이드를 사용합니다. 근접 라벨링 접근법은 APEX-seq에서 특정 세포 외 구획에서 RNA를 샘플링하는 방법을 따릅니다. 특히, 증가 된 해상도는 종종 형질전환 물질 (별표)의 생성을 필요로하고 이러한 방법은 따라서 주로 모델 종에 사용된다. TRAP은 특히 CW 매트릭스에서 방출하기 어려운 세포종뿐만 아니라 세포벽(CW) 또는 역학 신호와 관련된 식물 과학 연구에 적합합니다. B. TRAP 절차의 상세한 습식 실험실 단계: GFP 태그가 달린 리보좀 단백질을 구별된 세포 유형(예: 뿌리 내피)에서 발현하는 모종은 7일 동안 페트리 접시에서 재배되고 스냅 동결에 의해 수확된 뿌리 재료입니다. 총 RNA 제어 샘플은 원심 분리를 통해 파편을 펠릿하기 전에 균질화된 원유 추출물로부터 수집됩니다. 자기 항-GFP 비드는 면역 침전을 수행하기 위해 클리어된 추출물에 첨가된다. 배양 및 3개의 세척 단계 후, 폴리솜 관련 RNA(TRAP/폴리솜 RNA)는 페놀-클로로포름 추출을 통해 직접 수득된다. LCM : 레이저 캡처 미세 해부, FACS / 팬 : 형광 활성화 세포 / 핵 선별, APEX-seq: 엔지니어링 된 아스코르브 인 과산화제에 기초한 방법, INTACT: 특정 세포 유형에 태그 된 핵의 격리, SLAM-ITseq: THIOL(SH)-조직내 RNA의 대사 시퀀싱에 대한 연결 알킬레이션, 플루라-세크: 플루라-세크: 플루라-세크: 플루오라실 라벨RNA 시퀀싱(Biorender.com 이쪽을 클릭하십시오.
이 문서의 목표는 TRAP 메서드에 대한 자세한 설명을 제공하고 중요한 단계를 강조 표시하고 가능한 라이브러리 준비 방법에 대한 지침을 제공하는 것입니다.
일반 TRAP 실험은 본질적으로 다음 단계로 구성됩니다 (도 1B참조) ( (1) 리보솜 태깅 구조의 복제, 형질 전환 라인 생산 및 선택, 씨앗의 성장 및 벌킹, 살균 및 도금, 스트레스 적용 / 치료 (선택 사항) 및 조직 수확을 포함한 식물 재료의 준비; (2) 조 추출물의 조직 균질화 및 개간, 비드 세척 및 면역정화, 및 세척 단계를 포함하는 면역화; (3) RNA 추출 및 품질 평가; (4) 도서관 준비.
애기장대 뿌리는 모델 플랜트37,,38으로도입된 이래 식물 개발을 연구하는 모델 시스템이다. 여기서 TRAP의 적용은 식물 측면 루트 개발의 맥락에서 전시된다. 식물에서 전체 루트 시스템의 축적은이 프로그램의 실행에 의존하므로 유기체(39)의생존에 매우 중요합니다. 애기장대에서,측면 뿌리는 자일렘 혈관 옆에 상주하는 pericycle 조직에서 유래하고 따라서 자일렘 극 페리사이클이라고합니다 (XPP; 그림 2C참조)40. 뿌리 안쪽 깊숙이 위치한 일부 XPP 세포는 설립자 세포 정체성을 얻고 국소 호르몬 트리거에 따라 팽창과 반대로41을나누어 증식하기 시작합니다. 그러나 단단한 세포벽 매트릭스가 존재하기 때문에이 과정은 주변 조직에 기계적 응력을 가합니다. 특히, 횡근근성장축(42,,43,,4244)의방식이기 때문에, 과도내피증이 영향을 받는다. 실제로, 새로 형성된 프리모르듐은 지나치게 내피세포(도2C2)를통해 성장해야 하는 반면 피질과 표피 세포는 프리모르듐이 마침내45,,46으로나타난다. 우리의 실험실에 있는 최근 일은 내분비가 pericycle에 있는 확산을 수용하기 위하여 적극적으로 기여하고 있다는 것을 보여주었습니다. 내피 호르몬 신호의 표적 차단은 XPP세포(47)에서의제1 분열조차도 억제하기에 충분하다. 따라서, pericycle-endodermis 통신은 애기장대에서측근 발달을 위한 아주 초기 체크포인트를 구성합니다. 그러나 이 크로스토크가 어떻게 수행되는지는 알 수 없습니다. 이 수수께끼를 풀기 위해 XPP 및 내피 세포를 대상으로 하는 TRAP-seq 접근법을 선택했습니다. 측면 루트 프로그램에서 세포를 풍부하게하기 위해, 우리는 외인성으로 auxin 아날로그를 적용하여 호르몬 트리거를 모방 (1-naphthaleneacetic 산, NAA)48,이는 동시에 일시적으로 측면 루트 형성의 초기 단계를 해결 허용.
Protocol
Representative Results
Discussion
RPL18 현지화 패턴 검증
TRAP 실험에서 데이터의 잘못된 해석을 방지하는 데 중요한 것은 태그가 지정된 리보소말 소단위의 적절한 발현 패턴입니다. 따라서, RPL18에 에피토프 태그로서 GFP의 혼입은 매우 우아하게 동일한 조직에서 다각성 분획의 원하는 발현 패턴 및 연속적으로, 풀다운의 검증을 가능하게 한다. 적절한 프로모터 패턴을 보장하기 위한 보다 침습적인 접근법은 Jiao 및 M…
Divulgations
The authors have nothing to disclose.
Acknowledgements
우리는이 프로젝트의 초기 단계에서 중요한 전문가 조언을 취리히 유전 다양성 센터의 장 클로드 Walser에 감사드립니다. Vermeer 연구소의 작업은 SNF 교수 직교 보조금(PP00P3_157524)과 JEMV에 수여된 스위스 국립 과학 재단(SNSF)의 R’EQUIP 장비 보조금(316030_164086)에 의해 지원되었습니다.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

