Bir Hücre Tipine Özel Ölçekte Arabidopsis thaliana Kök Gelişimini Araştırmak Için Ribozom Affinity Arınma (TRAP) Çevirisi
Summary
Ribozom yakınlık arınması (TRAP) çevirisi, organ ve dokuların en az işlenmesi yle gelişim programlarını inceleme olanağı sunar. Protokol, yeşil floresan protein (GFP) etiketli ribozomal alt ünite ile hedeflenen hücrelerden yüksek kaliteli RNA verir. QRT-PCR veya RNA-seq gibi alt akış analiz araçları doku ve hücre tipine özgü ifade profillerini ortaya çıkarır.
Abstract
Bu makalede, ribozom afinite arınma (TRAP) yöntemi ve ardışık optimize edilmiş düşük girişli kitaplık hazırlama yöntemi ile farklı Arabidopsis thaliana kök hücre tiplerinden translatom verileri elde etmek için uygulamalı talimatlar veriyoruz.
Başlangıç malzemesi olarak, yeterli organizatörlerin kullanımı ile gfp etiketli ribozomal protein RPL18’i hücre tipine özgü bir şekilde ifade eden bitki hatlarını kullanırız. Önce immünpurifikasyon ve RNA ekstraksiyon, doku, doku bütünlüğünü korur ve aynı anda yüksek zamansal çözünürlük ile zaman serisi çalışmaların yürütülmesine olanak sağlar dondurulmuş snap. Özellikle, hücre duvarı yapıları bozulmadan kalır, bu da farklı hücre popülasyonlarını izole etmek için protoplasting dokuya dayanan floresan-aktive hücre tasnisi temelli yaklaşımlar gibi alternatif prosedürlerde büyük bir dezavantajdır. Ayrıca, yüksek kaliteli RNA elde edilmesine olanak sağlayan lazer yakalama mikrodiseksiyon tabanlı teknikler gibi doku fiksasyonu gerekli değildir.
Ancak, hücrelerin alt popülasyonlarından örnekleme ve sadece polizom ilişkili RNA izole ciddi RNA verimleri sınırlar. Bu nedenle, RNA-seq tarafından başarılı veri toplama için yeterince hassas kütüphane hazırlama yöntemleri uygulamak gereklidir.
Trap, hücre duvarıyla ilgili ve mekanik sinyal yolları içerdiğinden, birçok gelişimsel süreç hücre duvarı yla ilgili olduğu için bitki araştırmaları için ideal bir araç tır. Organizatörlerin belirli hücre popülasyonlarını hedeflemek için kullanımı, organ ve tek hücre düzeyi arasındaki boşluğu köprülemektedir ve bu da çok az çözünürlük veya çok yüksek maliyetlerden muzdariptir. Burada, lateral kök oluşumunda hücre-hücre iletişimini incelemek için TRAP uyguluyoruz.
Introduction
Yeni nesil sıralama tekniklerinin giderek artan uygulamasıyla, gelişim biyolojisinde uzamsal çözünürlük artırılabilir. Çağdaş çalışmalar, tek hücreli seviye,1,2,3,,4değilse, dokuların özel hücre tiplerine kadar incelenmesini amaçlamaktadır. Bu amaçla, son elli yıl içinde farklı yöntemlerbir bolluk geliştirilmiştir (Şekil 1Abakınız )5,6,,7,8,9,10,11,12,13,14,15.
Bitki bilimindeki pek çok araç, hayvan araştırmalarında öncülük eden tekniklerin adaptasyonları olmuştur. Burada ayrıntılı olarak sunduğumuz yöntem için durum böyle değildir. 2005 yılında, protein çevirisi güçlü bir arka plan ile donatılmış, Bailey-Serres Lab sonraki yakınlık saflaştırma16için ribozomal proteinleri mühendisi yola çıktı. Böylece, bir sakaroz gradyan ile ultracentrifugation dayalı ve 1960’lardan beri ribozomçevirme değerlendirmek için kullanılan zaman alıcı ve emek yoğun polizom profilleme, önlemek olabilir17,18. Yöntem o zamandan beri çevirisel ribozom afinite arınma (TRAP)16olarak anılacaktır. Bitkilerde başarılı translatome çalışmalardan sonra, Heiman ve ark. hayvanlar için TRAP adapte19 ve diğerleri maya20, Drosophila21, Xenopus22 ve zebra balığı23,24.
Model sisteminin genetik modifikasyonu TRAP için bir ön koşul olmasına rağmen, hangi türler genetik dönüşüm için uygun uygulama sınırlar, bir aynı anda özel ilgi ve bozulmamış doku / organ25 izole etmek için son derece zor hücrelerin alt kümeleri hedef bu itiraz koşum olabilir (örneğin, bir fare beyin veya enfekte bitki dokusunda mantar hyphae yüksek dallı dendritik hücreler). Bitkilerde, tüm hücreler hidrostatik iskelet26temelini oluşturan hücre duvarları ile yerinde tutulur. Bu matris bir bitki hücresi serbest etmek için, bilim adamları ya fiziksel lazer yakalama mikrodizeksiyon (LCM)27 veya hücre duvarları28enzimatik sindirim gerçekleştirilen yoluyla çevresindeki doku hücre kesip var . İkinci hücreler arasında, sözde protoplastlar, ilgi popülasyonu floresan etiketli ve floresan aktif hücre sıralama (FACS)7ile ayrılabilir. LCM genellikle sabit ve balmumu gömülü bir örnek gerektirir, hangi sonuçta onun RNA kalitesini bozar29. FACS tabanlı yöntemler yüksek kaliteli RNA verim, ancak protoplasting kendisi süreci gen ekspresyonu farklılıkları tanıttı30 ve modifiye ve kalın ikincil hücre duvarları ile dokuların tedavi etmek için kötü üne sahiptir. Ayrıca, bitkilerdeki birçok gelişimsel sürecin mekanik olarak iletilen sinyallere dayandığı varsayılır ve bu nedenle hücre duvarının bütünlüğü büyük önem taşımaktadır31. Çekirdek seviyesinde hareket ederek hücre izolasyonundan maksatlamak için kısayol kullanan iki yöntem, floresan tarafından aktive edilmiş nükleer sıralama (FANS) ve belirli hücre tiplerinde etiketlenmiş çekirdeklerin izolasyonudur (BOZULMASIN). TRAP olduğu gibi, onlar çekirdekleri işaretlemek için hücre tipine özgü organizatörleri kullanmak, daha sonra sıralama yoluyla zenginleştirilmiş olsun veya aşağı çekmek, sırasıyla8,15. Tüm bu yaklaşımlar için önemli bir sorun bir doku hücrelerinin alt kümelerinden yeterli RNA malzeme elde etmektir. TRAP hücresel RNA’ların sadece bir kısmını ele geçirirken, numune toplama önemli bir darboğazdır. Bu nedenle, düşük giriş tutarlarından yüksek kaliteli veri üretmek için özellikle hassas kitaplık hazırlama protokolleri gereklidir.
Kuruluşundan bu yana, TRAP ya DNA mikrodizileri ile birlikte kullanılmıştır ya da, sıralama maliyetleri son yıllarda önemli ölçüde düştü gibi, RNA-seq10,32,33. Sablok ve ark.34’teincelenmiş çok sayıda araştırma sorusu zaten açıklığa kavuşturulmuştur. Belirli hücre türlerini hedeflemek için farklı organizatörleri birleştirerek teknik çok yönlü olduğu için önümüzdeki yıllarda daha fazla rapor takip edeceğine eminiz. Sonunda, Bu indüklenebilir bir şekilde bile yapılacak, ve birçok biyotik ve abiyotik stres faktörlerine bitkinin reaksiyonu probing ile kombine edilebilir. Ayrıca, istikrarlı transgenik hatları mevcut değildir, kıllı kök ekspresyon sistemleri de başarıyla domates ve medicago35,36TRAP gerçekleştirmek için kullanılmıştır.
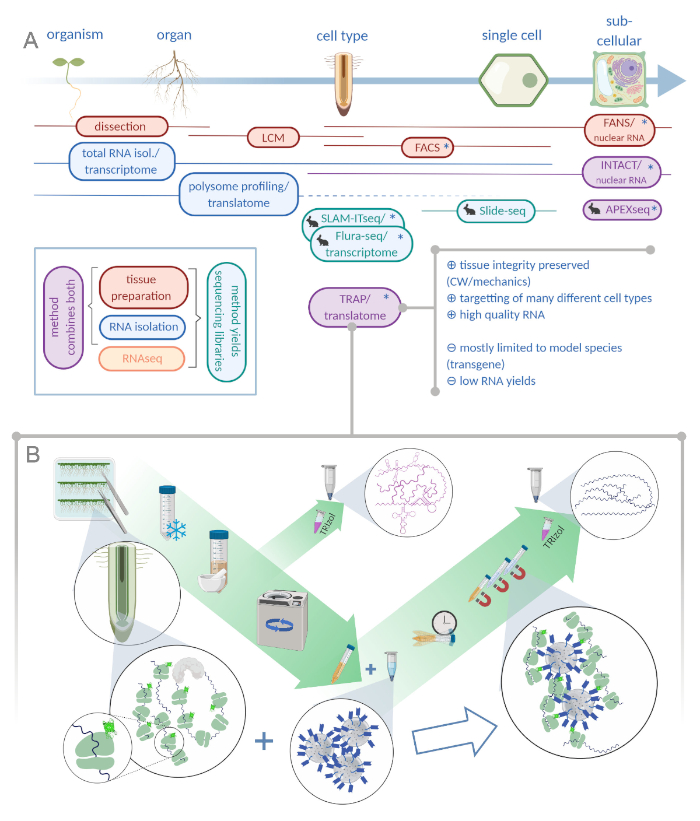
Şekil 1: Ribozom afinite arınma (TRAP) çevirisi “omiks” analiz portföyünü tamamlar. A. Analitik hassasiyet düzeylerini artırmak, aşağı tek hücreli hatta hücre altı çözünürlüğü yöntemleri veya bunların kombinasyonları bir bolluk ile elde edilebilir. Şema bitki ve hayvan alanında şu anda mevcut araçlar alabilen bir genel bakış sağlar. Hücresel çözünürlükte doku toplama lcm veya FACS gibi protokoller ile elde edilebilir, daha sonra standart transkripsime veya polizom profilleme / translatome analizi ile birleştiğinde. TRAP ve INTACT, epitop etiketlemeye dayalı olarak hem doku yakalama hem de RNA izolasyonu entegre ederler. Ancak, INTACT sadece hücre çekirdeklerini örnekler ve bu nedenle, transkripsiyon analizi özel bir durum oluşturur. Küçük bir tavşan simgesi hayvan alanında yeni geliştirilen yöntemleri işaretler: SLAM-ITseq ve Flura-seq izin veren enzim ifade hücrelerde değiştirilmiş uracil üsleri ile yeni doğan RNA metabolik targetting güveniyor iken, Slide-seq hücresel aralığında konumsal bilgi sağlayan DNA barkodları ile kaplı cam slayt kullanır. APEX-seq’da belirli hücre altı bölmelerde RNA örneklemesi için yakınlık etiketleme yaklaşımı izlenir. Özellikle, artan çözünürlük genellikle transgenik malzeme (yıldız) üretimi gerektirir ve bu nedenle bu yöntemler ağırlıklı olarak model türler için kullanılır. TRAP özellikle hücre duvarı (CW) veya mekanik sinyalizasyon ve CW matrisserbest bırakmak zor hücre türleri içeren bitki bilimi çalışmaları için uygundur. B. TRAP prosedürünün ayrıntılı ıslak laboratuvar adımları: Farklı hücre tiplerinde (örn. kök endodermisi) GFP etiketli ribozomal proteini ifade eden fideler Petri kaplarında yedi gün boyunca yetiştirilir ve kök malzemesi yapışma donma ile hasat edilir. Toplam RNA kontrol numunesi, santrifüj yoluyla enkaz peletleme önce homojenize ham ekstresi toplanır. Manyetik anti-GFP boncukimmünenfazla yapmak için temizlenmiş ekstrese eklenir. Kuluçka ve üç yıkama adımından sonra polizomla ilişkili RNA (TRAP/polizom RNA) doğrudan fenol-kloroform ekstraksiyonu ile elde edilir. LCM: lazer yakalama mikrodiseksiyon, FACS / FANLAR: floresan aktif hücre / nükleer sıralama, APEX-seq: mühendislik askorbate peroksidaz dayalı yöntem, INTACT: belirli hücre tiplerinde etiketli çekirdeklerin izolasyonu, SLAM-ITseq: dokuda RNA metabolik sıralama için thiol(SH) bağlı alkylation, Flura-seq: fluorourasil etiketli RNA sıralama (Biorender.com ile oluşturulmuş) Bu rakam daha büyük bir sürümünü görmek için lütfen buraya tıklayın.
Bu makalenin amacı, TRAP yönteminin ayrıntılı bir açıklamasını sağlamak, kritik adımları vurgulamak ve olası bir kitaplık hazırlama yöntemi için rehberlik sağlamaktır.
Genel bir TRAP deneyi temelde aşağıdaki adımlardan oluşacaktır (ayrıca bkz. Şekil 1B):(1) Ribozom etiketleme yapısının klonlanması, transgenik hat üretimi ve seçimi, tohumların büyümesi ve şişirilmesi, sterilizasyon ve kaplama, stres uygulaması/tedavisi (isteğe bağlı) ve doku hasadı; (2) doku homojenizasyonu ve ham ekstresi temizlenmesi, boncuk yıkama ve immünpurifikasyon ve yıkama adımları da dahil olmak üzere immünsafifikasyon; (3) RNA çıkarma ve kalite değerlendirmesi; ve (4) kütüphane hazırlama.
Arabidopsis kökü bir model bitki olarak piyasaya beri bitki geliştirme çalışma için bir model sistemi olmuştur37,38. Burada TRAP uygulaması bitki yanal kök gelişimi bağlamında sergilenir. Bitkilerde, tüm kök sisteminin birikimi bu programın yürütülmesine dayanır ve bu nedenle organizmanın hayatta kalması için çok önemlidir39. Arabidopsis’telateral kökler ksilem damarlarının yanında bulunan pericycle dokusundan kaynaklanır ve bu nedenle ksilem direk pericycle (XPP; bkz. Şekil 2C)40olarak adlandırılır. Kökün derinliklerinde bulunan bazı XPP hücreleri, kurucu hücre kimliği kazanır ve yerel bir hormonal tetikleyici üzerine, anticlinal41’işişirerek ve bölerek çoğalmaya başlar. Ancak, sert bir hücre duvarı matris varlığı nedeniyle, bu süreç çevre dokular üzerinde mekanik stres uygular. Özellikle, örten endodermis etkilenir, yanal kök büyüme ekseni şekilde olduğu gibi42,43,44. Gerçekten de, yeni oluşan primordium örten endodermis hücre yoluyla büyümek zorunda kalacak(Şekil 2C2) korteks ve epidermis hücreleri sadece sonunda ortaya çıkarmak için primordium için kenara itilirise 45,46. Laboratuarımızda yapılan son çalışmalar, endodermisin peridöngüsündeki çoğalmaya aktif olarak katkıda bulundugunu göstermiştir. Endodermal hormonal sinyalhedefli engelleme XPP hücrelerinde bile ilk bölümü inhibe etmek için yeterlidir47. Bu nedenle, pericycle-endodermis iletişim Arabidopsislateral kök gelişimi için çok erken bir kontrol noktası oluşturmaktadır. Ancak bu çapraz konuşmanın nasıl yapıldığı bilinmemektedir. Bu gizemi çözmek için XPP ve endodermal hücreleri hedeflemek için TRAP-seq yaklaşımını seçtik. Lateral kök programında hücreler için zenginleştirmek için, biz ekseksen bir auxin analog uygulayarak hormonal tetikleyici taklit (1-naftaneasetik asit, NAA)48, hangi aynı zamanda zamansal lateral kök oluşumunun ilk aşamasıçözmek için izin.
Protocol
Representative Results
Discussion
RPL18 yerelleştirme deseni doğrulaması
Herhangi bir TRAP deneyinden elde edilen verilerin yanlış yorumlanmasını önlemek için çok önemli olan, etiketli ribozomal alt birimin uygun ifade desenidir. Bu nedenle, RPL18 bir epitop etiketi olarak GFP dahil çok zarif istenilen ifade deseni doğrulama ve ardışık, aynı dokudan polizom fraksiyonu çekme sağlar. Uygun organizatör desenleri sağlamak için daha invaziv yaklaşımlar Jiao ve Mayerowitz 2010, GUS boyama gerektirir ve Tian ve ark 20…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Zürih Genetik Çeşitlilik Merkezi’nden Jean-Claude Walser’a bu projenin erken evresinde önemli uzman tavsiyeleri için teşekkür ederiz. Vermeer laboratuvarındaki çalışmalar, JEMV’ye verilen İsviçre Ulusal Bilim Vakfı’ndan (SNSF) SNF Profesörlük hibesi (PP00P3_157524) ve R’EQUIP ekipman hibesi (316030_164086) tarafından desteklendi.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

