Visualização de Proteínas de Reparação de DNA Interação por Imunofluorescência
Summary
Após danos no DNA, as células humanas ativam caminhos essenciais de reparação para restaurar a integridade de seu genoma. Aqui, descrevemos o método de imunofluorescência indireta como um meio de detectar proteínas de reparação de DNA, analisar seu recrutamento espacial e temporal e ajudar a interrogar a interação proteína-proteína nos locais de danos ao DNA.
Abstract
As células mamíferas são constantemente expostas a produtos químicos, radiações e subprodutos metabólicos que ocorrem naturalmente, que criam tipos específicos de insultos de DNA. Agentes genotóxicos podem danificar a espinha dorsal do DNA, quebrá-la ou modificar a natureza química das bases individuais. Após insulto de DNA, vias de resposta a danos de DNA (DDR) são ativadas e proteínas envolvidas no reparo são recrutadas. Uma infinidade de fatores estão envolvidos na detecção do tipo de dano e na ativação da resposta adequada ao reparo. A falha em ativar e recrutar corretamente fatores de DDR pode levar à instabilidade genômica, que está por trás de muitas patologias humanas, incluindo o câncer. Estudos de proteínas DDR podem fornecer insights sobre a resposta de medicamentos genotóxicos e mecanismos celulares de resistência a medicamentos.
Existem duas principais formas de visualizar proteínas in vivo: observação direta, marcando a proteína de interesse com uma proteína fluorescente e seguindo-a por imagem viva, ou imunofluorescência indireta em amostras fixas. Embora a visualização de proteínas fluorescentes marcadas permita um monitoramento preciso ao longo do tempo, a marcação direta em N ou C-terminus pode interferir na localização ou função da proteína. A observação de proteínas em sua versão endógena não modificada é preferida. Quando as proteínas de reparação de DNA são recrutadas para o insulto do DNA, sua concentração aumenta localmente e formam grupos, ou “focos”, que podem ser visualizados por imunofluorescência indireta usando anticorpos específicos.
Embora a detecção de focos proteicos não forneça uma prova definitiva de interação direta, a co-localização de proteínas nas células indica que elas se reagrupam ao local do dano e podem informar sobre a sequência de eventos necessários para formação complexa. A análise cuidadosa da sobreposição espacial de focos em células expressas do tipo selvagem ou versões mutantes de uma proteína pode fornecer pistas preciosas sobre domínios funcionais importantes para a função de reparação de DNA. Por último, a co-localização de proteínas indica possíveis interações diretas que podem ser verificadas por co-imunoprecipitação nas células, ou retirada direta usando proteínas purificadas.
Introduction
Células humanas são constantemente expostas a uma variedade de DNA danificando agentes de várias origens. Fontes exógenas consistem principalmente em exposição a radiações, produtos químicos (incluindo agentes quimioterápicos e alguns antibióticos), e vírus, enquanto as principais fontes endógenas incluem erros na replicação do DNA e estresse oxidativo. Os efeitos diretos da exposição genotoxica podem variar de uma base modificada a uma quebra de duplo fio de DNA potencialmente letal (DSB), dependendo do estresse e da dose de exposição. Em última análise, danos de DNA não reparados ou mal reparados podem levar ao acúmulo de mutações, rearranjos genômicos, instabilidade do genoma e eventualmente levar à carcinogênese1. As células mamíferas desenvolveram caminhos complexos para reconhecer tipos específicos de dano de DNA2,3 e repará-los em tempo hábil, sincronizados com a progressão do ciclo celular.
A radiação ionizante (IR) danifica a dupla hélice do DNA e cria quebras de dois fios (DSBs), uma das formas mais deletérias de dano de DNA. O complexo MRN (MRE11, RAD50, NBS1) funciona como um sensor de extremidades de DNA e ativa a proteína quinase ataxia telangiectasia mutada (ATM)4,5. Após a ativação inicial do ATM por extremidades de DNA, o ATM desencadeia uma cascata de eventos de DDR no local do rompimento, iniciando com um evento-chave, a fosforilação da variante histona H2AX6. Fosforilação H2AX sobre resíduo S139 ativa-o em γH2AX, abrangendo regiões até megabases em torno da lesão de DNA6,,7,,8,9. Este evento aumenta a acessibilidade ao DNA, levando ao recrutamento e acúmulo de outras proteínas de reparação de DNA7. Como o γH2AX é abundante e especificamente induzido em DSBs circundantes, ele pode ser facilmente visualizado usando anticorpos específicos, e é comumente usado como um marcador substituto para DSBs no campo de reparação de DNA. Uma vez que a ruptura é sinalizada, as células ativam suas vias de reparação de DNA e processam o dano de DNA. A proteína MDC1 (mediadora da proteína de ponto de verificação de dano de DNA 1) liga diretamente γH2AX10,interage com o ATM11 e também com NBS112,13. Contribui para aumentar a concentração do complexo de MRN no DSB e iniciar um ciclo positivo de feedback atm. γH2AX é rapidamente removido uma vez que a ruptura é reparada, consequentemente, permitindo o monitoramento da liberação DSB. Seguida pela microscopia, a diminuição do γH2AX ao longo do tempo fornece uma medição indireta de quebras residuais e eficiência de reparação de DNA.
As células eucarióticas podem reparar DSBs por várias vias, sendo as duas principais não homólogos a junção final (NHEJ) e recombinação homólogo (HR)(Figura 1). NHEJ essencialmente liga extremidades de dupla cadeia de DNA sem o uso de homologia estendida e opera durante todo o ciclo celular14,15. O RH torna-se predominante durante as fases S e G2, e é reprimido de outra forma, uma vez que requer uma cromátide irmã como modelo homólogo para reparo14,16. A escolha do caminho entre nhej e RH não depende apenas da proximidade física da cromátide irmã, mas também da extensão da ressecção final de DNA17,que inibe o NHEJ.
O reparo de DSB dependente de ecologia inicia-se pela degradação nucleolítica do fio de 5′ das extremidades de ruptura para gerar caudas de DNA de 3′ de fios únicos (ssDNA), um processo chamado de ressecção de 5′-3′. O complexo MRN inicia a ressecção final do DNA e a ressecção adicional é processada em combinação com BLM/EXO1 (proteína da síndrome de Bloom/exonuclease 1) ou BLM/DNA2 (replicação de DNA atp-dependente helicase/nuclease)18,19,20,21,22. A ressecção final do DNA é aprimorada pela CtIP (Proteína Interagindo com ADCBP) através de sua interação direta com o complexo MRN23 e recrutamento do BRCA1 (proteína de suscetibilidade tipo 1 do câncer de mama)24,25. Proteína de replicação A (RPA) se liga prontamente ao ssDNA exposto e, em seguida, é deslocada pela proteína recombinase RAD51 para formar um filamento nucleoproteína que catalisa a busca homólogo e a invasão de fios26,,27,28.
O início da ressecção é um passo crítico para a escolha do caminho de reparo. Uma vez iniciada a ressecção, as extremidades do DNA tornam-se substratos pobres para ligação pelo heterodimer Ku70/Ku80 (componente da via NHEJ) e as células são comprometidas com o HR17,,29,30. O heterodimer Ku70/Ku80 se liga aos fins do DSB, recrutando DNA-PKcs e p53 Binding Protein 1 (53BP1)29,30. O 53BP1 atua como uma barreira à ressecção no G1, bloqueando assim o RH ao promover a NHEJ31,32, mas é removido de forma dependente do BRCA1 na fase S, permitindo, consequentemente, que ocorra a ressecção33,34. Portanto, 53BP1 e BRCA1 desempenham papéis opostos no reparo do DSB, sendo 53BP1 um facilitador NHEJ enquanto o BRCA1 atua permitindo que as quebras sejam reparadas pelo RH.
Em laboratório, a formação de DSB pode ser induzida por radiação ionizante (IR). Enquanto este exemplo utiliza uma alta dose de 4 Gy, 1 Gy e 2 Gy também criam uma quantidade significativa de DSBs, adequado para a análise da formação de focos por proteínas abundantes. É importante notar que o tipo e a dose de radiação utilizada podem levar a diferentes lesões no DNA e na célula: enquanto o IR induz DSBs, também pode causar quebras de fios únicos ou modificação de base (ver35,36 para uma referência sobre transferência de energia linear de irradiação (LET) e tipo de dano de DNA). Para determinar a cinética da formação de focos induzidos por radiação ionizante (IRIF) e sua desobstrução, que indicam reparação dos danos e reversão da formação ativada de DDR8,,9,,37,,38, os focos podem ser monitorados em diferentes pontos de tempo após a radiação ionizante. O tempo de ativação e liberação de todas as principais proteínas de dano de DNA é conhecidocomo 39, e muitos são investigados como marcadores substitutos de eventos-chave. Por exemplo, pRPA, que possui alta afinidade com ssDNA é usado como substituto da ressecção de quebra, proteínas MRN (MRE11, RAD50, NBS1) e exonucleases também podem ser usadas para avaliar a eficiência da ressecção. Enquanto rad51, BRCA1, BRCA2 (proteína de suscetibilidade ao câncer de mama tipo 2) e PALB2 (parceiro e localizador do BRCA2) podem ser monitorados para avaliar a eficiência do RH, a presença das proteínas Ku ou 53BP1, são utilizados como marcadores do NHEJ (Figura 1).
À medida que as proteínas das máquinas de reparação de DNA recrutam umas às outras para a quebra e montagem em superconsetos, as interações DNA-proteína e proteína-proteína podem ser inferidas seguindo sua localização individual ao longo do tempo e analisando a co-localização de proteínas, conforme visualizado por sinais sobrepostos na célula40,,41,,42. Nas linhas celulares, a introdução de mutações pontuais ou exclusão em genes específicos de reparação de DNA, seja através da edição de genomas, ou pela superexpressão de mutantes à base de plasmídeos, permite a investigação de resíduos específicos e seu possível papel no reconhecimento de danos de DNA (por exemplo, co-localização com γH2AX) ou montagem complexa (co-localização com outra, ou várias proteínas), bem como seu impacto na reparação do DNA. Aqui, usamos a imunofluorescência indireta como meio para investigar a formação e resolução de DSBs seguindo os focos γH2AX ao longo do tempo. Também apresentamos um exemplo de formação de focos e análise de co-localização por um dos principais players no reparo de DSB: p53 Binding Protein 1 (53BP1)32. Como mencionado anteriormente, 53BP1 é considerado central para a escolha da via de reparação de DNA. Após o acúmulo de 53BP1 e sua co-localização com γH2AX fornece informações preciosas sobre a fase de ciclo celular, acúmulo de danos de DNA e via usada para reparar DSBs. O objetivo da imunolocalização indireta é avaliar a eficiência da reparação de danos do DNA nas linhas celulares, seguindo o IR como neste estudo, ou após a exposição a várias tensões na célula, desde o cruzamento de DNA até o bloqueio do garfo de replicação (uma lista de agentes nocivos ao DNA é fornecida na Tabela 1).
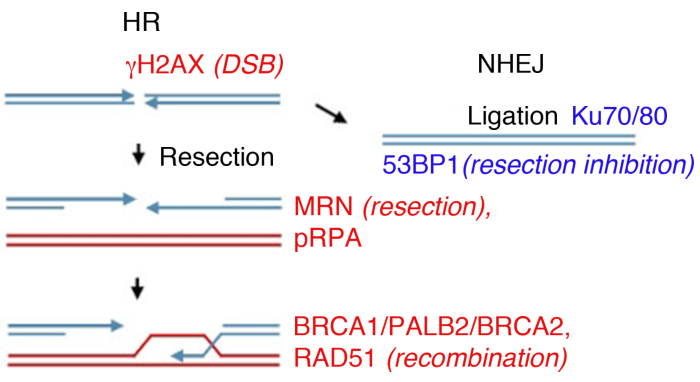
Figura 1: DNA quebra o fio duplo (DSB) caminhos de reparo.
O reparo do DSB envolve duas vias principais: Recombinação Homologous (RH, à esquerda) e End-Joining Não Homólogo (NHEJ, à direita). Após a quebra, as proteínas são ativadas para marcar a quebra (γH2AX), participar da ressecção final (MRN), revestir o ssDNA ressecado (pRPA), promover a recombinação (BRCA1, PALB2, BRCA2, RAD51) ou limitar a ressecção e promover o NHEJ (53BP1). Outras proteínas participam da reparação de danos, mas as proteínas listadas são rotineiramente seguidas por imunofluorescência indireta. Clique aqui para ver uma versão maior desta figura.
| Agente prejudicial ao DNA | Mecanismo de ação | Dose recomendada |
| raios-X | Radiação Formação de quebras duplas com alguns efeitos celulares descontrolados |
1-4 Gy |
| 36 Íons ar | Radiação Formação de quebras duplas encalhadas |
270 keV/μm |
| α-partículas | Radiação Formação de quebras duplas encalhadas |
116 keV/μm |
| Bleomicina | Inibidor da síntese de DNA | 0.4-2 μg/mL |
| Camptothecin | Inibidor de topoisomerase I | 10-200 nM |
| Cisplatina | Agente alquilante (induzir intrastras e crosslinks) |
0,25-2 μM |
| Doxorrubicina | Agente de intercalação Inibidor de topoisomerase II |
10-200 nM |
| Etoposide | Inibidor de topoisomerase II | 10 μM |
| Hidroxiuréia | Inibidor da síntese de DNA (por reductase ribonucleotídeo) |
10-200 μM |
| Metano-metil | Agente alquilante | 0,25-2 mM |
| Mitomicina C | Agente alquilante | 0,25-2 μM |
| Luz ultravioleta (UV) | Formação de dimers de timmidina (gerando distorção da cadeia de DNA) |
50-100 mJ/cm2 |
Tabela 1: Agentes genotóxicos. Exemplos de agentes nocivos do DNA, seu mecanismo de ação e os danos induzidos com base na concentração de trabalho sugerida.
Protocol
Representative Results
Discussion
A análise do tempo e eficiência do reparo de danos no DNA por microscopia tem se mostrado essencial para entender como funciona o maquinário de reparação de DNA, em células normais e em patologias humanas como o câncer.
O desenvolvimento de anticorpos específicos que permitem a detecção de proteínas ativadas em sua versão fosfoilada (como γH2AX, pRPA, pRAD50 e outros10,23,39,<s…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Este trabalho foi apoiado por uma bolsa da Fundação De San Antonio. O Mays Cancer Center é apoiado pelo núcleo de apoio ao centro de câncer NCI P30 CA054174. Gostaríamos de agradecer a Stephen Holloway por sua ajuda para fornecer os reagentes, e o laboratório Sung para fornecer espaço e capacidade de microscopia.
Materials
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
References
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600 (2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757 (2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428 (2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740 (2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819 (2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).

