DNA Onarım Proteinlerinin İmmünfluoresan Etkileşiminin Görselleştirilmesi
Summary
DNA hasarının ardından, insan hücreleri genomlarının bütünlüğünü geri yüklemek için gerekli onarım yollarını aktive ederler. Burada, dolaylı immünoffloresan yöntemini DNA onarım proteinlerini tespit etmek, mekansal ve zamansal işe alımlarını analiz etmek ve DNA hasarı nın olduğu yerlerde protein-protein etkileşimini sorgulamaya yardımcı olmak için bir araç olarak tanımlıyoruz.
Abstract
Memeli hücreleri sürekli kimyasallara maruz kalırlar, radyasyon, ve doğal olarak meydana gelen metabolik yan ürünler, DNA hakaret belirli türleri oluşturmak. Genotoksik ajanlar DNA omurgasına zarar verebilir, kırabilir veya tek tek bazların kimyasal doğasını değiştirebilir. DNA hakaretinin ardından DNA hasar yanıtı (DDR) yolları aktive edilir ve onarımda yer alan proteinler işe alınır. Hasarın türünü algılamada ve uygun onarım yanıtını etkinleştirmede çok sayıda faktör rol oynar. DDR faktörlerinin doğru bir şekilde etkinleştirilmesi ve işe alınmaması, kanser de dahil olmak üzere birçok insan patolojisinde yatan genomik istikrarsızlığa yol açabilir. DDR proteinleri çalışmaları genotoksik ilaç yanıtı ve ilaç direnci hücresel mekanizmaları içine anlayışlar sağlayabilir.
Proteinleri in vivo’dagörselleştirmenin iki ana yolu vardır: doğrudan gözlem, ilgi proteinini floresan proteinle etiketleyerek ve canlı görüntüleme ile takip ederek veya sabit numunelerde dolaylı immünofülans. Floresan etiketli proteinlerin görselleştirilmesi zaman içinde hassas izleme sağlarken, N- veya C-terminus doğrudan etiketleme protein lokalizasyonu veya fonksiyonu ile engelleyebilir. Proteinlerin değiştirilmemiş, endojen versiyonlarında gözlemi tercih edilir. DNA onarım proteinleri DNA hakaret işe olduğunda, onların konsantrasyonu yerel artar ve gruplar oluşturur, ya da “foci”, belirli antikorlar kullanılarak dolaylı immünoresans ile görselleştirilmiş olabilir.
Protein foci’nin saptanması doğrudan etkileşimin kesin bir kanıtı olmasa da, hücrelerdeki proteinlerin birlikte lokalizasyonu hasar mahallinde yeniden grupladıklarını ve karmaşık oluşum için gerekli olayların dizisini bilgilendirebileceklerini gösterir. Bir proteinin vahşi tipini veya mutant versiyonlarını ifade eden hücrelerdeki fosi uzamsal örtüşmelerin dikkatli analizi, DNA onarım fonksiyonu için önemli olan fonksiyonel etki alanları hakkında değerli ipuçları sağlayabilir. Son olarak, proteinlerin birlikte lokalizasyonu, hücrelerde ko-immünopresipitasyon veya saflaştırılmış proteinler kullanılarak doğrudan aşağı çekilme ile doğrulanabilecek olası doğrudan etkileşimleri gösterir.
Introduction
İnsan hücreleri sürekli çeşitli kökenlerden DNA zararlı ajanlar çeşitli maruz kalırlar. Ekzojen kaynaklar çoğunlukla radyasyona maruz ilerler, kimyasallar (kemoterapötik ajanlar ve bazı antibiyotikler dahil) ve virüsler, ana endojen kaynaklar DNA replikasyonu ve oksidatif stres hataları içerir iken. Genotoksik maruziyetin doğrudan etkileri, strese ve maruz kalma dozuna bağlı olarak modifiye edilmiş bir bazdan potansiyel olarak öldürücü DNA çift iplikçik kırılamı (DSB) arasında değişebilir. Sonuçta, onarılmamış veya yanlış onarılmış DNA hasarı mutasyonların birikimine yol açabilir, genomik yeniden düzenlemeler, genom istikrarsızlık ve sonunda karsinogenez yol 1 .1 Memeli hücreleri DNA hasarı belirli türleri tanımak için karmaşık yollar gelişti2,3 ve zamanında onları onarmak, hücre döngüsü ilerlemesi ile senkronize.
İzole radyasyon (IR) DNA çift sarkıklık zarar ve çift iplikçik sonları oluşturur (DSBs), DNA hasarının en zararlı formlarından biri. MRN (MRE11, RAD50, NBS1) DNA sensörü olarak karmaşık fonksiyonları biter ve protein kiaz ataksi telanjiektazi mutasyona (ATM)4,5aktive . DNA biter tarafından ATM ilk aktivasyonu ardından, ATM önemli bir olay ile başlayan, mola yerinde DDR olaylar bir çağlayan tetikler, histon varyantı H2AXfosforilasyon 6. Kalıntı S139 üzerindeki H2AX fosforilasyon γH2AX içine aktive, DNA lezyonu etrafında megabazlar kadar bölgeleri kapsayan6,7,8,9. Bu olay DNA erişilebilirliğini artırır, işe alma ve diğer DNA onarım proteinleri birikimine yol 7. γH2AX bol ve özellikle DSB’leri çevreleyen indüklenen olduğundan, belirli antikorlar kullanılarak kolayca görselleştirilebilir ve genellikle DNA onarım alanında DSB’ler için bir taşıyıcı belirteç olarak kullanılır. Mola sinyali verildikten sonra hücreler DNA tamir yollarını aktive eder ve DNA hasarını işlerler. Protein MDC1 (DNA hasar kontrol noktası protein arabulucusu 1) doğrudan γH2AX10bağlanır , ATM11 ile etkileşime ve ayrıca NBS112,13. DSB’deki MRN kompleksinin konsantrasyonunun artırılmasına ve olumlu bir ATM geri besleme döngüsübaşlatılmasına katkıda bulunur. γH2AX, mola onarıldıktan sonra hızla çıkarılır ve dsb açıklığının izlenmesine izin verilir. Mikroskopinin ardından γH2AX’ın zaman içinde azalması, artık sonları ve DNA onarım veriminin dolaylı olarak ölçülmesini sağlar.
Ökaryotik hücreler DSB’leri çeşitli yollarla onarabilirler, iki ana hücre homolog olmayan son birleştirme (NHEJ) ve homolog rekombinasyon (HR) dir. NHEJ aslında genişletilmiş homoloji kullanmadan DNA çift iplikçikli biter ligates ve hücre döngüsü boyunca çalışır14,15. Hr S ve G2 aşamalarında baskın hale gelir, ve aksi takdirde bastırılmış, onarım için homolog bir şablon olarak bir kardeş kromatit gerektirir beri14,16. NHEJ ve İk arasındaki yol seçimi sadece kardeş kromatit fiziksel yakınlığına bağlıdır, ama aynı zamanda DNA sonu rezeksiyon genişletmekbağlıdır 17, Hangi NHEJ inhibe.
Homolojiye bağımlı DSB onarımı, 5′ ipliğin kopma uçlarından nükleolitik bozulma ile başlatılarak 3′ tek iplikçikli DNA (ssDNA) kuyrukları oluşturmak için, 5′-3′ rezeksiyon olarak adlandırılan bir süreçtir. MRN kompleksi DNA sonu rezeksiyonu başlatır ve daha fazla rezeksiyon BLM / EXO1 (Bloom sendromu protein / ekonenaz 1) veya BLM / DNA2 (DNA replikasyonu ATP bağımlı helicase / nükleaz)18,19,20,21,22ile birlikte işlenir . DNA sonu rezeksiyonu CTIP (CtBP-etkileşen protein) mrn kompleksi ile doğrudan etkileşim yoluyla geliştirilmiştir23 ve BRCA1 işe (meme kanseri tip 1 duyarlılık proteini)24,25. Replikasyon proteinI A (RPA) derhal açığa çıkan ssDNA bağlanır ve daha sonra homolog arama ve iplikçik invazyonu katalizler bir nükleoprotein filament oluşturmak için rekombinaz protein RAD51 tarafından yerinden edilir26,27,28.
Rezeksiyon un başlatılması onarım yolu seçimi için kritik bir adımdır. Bir kez rezeksiyon başladı, DNA biter Ku70/Ku80 heterodimer (NHEJ yolu bileşeni) tarafından bağlayıcı için zayıf substratlar haline gelir ve hücreler HR17,,29,30kararlıdır . Ku70/Ku80 heterodimer DSB biter bağlanır, DNA-PKcs ve p53 Bağlayıcı Protein 1 (53BP1)29,,30işe. 53BP1 G1 rezeksiyon için bir bariyer olarak görür, böylece NHEJ teşvik ederken İk engelleme31,32, ama S fazında brca1 bağımlı bir şekilde kaldırılır, sonuç olarak rezeksiyon oluşmasına izin33,34. Bu nedenle, 53BP1 ve BRCA1 DSB onarımında karşıt roller oynarken, 53BP1 NHEJ kolaylaştırıcısı olurken BRCA1, hr aracılığıyla onarım molalarını sağlar.
Laboratuvarda, DSB oluşumu iyonlaştırıcı radyasyon (IR) ile indüklenebilir. Bu örnekte yüksek dozda 4 Gy, 1 Gy ve 2 Gy de bol proteinlerle fosi oluşumunun analizi için uygun miktarda DSB oluşturur. Kullanılan radyasyonun türü ve dozu DNA’da ve hücrede farklı lezyonlara yol açabilir dikkat etmek önemlidir: IR DSB’ler indükler iken, aynı zamanda tek iplikçik kırıkları veya baz modifikasyonu neden olabilir (ışınlama lineer enerji transferi bir referans için35,36 bakınız (LET) ve DNA hasarı türü). Radyasyona bağlı fok oluşumunun iyonlaştırıcı kinetikbelirlemek için (IRIF) oluşumu ve bunların temizliği, aktif DDR8,9,,37,38hasar ve ters onarım gösteren, fosi oluşumu iyonlaştırıcı radyasyon sonra farklı zaman noktalarında izlenebilir. Aktivasyon zamanlaması ve tüm büyük DNA hasarı proteinlerin temizlenmesi39bilinmektedir , ve birçok önemli olayların vekil işaretleri olarak araştırılmaktadır. Örneğin, ssDNA’ya yüksek afiniteye sahip olan pRPA, kırılma rezeksiyonunun taşıyıcı sı, MRN proteinleri (MRE11, RAD50, NBS1) ve eksonüekleaseler rezeksiyon verimliliğini değerlendirmek için de kullanılabilir. RAD51, BRCA1, BRCA2 (meme kanseri tip 2 duyarlılık proteini) ve PALB2 (BRCA2’nin partneri ve lokalizatörü) HR verimliliğini değerlendirmek için izlenebilse de, Ku proteinlerinin varlığı veya 53BP1, NHEJ’nin belirteçleri olarak kullanılır (Şekil 1).
DNA onarım makinelerinin proteinleri birbirlerini süper komplekslerde biraraya getirerek biraraya getirdikçe, DNA-protein ve protein-protein etkileşimleri zaman içinde bireysel lokalizasyonlarını takip ederek ve proteinlerin eş lokalizasyonunu analiz ederek çıkarılabilir, hücre40,41,42’dekisinyalleri örtüşerek görselleştirilmiştir. Hücre hatlarında, belirli DNA onarım genlerinde nokta mutasyonlarının veya silmenin genom düzenlemesi yoluyla ya da plazmid tabanlı mutantların aşırı ekspresyonu yoluyla başlatılması, spesifik kalıntıların araştırılmasına ve DNA hasarının tanınmasındaki olası rollerinin (örneğin, γH2AX ile birlikte lokalizasyon) veya karmaşık montajın (başka bir veya birkaç proteinle birlikte lokalizasyon) yanı sıra DNA onarımı üzerindeki etkilerinin araştırılmasına olanak sağlar. Burada, dsbs oluşumunu ve çözünürlüğünü araştırmak için bir ortalama olarak dolaylı immünfloresans kullanın γH2AX foci zaman içinde takip ederek. Ayrıca DSB onarımönemli bir oyuncu tarafından foci oluşumu ve co-lokalizasyon analizi bir örnek satıyoruz: p53 Bağlayıcı Protein 1 (53BP1)32. Daha önce de belirtildiği gibi, 53BP1 DNA onarım yolu seçimi merkezi olarak kabul edilir. ΓH2AX ile birlikte 53BP1 birikimi ve birlikte lokalizasyonu hücre döngüsü fazı, DNA hasar birikimi ve DSB’leri onarmak için kullanılan yol hakkında değerli bilgiler sağlar. Dolaylı immünolokalizasyonun amacı, bu çalışmada olduğu gibi IR’yi takip ederek veya hücredeki çeşitli streslere maruz kaldıktan sonra, DNA çapraz bağlantısından çoğaltma çatalının tıkanması için hücre hatlarındaki DNA hasar onarımının etkinliğini değerlendirmektir (DNAzararlı ajanların listesi Tablo 1’de verilmiştir).
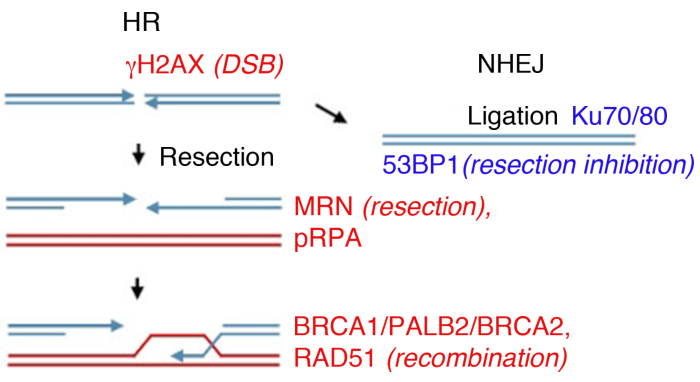
Şekil 1: DNA çift iplikçik kırıkları (DSB) onarım yolları.
DSB onarımı iki ana yolu içerir: Homolog Rekombinasyon (İk, sol) ve Non-Homolog End-Joining (NHEJ, sağ). Aradan sonra proteinler break (γH2AX) işaretlemek için aktive olsun, son rezeksiyon (MRN) katılmak, rezeke ssDNA (pRPA), rekombinasyon teşvik (BRCA1, PALB2, BRCA2, RAD51) veya rezeksiyon sınırlamak ve NHEJ (53BP1) teşvik. Diğer proteinler hasar onarımına katılırlar, ancak listelenen proteinler rutin olarak dolaylı immünfloresans tarafından takip edilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| DNA zararlı madde | Etki mekanizması | Önerilen doz |
| γ-ışınları/X-ışınları | Radyasyon Bazı kontrolsüz hücresel etkilerle çift iplikli kırıkların oluşması |
1-4 Gy |
| 36.000 Ar iyonları | Radyasyon Çift iplikli sonları oluşumu |
270 keV/μm |
| α parçacıkları | Radyasyon Çift iplikli sonları oluşumu |
116 keV/μm |
| Bleomisin | DNA sentezinin inhibitörü | 0.4-2 μg/mL |
| Camptothecin | Topoisomerase Inhibitörü I | 10-200 nM |
| Sisplatin | Alkylating ajan (intrastrand crosslinks indükleyen) |
0,25-2 μM |
| Doksorubisin | Ara ajan Topoisomerase Inhibitörü II |
10-200 nM |
| Etoposide | Topoisomerase Inhibitörü II | 10 μM |
| Hidroksyurea | DNA sentezinin inhibitörü (ribonükleotid redüktaz tarafından) |
10-200 μM |
| Metil metansülfonat | Alkylating ajan | 0,25-2 mM |
| Mitomisin C | Alkylating ajan | 0,25-2 μM |
| Ultraviyole (UV) ışık | Timidin dimers oluşumu (DNA zincirinin bozulmasını üreten) |
50-100 mJ/cm2 |
Tablo 1: Genotoksik ajanlar. DNA’ya zarar veren ajanlar, etki mekanizmaları ve önerilen çalışma konsantrasyonuna dayalı hasar örnekleri.
Protocol
Representative Results
Discussion
DNA hasar onarımının mikroskobik ile zamanlaması ve etkinliğinin analizi, DNA onarım makinelerinin normal hücrelerde ve kanser gibi insan patolojilerinde nasıl çalıştığını anlamak için gerekli olmuştur.
Onların fosforile sürümünde aktive proteinlerin tespitine olanak sağlayan spesifik antikorların geliştirilmesi (γH2AX, pRPA, pRAD50 ve diğerleri gibi10,23,39,<sup c…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Bu çalışma San Antonio Area Foundation’ın bir bağışı ile desteklenmiştir. Mays Kanser Merkezi NCI kanser merkezi destek çekirdek hibe P30 CA054174 tarafından desteklenir. Stephen Holloway’e reaktifleri ve Sung laboratuvarına alan ve mikroskopi kapasitesi sağladığı için yardımları için teşekkür ederiz.
Materials
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
References
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600 (2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757 (2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428 (2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740 (2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819 (2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).

