الليزر التقاط Microdissection RNA تسلسل لتحليل التعبير الجيني المكاني والزماني محددة في النباتات
Summary
هنا هو بروتوكول لالتقاط الليزر microdissection (LCM) من الأنسجة النباتية. LCM هي تقنية مجهرية لعزل مناطق الأنسجة بطريقة خالية من التلوث. ويشمل الإجراء تثبيت الأنسجة، التضمين البارافين، والتجزئة، LCM واستخراج الحمض النووي الريبي. يتم استخدام الحمض النووي الريبي في تحليل الأنسجة المصب محددة، حل مؤقتا من transcriptomes.
Abstract
ويخضع تطوير كائن معقد متعدد الخلايا لأنواع خلايا مختلفة لها ملامح مختلفة. لتحديد الشبكات التنظيمية النسخية التي تحكم العمليات التنموية من الضروري قياس ملامح التعبير الجيني المكاني والزماني لهذه الأنواع من الخلايا الفردية. ولذلك، فإن التبصر في الرقابة الوُقتائية على التعبير الجيني أمر ضروري لفهم كيفية تنظيم العمليات البيولوجية والإنمائية. هنا، نحن وصف طريقة التقاط الليزر microdissection (LCM) لعزل عدد صغير من الخلايا من ثلاثة أعضاء جنين الشعير على مدى فترة زمنية خلال عملية الإنبات تليها التنميط النص. وتتألف هذه الطريقة من تثبيت الأنسجة، وتجهيز الأنسجة، والبارافين تضمين، والقسم، LCM و RNA استخراج تليها في الوقت الحقيقي PCR أو RNA-seq. وقد مكنتنا هذه الطريقة من الحصول على ملامح مكانية والزمنية من نسخ أعضاء البذور من أعداد مختلفة من الخلايا (من عشرات إلى مئات)، مما يوفر خصوصية الأنسجة أكبر بكثير من التحليلات النموذجية لالأنسجة السائبة. من هذه البيانات كنا قادرين على تحديد ومقارنة الشبكات التنظيمية النسخية وكذلك التنبؤ بعوامل النسخ التنظيمية المرشحة للأنسجة الفردية. وينبغي أن تكون الطريقة قابلة للتطبيق على الأنسجة النباتية الأخرى مع الحد الأدنى الأمثل.
Introduction
يشمل تطوير النبات ونموه العمل المنسق للشبكات التنظيمية النسخية داخل خلايا مختلفة موجودة في بيئة خلوية معقدة. لفهم نشاط هذه الشبكات التنظيمية، نحن بحاجة إلى معرفة التعبير الجيني المكاني والزماني داخل أنواع الخلايا المختلفة عبر مراحل النمو. ومع ذلك، فإن تحليلات التعبير الجيني تجري بشكل أكثر شيوعا في أعضاء كاملة أو عينات الأنسجة السائبة بسبب التحدي التقني المتمثل في عزل وتحليل أعداد صغيرة من الخلايا. وقد سمحت الطريقة التي نصفها هنا الحصول على تحليل نسخي مكاني وزمني من خلال اقتران LCM مع RNA-seq.
وقد وضعت LCM قبل عقدين من قبل Emmert باك وزملاؤه1. وقد مكنت هذه التقنية الباحثين من عزل الخلايا المفردة أو مجموعات الخلايا بدقة عن بيئتهم باستخدام التصور المجهري المباشر والتلاعب بأشعة الليزر الضيقة1. ومنذ ذلك الحين تم استخدام هذه الطريقة على نطاق واسع في علم الأحياء السرطان وعلم الأمراض2,3. كما قامت العديد من مجموعات البحوث النباتية بتكييف LCM للاستخدام مع أنواع النباتات المختلفة وأنواع الأنسجة المختلفة4،5،66،7،8،9،10،11. في الآونة الأخيرة، استخدمت العديد من الأوراق أيضا LCM على بذور eudicot و monocot لدراسة الجنين، endosperms وغيرها من هياكل البذور أثناء تطوير البذور والإنبات10،12،13. معظم غيرها من أساليب العزل خلية واحدة شائعة الاستخدام مثل الأنابيب الدقيقة، وفرز الخلايا، وفصل المغناطيسي ومنصات microfluidic تعتمد على الهضم الأنزيمي أو التجانس الميكانيكية لفصل الخلايا. هذا قد يربك تعبير الجينات، وإدخال التحف الفنية التي تربك تفسير البيانات14،15. وتتطلب هذه الطرق أيضا المعرفة السابقة للجينات علامة لكل نوع الخلية لربط الخلايا المنفصلة إلى موقعها المكاني ونوع الخلية الحقيقية. وهناك مجموعة أخرى من التقنيات يعتمد على العزلة على أساس تقارب من الهياكل دون الخلوية بدلا من الخلايا الكاملة، على سبيل المثال سليمة (عزل نوكلي الموسومة في أنواع الخلايا) و TRAP (ترجمة تنقية تقارب الريبوسوم)16،17. ومع ذلك، فإن وضع العلامات على القرابة وتنقية النوى أو الريبوسومات أمر صعب تقنياً في الأنواع النباتية التي ليس لديها بروتوكولات تحويل راسخة. LCM يستفيد من تثبيت الأنسجة السريعة للحفاظ على مستويات النص وتحديد الأنسجة التقليدية عن طريق التصور المباشر للخلايا داخل سياقها الأنسجة / الجهاز الطبيعي، والذي يسمح للخلايا منفصلة لتكون معزولة في فترة قصيرة من الوقت18،19.
البروتوكول المقدم هنا هو طريقة محسنة لعزل خلايا محددة أو أنواع الخلايا من أقسام الأنسجة من بذور الحبوب، والتي يمكن تطبيقها على معظم الخلايا التي يمكن تحديدها من الناحية النسيجية. يوفر LCM طريقة خالية من الاتصال لعزل الخلايا ، مما يقلل إلى حد كبير من التلوث وزيادة سلامة الحمض النووي الريبي المستعاد. وعلاوة على ذلك، توضح هذه الطريقة قوة LCM على الدراسات واسعة النطاق الجينوم بدءا من كميات صغيرة من المواد البيولوجية. كما أننا نُصف التضخيم الخطي للرنا لتوليد مواد إدخال كافية لتحليلات النسخ/النسخ.
هناك عشر خطوات رئيسية في هذا البروتوكول LCM RNA-seq لـ transcriptomes المكانية والزمانية الخاصة بالأنسجة، بما في ذلك تثبيت عينات الأنسجة، والجفاف، وتسرب البارافين، والتضمين، والقطع، واللمس، واستخراج الحمض النووي الريبي، والتضخيم RNA، والكمية RNA وqRT-PCR و /أو RNA-seq(الشكل 1).
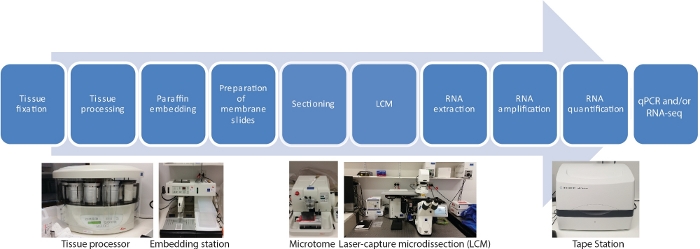
الشكل 1: مخطط انسيابي من LCM متبوعاً بـ RNA-seq أو qRT-PCR. LCM هو تقنية دقيقة مكانيا وخالية من الاتصال لجمع الخلايا من أقسام الأنسجة الثابتة باستخدام شعاع الليزر تحت التصور المجهري. تبدأ العملية مع تثبيت عينات الأنسجة ، تليها الجفاف باستخدام سلسلة متدرجة من الإيثانول والزيلين ، وانتهت مع تسلل البارافين. يمكن أن تكون العملية مؤتمتة بالكامل باستخدام معالج الأنسجة. مرة واحدة يتم اختراق الأنسجة مع البارافين، يتم تضمينه في قالب مع البارافين المنصهر باستخدام محطة تضمين. يتم إجراء اقطع باستخدام microtome تعيين إلى سمك المطلوب. يتم إعداد الشرائح وLCM التي أجريت مباشرة قبل RNA هو أن تستخرج من الخلايا التي تم التقاطها. ويتبع استخراج الحمض النووي الريبي مباشرة من قبل جولتين من تضخيم الحمض النووي الريبي قبل qRT-PCR و / أو RNA-seq. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
Representative Results
Discussion
وقد تم الحد من العديد من دراسات التعبير الجيني الخاصة بالأنسجة عن طريق تشريح اليد للعينات ، والتي تستغرق وقتا طويلا ، والعمالة المكثفة ، لديها خطر كبير من التلوث ويمكن أن تستخدم فقط العينات التي تعمل الإنسان هو بارعة بما فيه الكفاية لحصاد. LCM هو تقنية دقيقة وخالية من الاتصال لجمع الخلايا م?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
وقد دعم هذا العمل من قبل مجلس البحوث الأسترالية من التميز في بيولوجيا الطاقة النباتية (CE140100008) إلى JW. تم دعم M.G.L من قبل جامعة لا تروب منحة بدء. نشكر منصة لا تروب جينوميكس على دعمها في تسلسل عالي الإنتاجية وتحليل البيانات. نشكر البروفيسور المشارك ماثيو تاكر على مشورة الخبراء حول إنشاء LCM في مختبرنا.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
References
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

