Funksjonell vurdering av BRCA1-varianter ved hjelp av CRISPR-Medierte baseredigerere
Summary
Personer med BRCA1 mutasjoner har en høyere risiko for å utvikle kreft, noe som garanterer nøyaktig evaluering av funksjonen til BRCA1-varianter. Her beskrev vi en protokoll for funksjonell vurdering av BRCA1-varianter ved hjelp av CRISPR-medierte cytosinbaseredigerere som muliggjør målrettet C:G til T:A konvertering i levende celler.
Abstract
Nyere studier har undersøkt risikoen forbundet med BRCA1 genmutasjoner ved hjelp av ulike funksjonelle vurderingsmetoder som fluorescerende reporteranalyser, embryonale stamcellevariasjonsanalyser og terapeutiske legemiddelbaserte sensitivitetsanalyser. Selv om de har avklart mange BRCA1-varianter, er disse analysene som involverer bruk av eksogent uttrykte BRCA1-varianter forbundet med overuttrykksproblemer og kan ikke brukes på post-transkripsjonsregulering. For å løse disse begrensningene har vi tidligere rapportert en metode for funksjonell analyse av BRCA1-varianter via CRISPR-mediert cytosinbaseredigerer som induserer målrettet nukleotidsubstitusjon i levende celler. Ved hjelp av denne metoden identifiserte vi varianter hvis funksjoner forblir tvetydige, inkludert c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T og c.4986+5G>A, og bekreftet at CRISPR-medierte baseredigerere er nyttige verktøy for å reklassifisere variantene av usikker betydning i BRCA1. Her beskriver vi en protokoll for funksjonell analyse av BRCA1-varianter ved hjelp av CRISPR-basert cytosinbaseredaktør. Denne protokollen inneholder retningslinjer for valg av målområder, funksjonell analyse og evaluering av BRCA1-varianter.
Introduction
Brystkreft type 1 følsomhet genet (BRCA1) er en allment kjent tumor suppressor genet. Fordi BRCA1-genet er relatert til reparasjon av DNA-skader, vil mutasjoner i dette genet føre til en større risiko for kreftutvikling hos en individuell1. Brystkreft, ovariekreft, prostata og bukspyttkjertelkreft er knyttet til arvelige funksjonstap (LOF) mutasjoner av BRCA1 genet 2. Funksjonell vurdering og identifisering av BRCA1-varianter kan bidra til å forebygge og diagnostisere de ulike sykdommene. For å adressere funksjonen til BRCA1-varianter er det utviklet flere metoder for å undersøke patogeniteten til BRCA1-varianter som embryonale stamcellevariasjonsanalyser, fluorescerende reporteranalyser og terapeutiske legemiddelbaserte følsomhetsanalyser3,4,5,6. Selv om disse metodene har vurdert funksjonen til mange BRCA1-varianter, utgjør metodene som involverer eksogent uttrykt BRCA1-varianter begrensninger når det gjelder overuttrykk som kan påvirke nedstrømsregulering, gendosering og proteinfolding7. Videre kan disse analysene ikke utnyttes til posttranscriptional regulering som mRNA spleising, transkripsjon stabilitet, og effekten av uoversettet region8,9.
CRISPR-Cas9-systemet muliggjør målrettet genomredigering i levende celler ogorganismer 10. Gjennom en enkeltguide RNA, Cas9 kan indusere dobbel-strand pauser (DSBs) i kromosomalt DNA på bestemte genomisk loci for å aktivere to DNA reparasjonsveier: feilutsatt nonhomologous end-joining (NHEJ) pathway og feilfri homologi-rettet reparasjon (HDR) sti11. HDR er en presis reparasjonsmekanisme; DSBs indusert av Cas9-kjerner for HDR resulterer imidlertid ofte i uønsket innsetting og sletting (indel) mutasjon. I tillegg trenger den homologe donor DNA-maler for å reparere DNA-skader og har relativt lav effektivitet. Nylig har Cas9 nickase (nCas9) blitt smeltet sammen med cytidindeaminasedomener for målretting av C:G til T:A-erstatninger, uten behov for homologe DNA-maler og DNA-dobbelttrådbryter12,13,14,15. Ved hjelp av cytosinbaseredaktøren utviklet vi en ny metode for funksjonell analyse av BRCA1-varianter16.
I denne studien brukte vi CRISPR-mediert cytosinbaseredaktør, BE314, som induserer effektive C:G til T:A punktmutasjoner, for å implementere den funksjonelle vurderingen av BRCA1-varianter og vellykket identifisert funksjonene til flere BRCA1-varianter (figur 1).
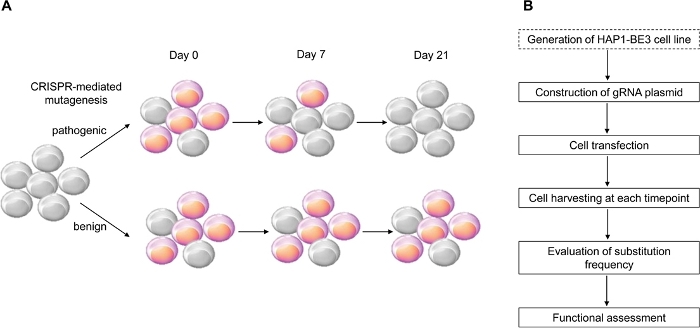
Figur 1: En oversikt over arbeidsflyten for funksjonell vurdering. (A) Skjematisk viser funksjonell vurdering av BRCA1. Fordi LOF av BRCA1 påvirker celle levedyktighet, når BRCA1 mutasjonen er patogen, cellene dør som passasjen tallet øker. (B) Stadier av funksjonsvurderingen av BRCA1. Stiplet boks er valgfritt. Det kan erstattes ved samtidig transfection av gRNA uttrykker og BE3 uttrykker plasmider DNA. Vennligst klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
Denne protokollen beskriver en enkel metode for funksjonelle vurderinger av BRCA1-varianter ved hjelp av CRISPR-meditert cytosinbaseredigerer. Protokollen beskriver metoder for utforming av gRNAs på mållocus og bygging av plasmid DNAs som de uttrykkes fra. Cytosinbaseredaktører induserer nukleotidkonvertering i et aktivt vindu (i tilfelle BE3, nukleotider 4–8 i PAM-distale enden av gRNA-målsekvensene). Forskeren bør nøye velge målsekvenser fordi alle cytosiner i aktivt vindu kan erstattes med thyminer. …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Dette arbeidet ble støttet av National Research Foundation of Korea (tilskudd 2017M3A9B4062419, 2019R1F1A1057637 og 2018R1A5A2020732 til Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
References
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Recherche en cancérologie. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

