Функциональная оценка вариантов BRCA1 с использованием базовых редакторов CRISPR-Mediated
Summary
Люди с мутациями BRCA1 имеют более высокий риск развития рака, что требует точной оценки функции вариантов BRCA1. В этом случае мы описали протокол функциональной оценки вариантов BRCA1 с использованием редакторов базы цитозина, опосредованной CRISPR, которые позволяют целевое преобразование C:G в T:A в живых клетках.
Abstract
Недавние исследования исследовали риски, связанные с мутациями генов BRCA1 с использованием различных функциональных методов оценки, таких как флуоресцентные анализы репортеров, анализ жизнеспособности эмбриональных стволовых клеток и терапевтические анализы чувствительности на основе наркотиков. Хотя они уточнили много вариантов BRCA1, эти анализы, связанные с использованием экзогенно выраженных вариантов BRCA1, связаны с проблемами переэкспрессии и не могут быть применены к пост-транскрипционные регулирования. Чтобы устранить эти ограничения, мы ранее сообщали о методе функционального анализа вариантов BRCA1 с помощью базового редактора цитозинов, опосредованного CRISPR, который вызывает целевую замену нуклеотида в живых клетках. Используя этот метод, мы определили варианты, функции которых остаются неоднозначными, в том числе c.-97C’gt;T, c.154C’gt;T, c.3847C’gt;T, c.5056C’gt;T, и c.4986’5G’gt;A, и подтвердили, что базовые редакторы CRISPR являются полезными инструментами для реклассификации вариантов неопределенного значения в BR1CA. Здесь мы описываем протокол функционального анализа вариантов BRCA1 с помощью базового редактора цитозина на основе CRISPR. Этот протокол содержит руководящие принципы для выбора целевых объектов, функционального анализа и оценки вариантов BRCA1.
Introduction
Ген восприимчивости к раку молочной железы типа 1(BRCA1)является широко известным геном супрессора опухоли. Поскольку ген BRCA1 связан с повреждением ДНК, мутации в этом гене приведут к большему риску развития рака у человека1. Рак молочной железы, яичников, простаты и поджелудочной железы связан с унаследованными потеря функции (LOF) мутации гена BRCA1 2. Функциональная оценка и выявление вариантов BRCA1 может помочь в профилактике и диагностике различных заболеваний. Для решения функции вариантов BRCA1, несколько методов были разработаны и широко используются для изучения патогенности вариантов BRCA1, таких как эмбриональные анализы жизнеспособности стволовых клеток, флуоресцентные анализы репортера, и терапевтические чувствительность наоснове наркотиков анализы 3,4,5,6. Хотя эти методы оценили функцию многих вариантов BRCA1, методы, включающие экзогенно выраженные варианты BRCA1, создают ограничения с точки зрения переэкспрессии, которые могут повлиять на регуляцию ниже по течению, дозировку генов исворачивание белка 7. Кроме того, эти анализы не могут быть использованы для посттранстуального регулирования, такие как сращивание мРНК, стабильность транскрипта иэффект непереводимой области 8,9.
Система CRISPR-Cas9 позволяет целенаправленно редактировать геном в живых клетках и организмах10. С помощью одногидной РНК, Cas9 может вызвать двухструнные перерывы (DSBs) в хромосомной ДНК на конкретных геномных локусов для того, чтобы активировать два пути восстановления ДНК: подверженных ошибкам нехомологических конце присоединения (NHEJ) путь и безошибочно гомологии направленного ремонта (HDR)путь 11. HDR является точным механизмом ремонта; однако DSBs, индуцированные ядром Cas9 для HDR, часто приводит к нежелательной вставке и удалению (инделя) мутации. Кроме того, он нуждается в гомологичные шаблоны донорской ДНК для восстановления повреждения ДНК и имеет относительно низкую эффективность. В последнее время Cas9 nickase (nCas9) были слиты с цитидин deaminase доменов для ориентации C:G к T: Замены, без необходимости гомологичные шаблоны ДНК и ДНК двойнойнити перерывы 12,13,14,15. Используя редактор базы цитозина, мы разработали новый метод функционального анализа вариантовBRCA1 16.
В этом исследовании мы использовали CRISPR-опосредованного редактора базы цитозина, BE314, который индуцирует эффективные C:G к T:A точечные мутации, для реализации функциональной оценки вариантов BRCA1 и успешно определили функции нескольких вариантов BRCA1 (рисунок 1).
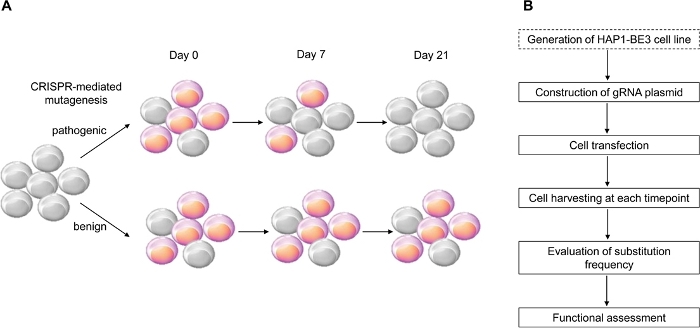
Рисунок 1: Обзор рабочего процесса для функциональной оценки. (A)Схема, показывающая функциональную оценку BRCA1. Поскольку LOF BRCA1 влияет на жизнеспособность клеток, когда мутация BRCA1 является патогенной, клетки умирают по мере увеличения числа проходов. (B)Этапы функциональной оценки BRCA1. Пунктирная коробка необязательна. Его можно заменить ко-трансфектированием экспрессии гРНК и BE3, выражаюющих ДНК плазмидов. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Protocol
Representative Results
Discussion
Этот протокол описывает простой метод функциональных оценок вариантов BRCA1 с использованием базового редактора CRISPR-медитируемого цитозина. В протоколе описаны методы проектирования ГРНК в целевом локусе и строительства плазмидных ДНК, из которых они выражены. Редакторы базы цито…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Эта работа была поддержана Национальным исследовательским фондом Кореи (гранты 2017M3A9B4062419, 2019R1F1A1057637, и 2018R1A5A2020732 до Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
References
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Recherche en cancérologie. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

