Imaging a cellule vive di array a cella singola (LISCA) - una tecnica versatile per quantificare la cinetica cellulare
Summary
Presentiamo un metodo per l’acquisizione di corsi di tempo reporter a fluorescenza da singole cellule utilizzando array micropatterned. Il protocollo descrive la preparazione di array a cella singola, la configurazione e il funzionamento della microscopia time-lapse di scansione a celle vive e uno strumento di analisi delle immagini open source per la preselezione automatizzata, il controllo visivo e il tracciamento dei corsi di tempo di fluorescenza integrati nelle celle per sito di adesione.
Abstract
Live-cell Imaging of Single-Cell Arrays (LISCA) è un metodo versatile per raccogliere corsi di tempo di segnali di fluorescenza da singole cellule ad alta produttività. In generale, l’acquisizione di corsi di tempo a cella singola da cellule coltivate è ostacolata dalla motilità cellulare e dalla diversità delle forme cellulari. I micro array adesivi standardizzano le condizioni a cella singola e facilitano l’analisi delle immagini. LISCA combina microarray a cella singola con microscopia time-lapse di scansione e elaborazione automatizzata delle immagini. Qui descriviamo le fasi sperimentali di prendere corsi di tempo di fluorescenza a singola cella in formato LISCA. Trasfetteiamo le cellule aderenti a un array di micropattern utilizzando la codifica mRNA per proteine fluorescenti verdi avanzate (eGFP) e monitoriamo la cinetica di espressione eGFP di centinaia di cellule in parallelo tramite microscopia time-lapse di scansione. Gli stack di dati delle immagini vengono elaborati automaticamente da un software di nuova sviluppo che integra l’intensità della fluorescenza sui contorni delle celle selezionati per generare corsi di tempo a fluorescenza a singola cella. Dimostriamo che i corsi di tempo di espressione eGFP dopo la trasfezione dell’mRNA sono ben descritti da un semplice modello di traduzione cinetica che rivela i tassi di espressione e degradazione dell’mRNA. Vengono discusse ulteriori applicazioni di LISCA per le correlazioni del tempo degli eventi di più marcatori nel contesto dell’apoptosi di segnalazione.
Introduction
Negli ultimi anni, l’importanza degli esperimenti a una cellula è diventata evidente. I dati provenienti da singole celle consentono l’analisi della variabilità da cella a cella, la risoluzione delle correlazioni dei parametri intracellulari e il rilevamento della cinetica cellulare che rimangono nascoste nelle misurazionidell’insieme 1,2,3. Al fine di studiare la cinetica cellulare di migliaia di singole cellule in parallelo, sono necessari nuovi approcci che consentano di monitorare le cellule in condizioni standardizzate per un periodo di tempo di diverse ore fino a diversi giorni seguito da un’analisi quantitativa dei dati 4. Qui presentiamo Live-cell Imaging of Single-Cell Arrays (LISCA), che combina l’uso di array microstrutturati con microscopia time-lapse e analisi automatizzata delle immagini.
Diversi metodi per generare array microstrutturati a cella singola sono stati stabiliti e pubblicati nella letteratura5,6. Qui, descriviamo brevemente il modello proteico avviato dal plasma su microscala (μPIPP). Un protocollo dettagliato della fabbricazione di array a cella singola utilizzando μPIPP si trova anche nel riferimento7. L’uso di matrici a cella singola consente l’allineamento di migliaia di celle su punti di adesione standardizzati che presentano microambientati definiti per ogni cella e riduce così una fonte di variabilità sperimentale (Figura 1A). Gli array a cella singola vengono utilizzati per monitorare i corsi di tempo dei marcatori fluorescenti allo scopo di indicare una varietà di processi cellulari. La microscopia a lungo termine in modalità time-lapse di scansione consente di monitorare una vasta area degli array a singola cella e quindi di campioare i dati a cella singola in alta velocità effettiva per un tempo di osservazione di diverse ore o addirittura giorni. In questo modo vengono generati stack di immagini a linee di tempo da ogni posizione della matrice (Figura 1B). Al fine di ridurre la grande quantità di dati immagine ed estrarre i corsi di tempo di fluorescenza a singola cella desiderati in modo efficiente, è necessaria un’elaborazione automatizzata delle immagini che sfrofitti il posizionamento delle cellule (Figura 1C).
La sfida di LISCA è adattare i protocolli sperimentali e gli strumenti computazionali per formare un saggio ad alta produttività che generi dati quantitativi e riproducibili della cinetica cellulare. In questo articolo forniamo una descrizione dettagliata dei singoli metodi e di come vengono combinati in un test LISCA. Ad esempio, discutiamo il corso del tempo dell’espressione di proteina fluorescente verde migliorata (eGFP) dopo la consegna artificiale di mRNA. L’espressione eGFP dopo la consegna dell’mRNA è descritta da equazioni della velocità di reazione che modellano la traduzione e la degradazione dell’mRNA. Adattando la funzione del modello per il corso del tempo della concentrazione eGFP alla lettura LISCA dell’intensità di fluorescenza per ogni singola cella nel tempo si ottiene la migliore stima dei parametri del modello come il tasso di degradazione dell’mRNA. Come risultato rappresentativo discutiamo dell’efficienza di erogazione dell’mRNA di due diversi agenti di trasfezione a base lipidica e di come differiscono le loro distribuzioni dei parametri.
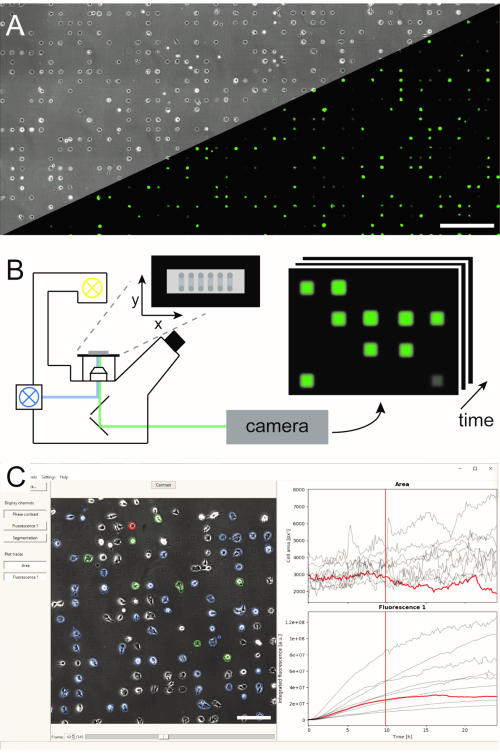
Figura 1: Rappresentazione del flusso di lavoro LISCA che combina(A) array a celle singole a micro pattern (B) microscopia time-lapse di scansione e (C) analisi automatica delle immagini delle serie di immagini registrate. Gli array a cella singola consistono in un modello bidimensionale di quadrati cell-adesivi con un interspazio repellente per cellule che porta a una disposizione delle cellule sul micropattern, come si può vedere nell’immagine a contrasto di fase e nell’immagine di fluorescenza delle cellule che esprimono eGFP (A). L’intera area microstrutturata viene immagine in modalità time-lapse di scansione che scatta ripetutamente immagini in una sequenza di posizioni (B). Le serie di immagini registrate vengono elaborate per leggere l’intensità di fluorescenza per cella nel tempo (C). Barre di scala: 500 μm(A),200 μm (C). Clicca qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Qui abbiamo descritto LISCA come una tecnica versatile per seguire la cinetica cellulare delle etichette fluorescenti intracellulari a livello a singola cellula. Per eseguire un esperimento LISCA riuscito, ciascuno dei passaggi descritti della sezione del protocollo deve essere stabilito singolarmente e quindi tutti i passaggi devono essere combinati. Ognuno dei tre aspetti principali di LISCA presenta passaggi cruciali.
Fabbricazione di microarray a cella singola
La qualità del m…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Questo lavoro è stato sostenuto da sovvenzioni della Fondazione tedesca della scienza (DFG) al Collaborative Research Center (SFB) 1032. Il sostegno del Ministero federale tedesco dell’istruzione, della ricerca e della tecnologia (BMBF) nell’ambito del progetto cooperativo 05K2018-2017-06716 Medisoft e una sovvenzione della Bayerische Forschungsstiftung sono riconosciuti con gratitudine. Anita Reiser è stata sostenuta da una Borsa di Studio DFG attraverso la Graduate School of Quantitative Biosciences Munich (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
References
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

