Визуализация живых клеток одноклеточных массивов (LISCA) - универсальный метод количественной оценки клеточной кинетики
Summary
Представлен метод получения флуоресцентных репортерных временных курсов из одиночных клеток с использованием микроструктурированных массивов. Протокол описывает подготовку одноэлементных массивов, настройку и эксплуатацию покадровой микроскопии сканирования живых клеток и инструмент анализа изображений с открытым исходным кодом для автоматизированного предварительного выбора, визуального контроля и отслеживания интегрированных в клетку курсов времени флуоресценции на сайт адгезии.
Abstract
Live-cell Imaging of Single-Cell Arrays (LISCA) является универсальным методом сбора временных курсов флуоресцентных сигналов от отдельных клеток с высокой пропускной способностью. В целом, приобретение одноклеточных временных курсов из культивируемых клеток затруднено подвижностью клеток и разнообразием форм клеток. Клеевые микрорешетки стандартизируют одноэлементные условия и облегчают анализ изображений. LISCA сочетает в себе одноэлементные микрочипы со сканирующей покадровой микроскопией и автоматизированной обработкой изображений. Здесь мы описываем экспериментальные этапы прохождения курсов времени флуоресценции с одной клеткой в формате LISCA. Мы трансфектируем клетки, прилипающие к микроструктурированному массиву, используя кодирование мРНК для усиленного зеленого флуоресцентного белка (eGFP), и отслеживаем кинетику экспрессии eGFP сотен клеток параллельно с помощью сканирующей покадровой микроскопии. Стеки данных изображения автоматически обрабатываются недавно разработанным программным обеспечением, которое интегрирует интенсивность флуоресценции по выбранным контурам клеток для создания одноэлементных курсов флуоресценции. Показано, что временные курсы экспрессии eGFP после трансфекции мРНК хорошо описываются простой кинетической моделью трансляции, которая выявляет скорость экспрессии и деградации мРНК. Обсуждаются дальнейшие применения LISCA для корреляций времени событий множественных маркеров в контексте сигнального апоптоза.
Introduction
В последние годы важность одноклеточных экспериментов стала очевидной. Данные от отдельных клеток позволяют проводить исследование межклеточной изменчивости, разрешение корреляций внутриклеточных параметров и обнаружение клеточной кинетики, которые остаются скрытыми в ансамблевых измерениях1,2,3. Для параллельного исследования клеточной кинетики тысяч одиночных клеток необходимы новые подходы, позволяющие контролировать клетки в стандартизированных условиях в течение периода времени от нескольких часов до нескольких дней с последующим количественным анализом данных 4. Здесь мы представляем Live-cell Imaging of Single-Cell Arrays (LISCA), которая сочетает в себе использование микроструктурированных массивов с покадровой микроскопией и автоматизированным анализом изображений.
Несколько методов генерации микроструктурированных одноклеточных массивов были установлены и опубликованы влитературе5,6. Здесь мы кратко опишем микромасштабное плазменно-инициированное белковое паттернирование (μPIPP). Подробный протокол изготовления одноэлеческого массива с использованием μPIPP также приведен в ссылке7. Использование одноклеточных массивов позволяет выровнять тысячи клеток по стандартизированным пятнам адгезии, представляющим определенные микросреды для каждой клетки, и, таким образом, уменьшает источник экспериментальной изменчивости(рисунок 1A). Одноклеточные массивы используются для мониторинга временных ходов флуоресцентных маркеров, направленных на указание на различные клеточные процессы. Долгосрочная микроскопия в режиме замедленной съемки сканирования позволяет контролировать большую площадь одноклеточных массивов и, следовательно, отбирать одноклеточные данные с высокой пропускной способностью в течение нескольких часов или даже дней. При этом генерируются стеки изображений из каждой позиции массива(рисунок 1B). Чтобы уменьшить большой объем данных изображения и эффективно извлечь желаемые одноэлементные курсы времени флуоресценции, требуется автоматизированная обработка изображений, которая использует преимущества позиционирования ячеек(рисунок 1C).
Задача LISCA состоит в том, чтобы адаптировать экспериментальные протоколы и вычислительные инструменты для формирования высокопроизводительного анализа, который генерирует количественные и воспроизводимые данные клеточной кинетики. В этой статье мы предоставляем пошаговое описание отдельных методов и того, как они сочетаются в анализе LISCA. В качестве примера мы обсуждаем временной ход экспрессии усиленного зеленого флуоресцентного белка (eGFP) после искусственной доставки мРНК. Экспрессия eGFP после доставки мРНК описывается уравнениями скорости реакции, моделируя трансляцию и деградацию мРНК. Подгонка функции модели для временного хода концентрации eGFP к показаниям LISCA интенсивности флуоресценции для каждой отдельной ячейки с течением времени дает наилучшие оценки параметров модели, таких как скорость деградации мРНК. В качестве репрезентативного результата мы обсуждаем эффективность доставки мРНК двух различных трансфекционных агентов на основе липидов и то, как различаются их распределения параметров.
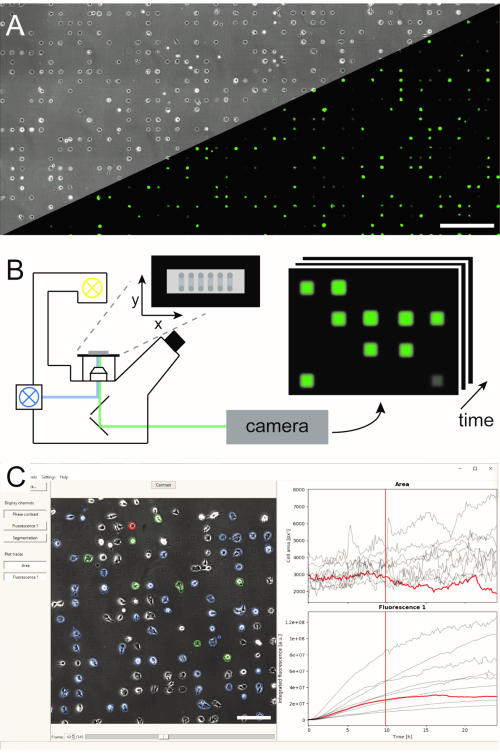
Рисунок 1:Представление рабочего процесса LISCA, сочетающего (A) микроструктурные одноклеточные массивы (B) сканирующей покадровой микроскопии и (C) автоматизированный анализ изображений записанных серий изображений. Одноклеточные массивы состоят из двумерного рисунка клеточно-адгезивных квадратов с клеточно-репеллентным межпространством, ведущим к расположению ячеек на микроструктуре, как видно на фазоконтрастном изображении, а также на флуоресцентном изображении эГФП-экспрессирующих клеток(А). Вся микроструктурированная область визуализируется в режиме сканирования покадровой съемки многократно, делая снимки в последовательности позиций(B). Записанные серии изображений обрабатываются для считывания интенсивности флуоресценции на ячейку с течением времени(C). Шкала стержней: 500 мкм(A),200 мкм(C). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
Здесь мы описали LISCA как универсальный метод для отслеживания клеточной кинетики внутриклеточных флуоресцентных меток на одноклеточном уровне. Для того, чтобы провести успешный эксперимент LISCA, каждый из описанных этапов раздела протокола должен быть установлен индивидуально, а зате?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Эта работа была поддержана грантами Немецкого научного фонда (DFG) Центру совместных исследований (SFB) 1032. Мы с благодарностью отмечаем поддержку Федерального министерства образования, исследований и технологий Германии (BMBF) в рамках совместного проекта 05K2018-2017-06716 Medisoft, а также грант от Bayerische Forschungsstiftung. Анита Рейзер была поддержана стипендией DFG через Высшую школу количественных биологических наук Мюнхена (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
References
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

