DNA-tethered RNA polymerase for programmerbar in vitro transkripsjon og molekylær beregning
Summary
Vi beskriver teknikk av en ny DNA-bundet T7 RNA-polymerase for å regulere in vitro transkripsjonsreaksjoner. Vi diskuterer trinnene for proteinsyntese og karakterisering, validerer konseptgodkjenningsregulering og diskuterer dens anvendelser innen molekylær databehandling, diagnostikk og molekylær informasjonsbehandling.
Abstract
DNA nanoteknologi muliggjør programmerbar selvmontering av nukleinsyrer i brukerforeskrevne former og dynamikk for ulike bruksområder. Dette arbeidet viser at begreper fra DNA nanoteknologi kan brukes til å programmere den enzymatiske aktiviteten til phage-avledede T7 RNA-polymerase (RNAP) og bygge skalerbare syntetiske genregulatoriske nettverk. For det første er en oligonukleotid-tethered T7 RNAP konstruert via uttrykk for en N-terminalt SNAP-merket RNAP og påfølgende kjemisk kobling av SNAP-taggen med en benzylguanin (BG)-modifisert oligonukleotid. Deretter brukes nukleinsyrestrengforskyvning til å programmere polymerasetranskripsjon på forespørsel. I tillegg kan hjelpekjernesyreenheter brukes som “kunstige transkripsjonsfaktorer” for å regulere interaksjonene mellom den DNA-programmerte T7 RNAP med sine DNA-maler. Denne in vitro-transkripsjonsregulatoriske mekanismen kan implementere en rekke kretsoppførseler som digital logikk, tilbakemelding, kaskade og multipleksing. Komponerbarheten av denne genregulatoriske arkitekturen letter designabstraksjon, standardisering og skalering. Disse funksjonene vil muliggjøre rask prototyping av in vitro genetiske enheter for applikasjoner som bio-sensing, sykdomsdeteksjon og datalagring.
Introduction
DNA-databehandling bruker et sett med designet oligonukleotider som medium for beregning. Disse oligonukleotidene er programmert med sekvenser for dynamisk montering i henhold til brukerspesifikk logikk og reagerer på spesifikke nukleinsyreinnganger. I konseptgodkjenningsstudier består utgangen av beregningen vanligvis av et sett fluorescerende merket oligonukleotider som kan oppdages via gelelektroforese eller fluorescensplatelesere. I løpet av de siste 30 årene har stadig mer komplekse DNA-beregningskretser blitt demonstrert, for eksempel ulike digitale logikkkaskader, kjemiske reaksjonsnettverk og nevrale nettverk1,2,3. For å hjelpe til med utarbeidelsen av disse DNA-kretsene, har matematiske modeller blitt brukt til å forutsi funksjonaliteten til syntetiske genkretser4,5og beregningsverktøy er utviklet for ortogonal DNA-sekvensdesign6,7,8,9,10 . Sammenlignet med silisiumbaserte datamaskiner inkluderer fordelene med DNA-datamaskiner deres evne til å kommunisere direkte med biomolekyler, operere i løsning i fravær av strømforsyning, samt deres generelle kompaktitet og stabilitet. Med ankomsten av neste generasjons sekvensering har kostnadene ved å syntetisere DNA-datamaskiner gått ned de siste to tiårene med en hastighet raskere enn Moores lov11. Anvendelser av slike DNA-baserte datamaskiner begynner nå å dukke opp, for eksempel for sykdomsdiagnose12,13, for å drive molekylær biofysikk14, og som datalagringsplattformer15.
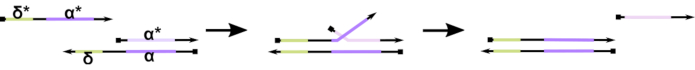
Figur 1: Mekanisme for tåholdmediert DNA-strengforskyvning. Tåholdet, δ, er en gratis, ubundet sekvens på en delvis dupleks. Når et komplementært domene (δ*) innføres på en annen tråd, fungerer det frie δ-domenet som et tåhold for hybridisering, slik at resten av tråden (ɑ*) sakte kan fortrenge konkurrenten gjennom en zipping / unzipping reversibel reaksjon kjent som strandmigrasjon. Etter hvert som lengden på δ øker, reduseres ΔG for fremoverreaksjonen, og forskyvning skjer lettere. Klikk her for å se en større versjon av denne figuren.
Til dags dato bruker flertallet av DNA-datamaskiner et veletablert motiv innen dynamisk DNA nanoteknologi kjent som tåholdmediert DNA-strandforskyvning (TMDSD, figur 1)16. Dette motivet består av en delvis dobbeltstrenget DNA (dsDNA) dupleks som viser korte “tåhold” overheng (dvs. 7- til 10 nukleotider (nt)). Nukleinsyre “input” tråder kan samhandle med de delvise dupleksene gjennom tåholdet. Dette fører til forskyvning av en av trådene fra den delvise dupleksen, og denne frigjorte strengen kan deretter tjene som inngang for nedstrøms delvise duplekser. Dermed muliggjør TMDSD signalkaskade og informasjonsbehandling. I prinsippet kan ortogonale TMDSD-motiver operere uavhengig i løsning, noe som muliggjør parallell informasjonsbehandling. Det har vært en rekke variasjoner på TMDSD-reaksjonen, for eksempel tåholdmediert DNA-strandutveksling (TMDSE)17, “lekkasjeløse” tåhold med dobbeltlange domener18, sekvens-mismatched tåhold19, og “handhold”-mediert strandforskyvning20. Disse innovative designprinsippene gir mer finjustert TMDSD-energi og dynamikk for å forbedre YTELSEN til DNA-databehandling.
Syntetiske genkretser, som transkripsjonelle genkretser, er også i stand til beregning21,22,23. Disse kretsene er regulert av proteintranskripsjonsfaktorer, som aktiverer eller undertrykker transkripsjon av et gen ved å binde seg til spesifikke regulatoriske DNA-elementer. Sammenlignet med DNA-baserte kretser har transkripsjonskretser flere fordeler. For det første har enzymatisk transkripsjon en mye høyere omsetningshastighet enn eksisterende katalytiske DNA-kretser, og genererer dermed flere kopier av utdata per enkelt kopi av inngang og gir et mer effektivt middel for signalforsterkning. I tillegg kan transkripsjonskretser produsere forskjellige funksjonelle molekyler, for eksempel aptamers eller messenger RNA (mRNA) koding for terapeutiske proteiner, som beregningsutganger, som kan utnyttes til forskjellige applikasjoner. Imidlertid er en stor begrensning av nåværende transkripsjonskretser deres mangel på skalerbarhet. Dette er fordi det er et svært begrenset sett med ortogonale proteinbaserte transkripsjonsfaktorer, og de novo-design av nye proteintranskripsjonsfaktorer forblir teknisk utfordrende og tidkrevende.
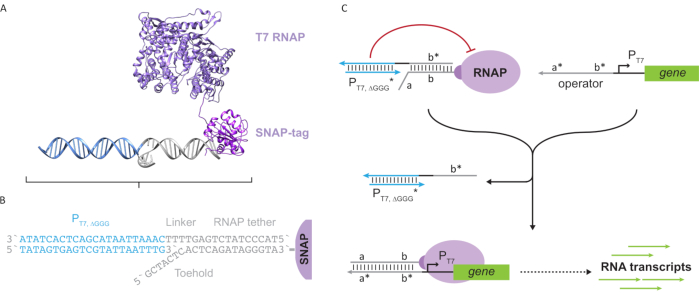
Figur 2: Abstraksjon og mekanisme for polymerasekomplekset “tether” og “cage”. (A og B) En oligonukleotidetets tether er enzymatisk merket til en T7-polymerase gjennom SNAP-tag-reaksjonen. Et bur bestående av en “faux” T7-promotor med et tether-komplementoverheng gjør det mulig å hybridisere til tether og blokkere transkripsjonsaktivitet. (C) Når operatoren (a*b*) er til stede, binder den seg til tåholdet på oligonukleotidets tether (ab) og fortrenger b* -regionen i buret, slik at transkripsjon kan forekomme. Denne figuren er endret fra Chou og Shih27. Forkortelser: RNAP = RNA polymerase. Klikk her for å se en større versjon av denne figuren.
Dette dokumentet introduserer en ny byggestein for molekylær databehandling som kombinerer funksjonaliteten til transkripsjonskretser med skalerbarheten til DNA-baserte kretser. Denne byggesteinen er en T7 RNAP som er kovalent festet med en enkeltstrenget DNA-tether (figur 2A). For å syntetisere denne DNA-tethered T7 RNAP, ble polymerasen smeltet sammen til en N-terminal SNAP-tag24 og rekombinant uttrykt i Escherichia coli. SNAP-taggen ble deretter reagert med et oligonukleotid funksjonalisert med BG-substratet. Oligonukleotid-tetheren tillater posisjonering av molekylære gjester i nærheten av polymerasen via DNA-hybridisering. En slik gjest var en konkurransedyktig transkripsjonsblokkering referert til som et “bur”, som består av en “faux” T7 promotør DNA-dupleks uten gen nedstrøms (Figur 2B). Når det bindes til RNAP via sin oligonukleotid-tether, stopper buret polymeraseaktivitet ved å utkonkurrere andre DNA-maler for RNAP-binding, noe som gjør RNAP i en “OFF” -tilstand (Figur 2C).
For å aktivere polymerasen til en “ON” tilstand, ble T7 DNA-maler med enkeltstrengede “operatør” domener oppstrøms for T7-promotoren av genet designet. Operatørdomenet (dvs. domene a*b* figur 2C) kan utformes for å fortrenge merden fra RNAP via TMDSD og plassere RNAP-proksimalt til T7-promotoren av genet, og dermed initiere transkripsjon. Alternativt ble DNA-maler også designet der operatørsekvensen var komplementær til hjelpekjernesyrestrenger som kalles “kunstige transkripsjonsfaktorer” (dvs. TFA- og TFB-tråder i figur 3A). Når begge trådene introduseres i reaksjonen, samles de på operatørstedet og oppretter et nytt pseudo-sammenhengende domene a*b*. Dette domenet kan deretter fortrenge merden via TMDSD for å starte transkripsjon (Figur 3B). Disse trådene kan leveres enten eksogent eller produseres.
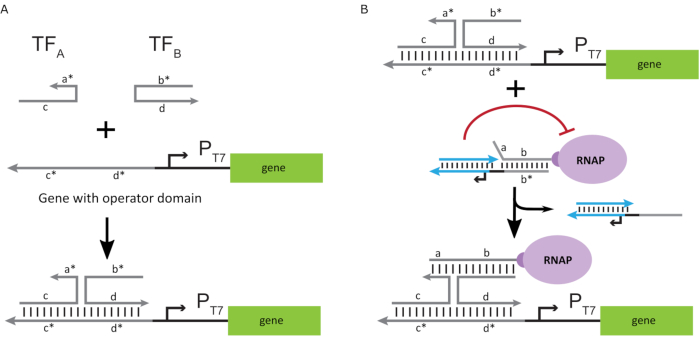
Figur 3: Selektiv programmering av polymeraseaktivitet gjennom en tre-komponent bryteraktivator. (A) Når transkripsjonsfaktorene (TFA og TFB) er til stede, binder de seg til operatørdomenet oppstrøms for promotoren, og danner en pseudo-enkeltstrenget sekvens ( a *b *) som er i stand til å fortrenge buret gjennom tåhold mediert DNA-forskyvning. (B) Dette a * b * domenet kan fortrenge buret via TMDSD for å starte transkripsjon. Denne figuren er endret fra Chou og Shih27. Forkortelser: TF = transkripsjonsfaktor; RNAP = RNA polymerase; TMDSD = tåholdmediert DNA-strengforskyvning. Klikk her for å se en større versjon av denne figuren.
Bruken av nukleinsyrebaserte transkripsjonsfaktorer for in vitro transkripsjonsregulering tillater skalerbar implementering av sofistikert kretsatferd som digital logikk, tilbakemelding og signalkaskade. For eksempel kan man bygge logikk gate kaskader ved å designe nukleinsyre sekvenser slik at transkripsjoner fra et oppstrøms gen aktiverer et nedstrøms gen. Et program som utnytter kaskade- og multipleksing gjort i stand til denne foreslåtte teknologien er utviklingen av mer sofistikerte molekylære databehandlingskretser for bærbar diagnostikk og molekylær databehandling. I tillegg kan integrering av molekylær databehandling og de novo RNA-syntesefunksjoner muliggjøre nye applikasjoner. For eksempel kan en molekylær krets utformes for å oppdage en eller en kombinasjon av brukerdefinerte RNAer som innganger og utgangsterapeutiske RNAer eller mRNAer som koder funksjonelle peptider eller proteiner for medisinske applikasjoner.
Protocol
Representative Results
Discussion
Denne studien demonstrerer en DNA nanoteknologi-inspirert tilnærming for å kontrollere aktiviteten til T7 RNA-polymerase ved å kovalent koble en N-terminalt SNAP-merket rekombinant T7 RNAP med en BG-funksjonalisert oligonukleotid, som senere ble brukt til å programmere TMDSD-reaksjoner. Ved design ble SNAP-taggen plassert ved polymerasens N-terminus, da C-terminus av wild-type T7 RNAP er begravet i proteinstrukturkjernen og gjør viktige kontakter med DNA-malen28. Tidligere forsøk på å modi…
Divulgations
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C anerkjenner sjenerøs støtte fra New Frontiers in Research Fund-Exploration (NFRF-E), Natural Sciences and Engineering Research Council of Canada (NSERC) Discovery Grant, og University of Toronto’s Medicine by Design Initiative, som mottar støtte fra Canada First Research Excellence Fund (CFREF).
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
References
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Biochimie. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

