DNA-tøjret RNA Polymerase til programmerbar In vitro transskription og molekylær beregning
Summary
Vi beskriver ingeniørien af en ny DNA-tøjret T7 RNA polymerase til regulering af in vitro transskription reaktioner. Vi diskuterer trinene til proteinsyntese og karakterisering, validere proof-of-concept transskriptionel regulering, og diskutere dens anvendelser i molekylær computing, diagnostik, og molekylær informationsbehandling.
Abstract
DNA nanoteknologi muliggør programmerbar selvmontering af nukleinsyrer i brugerordinerede former og dynamikker til forskellige applikationer. Dette arbejde viser, at begreber fra DNA nanoteknologi kan bruges til at programmere den enzymatiske aktivitet af phage-afledt T7 RNA polymerase (RNAP) og opbygge skalerbare syntetiske genregulerende netværk. For det første konstrueres en RNAP,der er bundet af RNAP med oliigonuleotid, via udtryk for en N-terminalt SNAP-mærket RNAP og efterfølgende kemisk kobling af SNAP-mærket med en benzylguanine (BG)-modificeret oligonukleotid. Dernæst bruges nukleinsyrestrengforskydning til at programmere polymerasetransskription on-demand. Derudover kan hjælpekernekernesyresamlinger bruges som “kunstige transskriptionsfaktorer” til at regulere samspillet mellem den DNA-programmerede T7 RNAP med sine DNA-skabeloner. Denne in vitro transskription reguleringsmekanisme kan gennemføre en række kredsløb adfærd såsom digital logik, feedback, kaskade, og multiplexing. Sammensætningen af denne genregulerende arkitektur letter design abstraktion, standardisering og skalering. Disse funktioner vil gøre det muligt hurtigt at prototyping af in vitro genetiske anordninger til applikationer såsom bio-sensing, sygdomsdetektering, og datalagring.
Introduction
DNA computing bruger et sæt designet oligonukleotider som medium til beregning. Disse oligonukleotider er programmeret med sekvenser til dynamisk at samle i henhold til brugerdefineret logik og reagere på specifikke nukleinsyreinput. I proof-of-concept-undersøgelser består beregningens output typisk af et sæt fluorescerende mærkede oligonuleotider, der kan påvises via gelelektroforese eller fluorescenspladelæsere. I løbet af de sidste 30 år er stadig mere komplekse DNA-beregningskredsløb blevet påvist, såsom forskellige digitale logiske kaskader, kemiske reaktionsnetværk og neurale netværk1,2,3. For at hjælpe med udarbejdelsen af disse DNA-kredsløb er matematiske modeller blevet brugt til at forudsige funktionaliteten af syntetiske genkredsløb4,5og beregningsværktøjer er udviklet til ortogonalt DNA-sekvensdesign6,7,8,9,10 . Sammenlignet med siliciumbaserede computere omfatter fordelene ved DNA-computere deres evne til at kommunikere direkte med biomolekyler, fungere i opløsning i mangel af en strømforsyning samt deres samlede kompakthed og stabilitet. Med fremkomsten af næste generations sekventering, omkostningerne ved at syntetisere DNA-computere har været faldende i de sidste to årtier med en hastighed hurtigere end Moores lov11. Anvendelser af sådanne DNA-baserede computere er nu begyndt at dukke op, såsom for sygdomsdiagnose12,13, til kraftoverførsel molekylær biofysik14,og som datalagring platforme15.
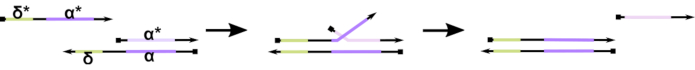
Figur 1: Mekanisme for toehold-medieret DNA-strengforskydning. Toehold, δ, er en gratis, ubundet sekvens på en delvis duplex. Når et supplerende domæne (δ*) introduceres på en anden streng, fungerer det frie δ domæne som et fodfæste for hybridisering, hvilket giver mulighed for, at resten af strengen (ɑ*) langsomt fortrænger sin konkurrent gennem en zippe / unzipping reversibel reaktion kendt som strengmigration. Efterhånden som længden af δ øges, falder ΔG for fremadrettede reaktioner, og forskydning sker lettere. Klik her for at se en større version af dette tal.
Til dato anvender de fleste DNA-computere et veletableret motiv inden for dynamisk DNA-nanoteknologi kendt som toehold-medieret DNA-strengforskydning (TMDSD, figur 1)16. Dette motiv består af en delvist dobbeltstrenget DNA (dsDNA) duplex, der viser korte “toehold” udhæng (dvs. 7- til 10 nukleotider (nt)). Nukleinsyre “input” tråde kan interagere med den delvise duplexes gennem fodfæste. Dette fører til forskydning af en af strengene fra den delvise duplex, og denne frigjorte streng kan derefter tjene som input til downstream delvise duplexes. Således muliggør TMDSD signalkascading og informationsbehandling. I princippet kan ortogonale TMDSD-motiver fungere uafhængigt i opløsning, hvilket muliggør parallel informationsbehandling. Der har været en række variationer over TMDSD-reaktionen, såsom toehold-medieret DNA-strengudveksling (TMDSE)17, “leakless” toeholds med dobbeltlange domæner18, sekvens-mismatched toeholds19og “handhold”-medieret strengforskydning20. Disse innovative designprincipper giver mulighed for mere finjusterede TMDSD-energiske og dynamikker til forbedring af DNA-databehandlingens ydeevne.
Syntetiske genkredsløb, såsom transskriptionsgenkredsløb, er også i stand til beregning21,22,23. Disse kredsløb er reguleret af protein transskription faktorer, som aktiverer eller undertrykke transskription af et gen ved at binde sig til specifikke regulatoriske DNA-elementer. Sammenlignet med DNA-baserede kredsløb har transskriptionskredsløb flere fordele. For det første har enzymatisk transskription en meget højere omsætningshastighed end eksisterende katalytiske DNA-kredsløb, hvilket genererer flere kopier af output pr. enkelt kopi af input og giver et mere effektivt middel til signalforstærkning. Derudover kan transskriptionskredsløb producere forskellige funktionelle molekyler, såsom aptamers eller messenger RNA (mRNA) kodning til terapeutiske proteiner, som beregningsoutput, som kan udnyttes til forskellige applikationer. Men en stor begrænsning af de nuværende transskriptionskredsløb er deres manglende skalerbarhed. Dette skyldes, at der er et meget begrænset sæt ortogonale proteinbaserede transskriptionsfaktorer, og de novo-design af nye proteintransskriptionsfaktorer er fortsat teknisk udfordrende og tidskrævende.
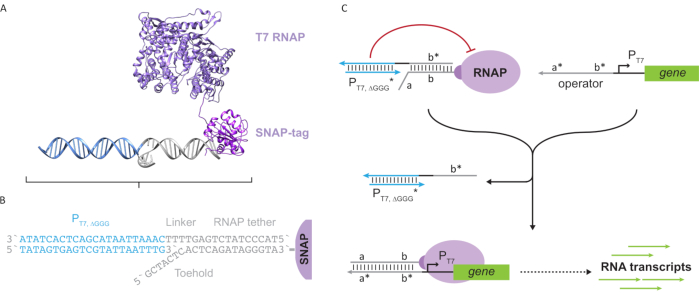
Figur 2: Abstraktion og mekanisme af “tether” og “bur” polymerase kompleks. (A og B) En oligonuleotid tether er enzymatisk mærket til en T7 polymerase gennem SNAP-tag reaktion. Et bur bestående af en “faux” T7 promotor med en tether-supplement udhæng gør det muligt at hybridisere til tøjre og blokere transskriptionelle aktivitet. (C) Når operatøren (a*b*) er til stede, binder den sig til fodfæste på oligonucleotidsbindingen(ab) og fortrænger burets b*-region, således at transskriptionen kan forekomme. Dette tal er blevet ændret fra Chou og Shih27. Forkortelser: RNAP = RNA polymerase. Klik her for at se en større version af dette tal.
Dette papir introducerer en ny byggesten til molekylær databehandling, der kombinerer funktionaliteterne i transskriptionskredsløb med skalerbarheden af DNA-baserede kredsløb. Denne byggesten er en T7 RNAP kovalent fastgjort med en enkeltstrenget DNA-tether (Figur 2A). For at syntetisere denne DNA-tøjrede T7 RNAP blev polymerase smeltet til en N-terminal SNAP-tag24 og rekombinant udtrykt i Escherichia coli. SNAP-mærket blev derefter reageret med et oligonuleotid, der blev funktionaliseret med BG-substratet. Oligonucleotid tether tillader positionering af molekylære gæster i nærheden af polymerase via DNA hybridisering. En sådan gæst var en konkurrencedygtig transskriptionsblokker kaldet et “bur”, som består af en “faux” T7 promotor DNA-duplex uden gen nedstrøms (Figur 2B). Når buret er bundet til RNAP via sinoligonucleotid tether, bremser det polymeraseaktiviteten ved at udkonkurrere andre DNA-skabeloner til RNAP-binding, hvilket gør RNAP i en “OFF”-tilstand (figur 2C).
For at aktivere polymerase til en “ON” tilstand blev T7 DNA-skabeloner med enkeltstrengede “operatør” domæner opstrøms af T7-promotoren af genet designet. Operatøren domæne (dvs. domæne a * b * Figur 2C) kan være designet til at fortrænge buret fra RNAP via TMDSD og placere RNAP proksimale til T7 promotor af genet, og dermed indlede transskription. Alternativt blev DNA-skabeloner også designet, hvor operatørsekvensen var et supplement til hjælpekernekernesalve, der kaldes “kunstige transskriptionsfaktorer” (dvs. TFA- og TFB-strenge i figur 3A). Når begge tråde introduceres i reaktionen, samles de på operatørwebstedet og opretter et nyt pseudo-sammenhængende domæne a * b *. Dette domæne kan derefter fortrænge buret via TMDSD for at indlede transskription (Figur 3B). Disse tråde kan leveres enten eksogent eller produceres.
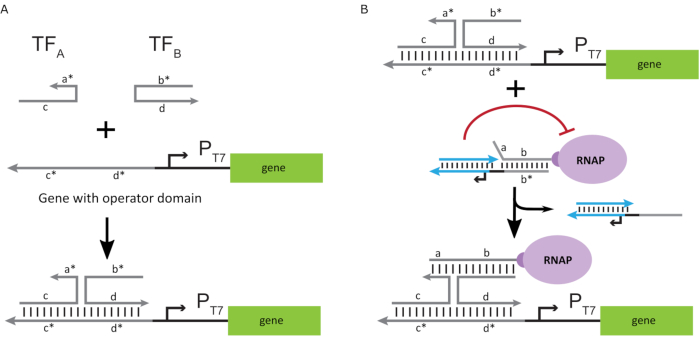
Figur 3: Selektiv programmering af polymeraseaktivitet gennem en trekomponent switch-aktivator. (A) Når transskriptionsfaktorerne (TFA og TFB) er til stede, binder de sig til operatordomænet opstrøms promotoren og danner en pseudostrenget sekvens (a*b*), der kan fortrænge buret gennem toehold medieret DNA-forskydning. (B) Dette a * b * domæne kan fortrænge buret via TMDSD at indlede transskription. Dette tal er blevet ændret fra Chou og Shih27. Forkortelser: TF = transskriptionsfaktor; RNAP = RNA polymerase; TMDSD = toehold-medieret DNA-strengforskydning. Klik her for at se en større version af dette tal.
Brugen af nukleinsyre-baserede transskription faktorer for in vitro transskriptionion regulering tillader skalerbar gennemførelse af sofistikerede kredsløb adfærd såsom digital logik, feedback, og signal cascading. For eksempel kan man bygge logik gate kaskader ved at designe nukleinsyre sekvenser således, at udskrifter fra en opstrøms gen aktivere en downstream gen. Et program, der udnytter kaskade og multiplexing gjort i stand til af denne foreslåede teknologi er udviklingen af mere sofistikerede molekylære computing kredsløb til bærbare diagnostik og molekylær databehandling. Derudover kan integration af molekylær databehandling og de novo RNA-syntesefunktioner muliggøre nye applikationer. For eksempel kan et molekylært kredsløb være designet til at detektere en eller en kombination af brugerdefinerede RNA’er som input og output terapeutiske RNA’er eller mRNAs, der kodning funktionelle peptider eller proteiner til punkt-of-care medicinske applikationer.
Protocol
Representative Results
Discussion
Denne undersøgelse viser en DNA nanoteknologi-inspireret tilgang til at kontrollere aktiviteten af T7 RNA polymerase ved kovalent kobling en N-terminalt SNAP-tagged rekombinant T7 RNAP med en BG-funktionaliseret oligonukleotid, som efterfølgende blev brugt til at programmere TMDSD reaktioner. Ved design blev SNAP-tag placeret på N-terminus af polymerase, da C-endestationen for vild type T7 RNAP er begravet i proteinstrukturkernen og gør vigtige kontakter med DNA-skabelonen28. Tidligere forsøg…
Divulgations
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C anerkender generøs støtte fra New Frontiers in Research Fund-Exploration (NFRF-E), Natural Sciences and Engineering Research Council of Canada (NSERC) Discovery Grant og University of Toronto’s Medicine by Design Initiative, som modtager støtte fra Canada First Research Excellence Fund (CFREF).
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
References
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Biochimie. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

