Anopheles 감비아에서 기능 유전학을 위한 GAL4-UAS 시스템을 이용하십시오
Summary
이 양파르트 갈4-UAS 시스템은 Anopheles 감비아에서 기능성 유전 분석을 허용하는 제어된 스파티오 측량 방식으로 유전자 발현을 수정하기 위한 다목적 도구입니다. 이 시스템을 사용하기 위해 설명된 절차는 형광 단백질 마커 및 배아 고정을 위한 pupae의 반 표준화된 복제 전략, 섹스링 및 검열입니다.
Abstract
이중 형 GAL4-UAS 시스템은 기능성 유전 분석을위한 다재다능하고 강력한 도구입니다. 시스템의 본질은 조직 특정 방식으로 효모 전사 인자 GAL4를 표현하는 형질전환 ‘드라이버’ 라인을 교차하는 것으로, GAL4를 결합하는 업스트림 활성화 서열(UAS)에 의해 발현이 조절되는 후보 유전자/RNA 간섭 구조를 운반하는 형질형 ‘응답자’ 라인을 갖는다. 이어지는 자손에서, 유전자 또는 침묵 구조는 이렇게 규정된 현면 방식으로 발현되어 결과 표현형을 분석하고 유전자 기능을 유추할 수 있게 한다. 이 바이너리 시스템은 심한 피트니스 비용이 유도되더라도 여러 조직 별 패턴에서 트랜스진 발현에 의해 생성된 표현형을 선별하는 실험적 접근법의 유연성을 가능하게 한다. 우리는 Anopheles 감비아에 대 한이 시스템을 적응, 아프리카에 있는 주요 말라리아 벡터.
이 문서에서는 GAL4-UAS 분석 중에 사용되는 몇 가지 일반적인 절차를 제공합니다. 우리는 이미 생성 된 An. 감비아 GAL4-UAS 라인뿐만 아니라 업 규제 및 RNAi 노크다운을위한 새로운 응답자 구조의 복제를 설명합니다. 우리는 유전 십자가를 확립하기 위하여 모기 강아지의 성화를 위한 단계별 가이드를 지정합니다, 또한 드라이버와 응답자 삽입을 태그하는 형광 유전자 마커의 상속을 따르는 검열 자성을 포함합니다. 우리는 또한 배아 발달을 연구하기 위하여 An. 감비아 어 태아를 지우기 위한 프로토콜을 제시합니다. 마지막으로, 대상 유전자의 GAL4 다운스트림의 CRISPR/Cas9 삽입을 통해 드라이버 라인을 생성하는 방법의 잠재적 인 적응을 소개합니다.
Introduction
이 양파르트-UAS 시스템은 곤충 모델 유기체 Drosophila melanogaster1,2,3에서 유전자의 기능적 특성화의 주력이다. GAL4-UAS 시스템을 사용하기 위해, 변성 드라이버 라인은, 규제 서열의 통제하에 효모 전사 인자 GAL4를 표현하고, GAL4에 의해 인식되는 업스트림 활성화 서열(UAS)에 의해 제어되는 관심 또는 RNA 간섭(RNAi) 구조의 유전자를 운반하는 응답자 라인과 교차된다. 본 십자가의 자손은 GAL4 발현을 제어하는 프로모터에 의해 지시된 현면 패턴에 대한 관심의 변형을 표현한다(도 1). 운전자-응답자 십자가의 자손에 의해 표시되는 표현형은 후보 유전자의 기능을 해명하기 위하여 평가될 수 있다. D. 멜라노가스터는 다른 유기체로부터 유전자를 검사하는 데 사용되었지만4,5,6,7, GAL4-UAS 시스템은 이제 관심있는 종에 대한 직접 분석을 제공하기 위해 의료 및 농업 중요성의 곤충에 사용하기 위해 적응되어 8,9,9,10,11,12,13,14.
아프리카 말라리아 모기에서, Anopheles 감비아, GAL4-UAS 시스템은 세포주 공동 형질제9에 의해 처음으로 시험되었습니다. 여러 개의 구조는 서로 다른 쌍방향 조합의 효율성을 위해 분석되었으며, 14개의 UAS가 작은 인공 인트론(UAS-14i)으로 보충되어 GAL4 드라이버 패널과 함께 사용할 때 가장 광범위한 활성화 잠재력을 나타낸다는 것을 발견했습니다. 생체 내 기능을 입증하기 위해, 이러한 구조는 PiggyBac 변환8에 의해 두 개의 별도의 형질형 An. 감비아 라인을 만드는 데 사용되었다: midgut 특정 프로모터에 의해 구동 GAL4를 운반하는 드라이버 라인, 그리고 UAS 서열의 규정에 따라 루시파라제및 향상된 황색 형광 단백질 (eYFP) 유전자를 모두 포함하는 응답자 라인. 자손의 직감 특이적 루시파제 활동과 형광은 시스템이 Anopheles에서 효율적이라고 지적했다. 그 이후, 운전자 라인은 이노뉴테15 및 혈구16을 포함한 벡터 용량 및 살충제 저항에 중요한 다른 조직에서 전경과를 발현하고, 유비쿼터스 패턴10에 가깝게 만들어졌다. 수많은 UAS 라인은 또한 대사 및 격리 중재 살충제 저항, 큐티칸 탄화수소 합성 및 형광 태그 다른 세포 및 조직 유형 (표 1)에 관여하는 것으로 생각 되는 분석 유전자에 생성 되었습니다. 응답자 라인의 경우, 이제 ΦC31 촉매 재조합 카세트 교환17,18에 의해 생성되어 UAS 규제 유전자의 게놈 컨텍스트를 수정하여 트랜스진의 현장 지향적 통합이 수행된다. 이러한 방식으로, 유전자 발현은 게놈 삽입 위치에 대해 정상화되어 상이한 후보 유전자의 표현 효과보다 정확한 비교를 가능하게 한다.
현재까지 생성된 응답자 라인은 높은 수준에서 트랜스진을 발현하거나 RNA 간섭(RNAi)을 통해 유전자 발현을 감소시키도록 설계되었습니다. 일반적으로 cDNA 클론은 UAS 서열에 융합되어 적절한 발현 플라스미드를 생성하지만 전체 게놈 서열도 복제에 너무 크지 않다고 가정할 수 있습니다. 침묵 구조를 생성하기 위해, 우리는 RNAi를 자극하는 헤어 핀 dsRNA를 형성하는 적절한 탠덤 반전 시퀀스를 얻기 위해 세 가지 다른 방법을 사용했다. 여기에는 융합 PCR, 비대칭 PCR 및 헤어핀 구조의 상업적 합성이 포함되었습니다. 각 방법에 공통적으로 복제 안정성을 제공하기 위해 반전된 시퀀스 사이에 인트론 시퀀스를 포함시키는 것이 일반적이다. 관심/RNAi 구조유전자를 삽입할 수 있는 응답자 플라스미드가 개발되었다15. 이 플라스미드는 또한 RMCE에 필요한 ΦC31 attB 사이트를 수행합니다 (RCME 기술을 자세히 설명하는 JoVE 종이와 함께 아돌피에 설명). 과발현을 위해 이러한 플라스미드 중 하나에 삽입시퀀스를 선택할 때 필요한 중요한 단계를 다루는 프로토콜이 이 원고에 포함되어 있습니다. 또한 RNAi 헤어핀 구조 생성을 위한 두 가지 프로토콜이 설명되고 설명되어 있습니다.
새로운 라인을 만들 때, 희귀 한 형질 전환 개인의 식별은 설립하고 형질 전환 식민지를 유지하기 위해 번식하는 것이 중요합니다. 가장 중요한 것은 GAL4-UAS 시스템의 경우 응답자와 운전자 라인을 구별하여 교차를 설정하고 두 트랜스유전자를 모두 수행하는 개별 자손을 식별할 필요가 있습니다. 이것은 드라이버와 응답자 카세트에 연결된 다른 지배적인 선택 가능한 마커 유전자를 사용하여 달성됩니다. 가장 일반적으로 이들은 광학 필터를 사용하여 명확하게 구별되는 형광 마커 유전자입니다 (예를 들어, eYFP, eCFP, dsRed). 이 이상과 오염의 식별을 쉽게하기 때문에 마커는 알려진 신뢰할 수있는 실각 측량 패턴으로 표현되는 것이 중요합니다. 형광 마커 유전자 발현은 합성 3xP3 프로모터에 의해 일상적으로 조절되며, 이는 An. 감비아 개발의 모든 단계에서 눈과 복부 신경리아 특이적 발현을 일으킨다. 3xP3 에 의해 제어되는 형광 마커는 이 문서에 기재된 모든 변환 플라스미드에 포함되어 있습니다. 형광을 검사하는 데 사용되는 일반적인 방법을 자세히 설명하는 프로토콜 An. 감비아 에 푸파에 GAL4-UAS 라인이 여기에 포함되어 있습니다.
GAL4-UAS 시스템의 핵심 요소 중 하나는 차별화된 표시된 드라이버 및 응답자 라인을 교차해야 한다는 것입니다. 각 줄에서이 남성과 여성은 짝짓기 전에 분리해야합니다. 성인은 시력에 의해 쉽게 구별 할 수 있습니다, 그러나, 유전 십자가를 확립하기위한 것은 짝짓기가 발생하지 않았는지 확인하기 위해 성인 출현 전에 남녀를 분리하는 것이 합리적이다. 남성과 여성의 일반적인 크기 차이는 너무 가변적이며, 성결정의 효율적이고 신뢰할 수 있는 방법이 될 수 있다20. 대신 외부 생식기에 명확한 형태적 차이는 An. 감비아에서 섹스에 대한 신뢰할 수있는 기초를 제공합니다. 이 기사에서는 적절한 십자가를 세우기 위해 An. 감비아 푸를 섹스하는 신뢰할 수있는 방법을 설명합니다.
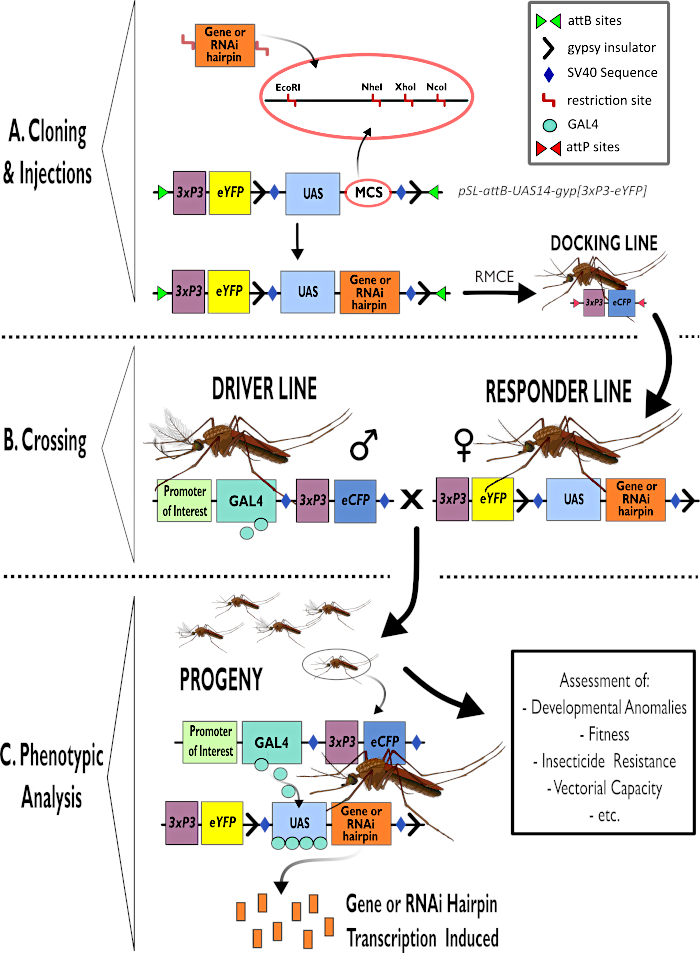
그림 1 – 아노펠스 감비아에서 이중 PARTite GAL4-UAS 시스템을 사용하기 위한 공정의 다이어그램 표현. (A) 예시 벡터(pSL-attB-UAS14-gyp[3xp3-eYFP]의 주요 구성 요소는 사용 가능한 제한 부위(EcoRI, NheI, XhoI 및 NcoI)를 상세히 묘사하며, 이는 관심 있는 유전자에 대한 헤어핀 구조 또는 코딩 서열을 삽입하는 데 사용하기에 적합한 다중 복제 부위 내에서 묘사된다. 도킹 라인의 구조도 묘사됩니다. (B) 교차 단계는 운전자 라인(3xP3 프로모터에 의해 구동되는 관심 및 eCFP의 발기인에 의해 GAL4 드라이버를 운반)과 응답자 라인에서 여성의 사용을 나타내는 것으로 도시된다(UAS 프로모터에 의해 제어되는 관심 또는 헤어핀 구조의 유전자를 운반하고 3xP3 프로모터에 의해 제어되는 eYFP 마커). (C) B에서 십자가의 자손에 대한 관심 유전자의 GAL4 구동 발현과 평가되는 전형적인 표현형의 일부 목록의 다이어그램 표현. 약어: 다중 복제 부위(MCS), 재조합 중재 카세트 교환(RMCE), 업스트림 액티베이터 서열(UAS), 향상된 황색 형광 단백질(eYFP), 향상된 시안 형광 단백질(eCFP). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그것은 더 선형 접근 에 비해 뚜렷한 장점을 가지고 GAL4-UAS 시스템의 쌍층 특성을 제공하는 십자가의 사용이다. 예를 들어, 드라이버 와 응답자 라인의 더 많은 조합은 새로운 형질 전환선이 생성되고 각 프로모터/유전자 조합에 대해 유지되어야 하는 경우 실현 가능한 것보다 평가될 수 있습니다. 더 중요한 것은, 선형 시스템에서 생성/유지가 어려운 발현이 위태로빙될 때 치명적또는 멸균 표현형을 생성하는 유전자의 분석을 허용한다. 이러한 치명적인 표현형은 유전자 기능 및 현면 발현에 따라 모든 발달 단계에서 나타날 수 있지만 배아 발달 중에 가장 자주 관찰됩니다. 모기 배아 발달을 시각화하려면 계란을 코팅하는 불투명한 초리온을 비우는 것이 필요합니다. Trpiš (1970)21 및 Kaiser 외.(2014)22에 기술된 방법에 따라, 우리는 구조적 무결성을 유지하면서 배아를 고치는 데 사용하는 프로토콜을 설명하고, 미세한 시각화 및 이미징을 허용하는 엔도토리온을 지우기 위해 표백합니다.
Protocol
Representative Results
Discussion
모기 유전자 기능을 이해하는 것은 Anopheles를 통제하고 말라리아 전송에 영향을 미치기 위하여 새로운 접근을 개발하는 것이 중요합니다. 설명된 GAL4-UAS 시스템은 후보 유전자의 기능적 분석을 위한 다재다능하고 강력한 시스템으로 현재까지 우리는 살충제 저항17 및 큐티큘러 탄화수소 생산의 유전적 기초를 검사하고, 형광태?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
우리는 감사로 LSTM과 IVCC (아드리아나 아돌피), BBSRC (새로운 조사자 상 (AL), MRC (BCP에 박사 학생 교직 박사 학생: MR / P016197/1), 웰컴 (경 헨리 웰컴 박사 후 박사 과정 펠로우십 LG: 215894/ Z/ 19/Z) 그 제안에 Gal4UAS 분석을 통합했습니다.
Materials
| 100 x 15 mm plastic Petri dish | SLS | 2175546 | Pack of 10 |
| 1000 µL Gilson Pipette | Gilson | F144059P | |
| 20/25 mL Universal Tubes | Starlab | E1412-3020 | Pack of 400 |
| 3 mL Pasteur Pipettes | SLS | G612398 | Greiner Pasteur pipette 3 mL sterile individually wrapped |
| 50 mL Falcon Tubes | Fisher Scientific | 11512303 | |
| Absolute Ethanol | Fisher Scientific | BP2818-500 | 500 mL |
| Acetic Acid | SLS | 45726-1L-F | 1 L |
| Cages | SLS | E6099 | 30x30x30 with screen port |
| Fine Paint Brushes | Amazon | UKDPB66 | KOLAMOON 9 Pieces Detail Painting Brush Set Miniture Brushes for Watercolor, Acrylic Painting, Oil Painting (Wine Red) |
| Fish food | Amazon | Tetra Min Fish Food, Complete Food for All Tropical Fish for Health, Colour and Vitality, 10 L | |
| Formaldehyde Solution | Sigma Aldrich | F8775 | |
| Mouth Aspirator | John Hock | 612 | |
| Pond Salt | Amazon | Blagdon Guardian Pond Tonic Salt, for Fish Health, Water Quality, General Tonic, pH Buffer, 9.08 kg, treats 9,092 L | |
| Pupae Pots | Cater4you | SP8OZ | 250 pots with lids |
| Small Plastic Buckets | Amazon | 2.5 L White Plastic Pail Complete with White Lid (Pack of 10) | |
| Sodium Hypochlorite | Fisher Scientific | S25552 |
References
- Brand, A. H., Perimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Duffy, J. B. GAL4 system in drosophila: A fly geneticist’s swiss army knife. Journal of Genetics and Development. 34 (1-2), 1-15 (2002).
- Dow, J. A. . ELS. , (2012).
- Edi, C. V., et al. CYP6 P450 Enzymes and ACE-1 Duplication Produce Extreme and Multiple Insecticide Resistance in the Malaria Mosquito Anopheles gambiae. PLoS Genetics. 10 (3), 1004236 (2014).
- Daborn, P. J., et al. Using Drosophila melanogaster to validate metabolism-based insecticide resistance from insect pests. Insect Biochemistry and Molecular Biology. 42 (12), 918-924 (2012).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. Genes Genomes Genetics. 7 (6), 1819-1832 (2017).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), (2014).
- Lynd, A., Lycett, G. J. Development of the Bi-Partite Gal4-UAS System in the African Malaria Mosquito, Anopheles gambiae. PLoS ONE. 7 (2), 31552 (2012).
- Lynd, A., Lycett, G. J. Optimization of the Gal4-UAS system in an Anopheles gambiae cell line. Insect Molecular Biology. 20 (5), 599-608 (2011).
- Adolfi, A., Pondeville, E., Lynd, A., Bourgouin, C., Lycett, G. J. Multi-tissue GAL4-mediated gene expression in all Anopheles gambiae life stages using an endogenous polyubiquitin promoter. Insect Biochemistry and Molecular Biology. 96, 1-9 (2018).
- Kokoza, V. A., Raikhel, A. A. Targeted gene expression in the transgenic Aedes aegypti using the binary Gal4-UAS system. Insect Biochemistry and Molecular Biology. 41, 637-644 (2011).
- O’Brochta, D. A., Pilitt, K. L., Harrell, R. A., Aluvihare, C., Alford, R. T. Gal4-based Enhancer-Trapping in the Malaria Mosquito Anopheles stephensi. Genes Genomes Genetics. 2, 21305-21315 (2012).
- Zhao, B., et al. Regulation of the Gut-Specific Carboxypeptidase: A Study Using the Binary Gal4/UAS System in the Mosquito Aedes Aegypti. Insect Biochemistry and Molecular Biology. 54, 1-10 (2014).
- Imamura, M., et al. Targeted Gene Expression Using the GAL4/UAS System in the Silkworm Bombyx mori. Génétique. 165 (3), 1329-1340 (2003).
- Lynd, A., et al. Development of a functional genetic tool for Anopheles gambiae oenocyte characterisation: application to cuticular hydrocarbon synthesis. bioRxiv. , (2019).
- Pondeville, E., et al. Hemocyte-targeted gene expression in the female malaria mosquito using the hemolectin promoter from Drosophila. Insect Biochemistry and Molecular Biology. 120, 103339 (2020).
- Adolfi, A., et al. Functional genetic validation of key genes conferring insecticide resistance in the major African malaria vector, Anopheles gambiae. Proceedings of the National Academy of Sciences of the United States of America. 116 (51), 25764-25772 (2019).
- Pondeville, E., et al. Efficient integrase-mediated site-specific germline transformation of Anopheles gambiae. Nature Protocols. 9 (7), 1698-1712 (2014).
- Horn, C., Schmid, B. G. M., Pogoda, F. S., Wimmer, E. A. Fluorescent transformation markers for insect transgenesis. Insect Biochemistry and Molecular Biology. 32, 1221-1235 (2002).
- Clements, A. . A. Biology of Mosquitoes, Volume 1: Development, Nutrition and Reproduction. 1, (1992).
- Trpiš, M. A new bleaching and decalcifying method for general use in zoology. Canadian Journal of Zoology. 48, 892-893 (1970).
- Kaiser, M. L., Duncan, F. D., Brooke, B. D. Embryonic Development and Rates of Metabolic Activity in Early and Late Hatching Eggs of the Major Malaria Vector Anopheles gambiae. PLoS ONE. 9 (12), 114381 (2014).
- Grigoraki, L., Grau-Bové, X., Yates, H. C., Lycett, G. J., Ranson, H. Isolation and transcriptomic analysis of Anopheles gambiae oenocytes enables the delineation of hydrocarbon biosynthesis. eLife. 9, 58019 (2020).
- Xiao, Y. -. H., Yin, M. -. H., Hou, L., Pei, Y. Direct amplification of intron-containing hairpin RNA construct from genomic DNA. BioTechniques. 41 (5), 548-552 (2006).
- Livak, K. J. Organization and Mapping of a Sequence on the Drosophila melanogaster X and Y Chromosomes That Is Transcribed during Spermatogenesis. Génétique. 107 (4), 611-634 (1984).
- MR4, CDC, NEI & beiResources. . The MR4 Methods in Anopheles Research Laboratory Manual. 5th Edition. , (2015).
- Sik Lee, Y., Carthew, R. W. Making a better RNAi vector for Drosophila: use of intron spacers. Methods. 30 (4), 322-329 (2003).
- Cha-aim, K., Hoshida, H., Fukunaga, T., Akada, R., Peccoud, J. . Gene Synthesis: Methods and Protocols. , 97-110 (2012).
- Cavener, D. R. Comparison of the consensus sequence flanking translational start sites in Drosophila and vertebrates. Nucleic Acids Research. 15 (4), 1353-1361 (1987).
- Wang, Y., Wang, F., Wang, R., Zhao, P., Xia, Q. 2A self-cleaving peptide-based multi-gene expression system in the silkworm Bombyx mori. Scientific Reports. 5, (2015).
- Galizi, R., et al. A synthetic sex ratio distortion system for the control of the human malaria mosquito. Nature Communications. 5, 3977 (2014).
- Kondo, S., et al. Neurochemical organisation of the Drosophila Brain Visualised by Endogenously Tagged Neurotransmitter Receptors. Cell Reports. 30 (1), 284-297 (2020).
- Lee, P. -. T., et al. A gene-specific T2A-GAL4 library for Drosophila. eLife. 7, 35574 (2018).
- Marois, E., et al. High-throughput sorting of mosquito larvae for laboratory studies and for future vector control interventions. Malaria Journal. 11, 302 (2012).
- Crawford, J. E., et al. Efficient production of male Wolbachia-infected Aedes aegypti mosquitoes enables large-scale suppression of wild populations. Nature Biotechnology. 38 (4), 482-492 (2020).
- Goltsev, Y., et al. Developmental and evolutionary basis for drought tolerance of the Anopheles gambiae embryo. Biologie du développement. 330 (2), 462-470 (2009).
- Rezende, G. L., et al. Embryonic desiccation resistance in Aedes aegypti: presumptive role of the chitinized Serosal Cuticle. BMC Developmental Biology. 8 (1), 82 (2008).
- Vargas, H. C. M., Farnesi, L. C., Martins, A. J., Valle, D., Rezende, G. L. Serosal cuticle formation and distinct degrees of desiccation resistance in embryos of the mosquito vectors Aedes aegypti, Anopheles aquasalis and Culex quinquefasciatus. Journal of Insect Physiology. 62, 54-60 (2014).
- Chang, C. -. H., et al. The non-canonical Notch signaling is essential for the control of fertility in Aedes aegypti. PLOS Neglected Tropical Diseases. 12 (3), 0006307 (2018).
- Clemons, A., Flannery, E., Kast, K., Severson, D., Duman-Scheel, M. Immunohistochemical Analysis of Protein Expression during Aedes aegypti Development. Spring Harbor Protocols. 10, 1-4 (2010).
- Juhn, J., James, A. A. Hybridization in situ of Salivary Glands, Ovaries and Embryos of Vector Mosquitoes. Journal of Visualized Experiments. , e3709 (2012).

