Usando o sistema GAL4-UAS para genética funcional em Anopheles gambiae
Summary
O sistema bipartite GAL4-UAS é uma ferramenta versátil para modificação da expressão genética de forma espástica controlada que permite a análise genética funcional em Anopheles gambiae. Os procedimentos descritos para o uso deste sistema são uma estratégia de clonagem semi-padronizada, sexagem e triagem de pupas para marcadores de proteína fluorescente e fixação de embriões.
Abstract
O sistema bipartite GAL4-UAS é uma ferramenta versátil e poderosa para análise genética funcional. A essência do sistema é cruzar linhas transgênicas de “driver” que expressam o fator de transcrição de leveduras GAL4 de forma específica do tecido, com linhas transgênicas de “responder” carregando uma construção de interferência genética/RNA candidata cuja expressão é controlada por Sequências de Ativação Upstream (UAS) que ligam GAL4. Na prole resultante, o gene ou a construção silenciante é, portanto, expressa de forma espesso prescrita, permitindo que os fenótipos resultantes sejam avaliados e a função genética inferida. O sistema binário permite flexibilidade em abordagens experimentais para tela fenótipos gerados pela expressão transgênica em múltiplos padrões específicos do tecido, mesmo que os custos severos de aptidão sejam induzidos. Adaptamos este sistema para Anopheles gambiae, o principal vetor de malária na África.
Neste artigo, fornecemos alguns dos procedimentos comuns utilizados durante a análise GAL4-UAS. Descrevemos as linhas An. gambiae GAL4-UAS já geradas, bem como a clonagem de novas construções de respondente para a regulação e o knockdown rnai. Especificamos um guia passo a passo para o sexing de pupas de mosquito para estabelecer cruzes genéticas, que também inclui a prole de triagem para seguir a herança de marcadores genéticos fluorescentes que marcam as inserções do motorista e do respondente. Também apresentamos um protocolo para limpar embriões de An. gambiae para estudar o desenvolvimento embrionário. Finalmente, introduzimos possíveis adaptações do método para gerar linhas de driver através da inserção CRISPR/Cas9 de genes alvo.
Introduction
O sistema bipartite GAL4-UAS é o cavalo de trabalho da caracterização funcional de genes no organismo modelo de insetos Drosophila melanogaster1,2,3. Para usar o sistema GAL4-UAS, as linhas de driver transgênicas, expressando o fator de transcrição da levedura GAL4 sob controle de uma sequência regulatória, são cruzadas com linhas de resposta carregando um gene de interesse ou interferência de RNA (RNAi) construída controlada por uma Sequência de Ativação Upstream (UAS) reconhecida pela GAL4. A descendência dessa cruz expressa o transgene de interesse em um padrão espítetempo ditado pelo promotor que controla a expressão GAL4 (Figura 1). Fenótipos exibidos por descendentes de cruzes de respondente condutor podem ser avaliados para elucidar a função dos genes candidatos. Embora d. melanogaster tenha sido usado para examinar genes de outros organismos4,5,6,7, o sistema GAL4-UAS foi agora adaptado para uso em insetos de importância médica e agrícola para fornecer análise direta nas espécies de interesse 8,9,10,11,12,13,14.
No mosquito africano da malária, Anopheles gambiae, o sistema GAL4-UAS foi testado pela primeira vez pela co-transfecção da linha celular9. Construções múltiplas foram avaliadas para eficiência em diferentes combinações de pairwise e descobriram que 14 UAS repetidas com um pequeno intron artificial (UAS-14i) exibiam a maior gama de potencial de ativação quando usado com um painel de drivers GAL4. Para demonstrar a funcionalidade in vivo, essas construções foram então usadas para criar duas linhas transgênicas de An. gambiae separadas pela Transformação PiggyBac8: uma linha de driver que transporta GAL4 conduzida por um promotor específico midgut, e uma linha de resposta contendo tanto a luciferase quanto os genes de proteína fluorescente amarela aprimorada (eYFP) sob regulação das sequências UAS. A atividade de luciferase específica intestinal e a fluorescência na prole indicaram que o sistema era eficiente em Anopheles. Desde então, foram criadas linhas de condutores expressando transgenes em outros tecidos importantes para a capacidade vetorial e resistência a inseticidas, incluindo oenocitos15 e hemócitos16, e em um padrão próximo ao onipresente10. Numerosas linhas de UAS também foram geradas para avaliar genes considerados envolvidos no metabolismo e sequestração mediada resistência a inseticidas, síntese de hidrocarbonetos cuticulares e para marcar fluorescente diferentes tipos de células e tecidos (Tabela 1). Para as linhas de resposta, a integração direcionada ao local do transgene é agora realizada pelo ΦC31 catalisado troca de cassete17,18 para corrigir o contexto genômico dos genes regulados pela UAS. Dessa forma, a expressão transgênica é normalizada em relação à localização de inserção genômica, permitindo uma comparação mais precisa dos efeitos fenotípicos de diferentes genes candidatos.
As linhas de respondente criadas até o momento são projetadas para expressar o transgene em níveis elevados ou reduzir a expressão genética através da interferência de RNA (RNAi). Normalmente, os clones cDNA são fundidos à sequência UAS para gerar plasmídeos de expressão adequados, porém sequências genômicas completas também são viáveis assumindo que eles não são muito grandes para clonagem. Para gerar construções de silenciamento, utilizamos três métodos diferentes para obter sequências invertidas tandem adequadas que formam o dsRNA que estimula o RNAi. Estes incluíram PCR de fusão, PCR assimétrico e síntese comercial de construções de grampos de cabelo. Comum a cada método é a inclusão de uma sequência intron entre as sequências invertidas para fornecer estabilidade de clonagem. Plasmids de resposta nos quais um gene de interesse/construção RNAi pode ser inserido foram desenvolvidos15. Esses plasmídeos também carregam os locais de attB ΦC31 necessários para rmce (descrito em Adolfi acompanhando o artigo jove que descreve a técnica RCME em detalhes). Protocolos que abrangem as etapas importantes necessárias ao selecionar a sequência para inserção em um desses plasmídeos para superexpressão estão incluídos neste manuscrito. Além disso, dois protocolos para criação de construção de grampos rnai são descritos e ilustrados.
Ao criar novas linhas, a identificação de indivíduos transgênicos raros é crucial para procriar para estabelecer e manter colônias transgênicas. Mais importante para o sistema GAL4-UAS, é necessário distinguir as linhas de respondente e motorista para estabelecer cruzes e identificar descendentes individuais que carregam ambos transgenes. Isso é conseguido usando diferentes genes de marcadores selecionáveis dominantes ligados às fitas do motorista e do respondente. Mais comumente estes são genes marcadores fluorescentes que são claramente distinguíveis usando filtros ópticos (por exemplo, eYFP, eCFP, dsRed). É importante que os marcadores sejam expressos em um padrão espíte conhecido e confiável, pois isso facilita a identificação de anormalidades e contaminação. A expressão genética do marcador fluorescente é rotineiramente regulada pelo promotor 3xP3 sintético, que causa expressão específica de gânglios oculares e ventral em todas as etapas do desenvolvimento de An. gambiae19. Marcadores fluorescentes controlados por 3xP3 estão incluídos em todos os plasmídeos de transformação descritos neste artigo. Um protocolo detalhando os métodos comuns usados para tela fluorescente Uma. gambiae pupae GAL4-UAS está incluído aqui.
Um dos elementos-chave do sistema GAL4-UAS é a necessidade de cruzar as linhas de driver e responder marcadas diferencialmente. Para fazer este macho e fêmeas de cada linha deve ser separado antes do acasalamento. Os adultos são facilmente distinguíveis pela visão, no entanto, para estabelecer cruzes genéticas é sensato separar os sexos antes do surgimento de adultos para garantir que o acasalamento não tenha ocorrido. A diferença geral de tamanho entre macho e fêmea An. gambiae pupae é muito variável para ser um método eficiente e confiável de determinação sexual20. Em vez disso, diferenças morfológicas claras na genitália externa fornecem uma base confiável para o sexing em An. gambiae. Neste artigo, descrevemos um método confiável para sexar An. gambiae pupae para configurar cruzes apropriadas.
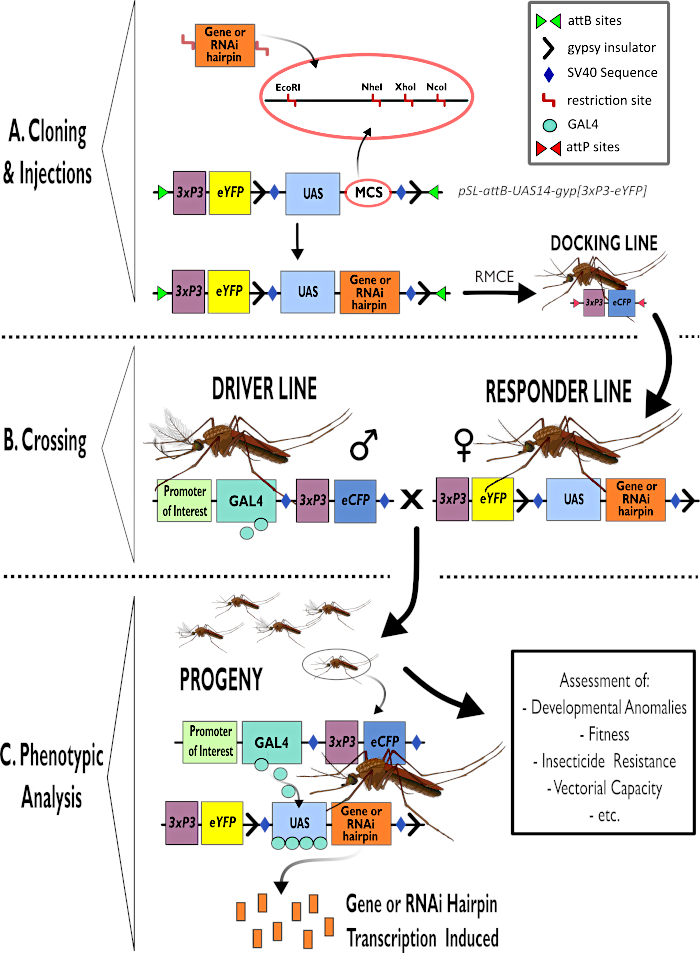
Figura 1 – Representação diagramática do processo para o uso do sistema bipartite GAL4-UAS em Anopheles gambiae. (A) Os principais componentes de um vetor de exemplo (pSL-attB-UAS14-gyp[3xp3-eYFP]) são retratados, detalhando os locais de restrição disponíveis (EcoRI, NheI, XhoI e NcoI) dentro dos múltiplos locais de clonagem que são adequados para uso para inserir a construção de grampos ou sequência de codificação para o interesse genético. A estrutura da linha de acoplamento também é retratada. (B) A etapa de travessia é ilustrada indicando o uso de machos da linha de motorista (transportando condutor gal4 por um promotor de interesse e eCFP conduzido pelo promotor 3xP3) e fêmeas da linha de resposta (carregando o gene de interesse ou construção de grampos controlado por um promotor da UAS e um marcador eYFP controlado pelo promotor 3xP3). (C) Uma representação diagramática da expressão de condução GAL4 do gene de interesse na prole da cruz em B e uma lista de alguns dos fenótipos típicos que são avaliados. Abreviaturas: Multiple Cloning Site (MCS), Recombinase mediated cassette exchange (RMCE), Upstream Activator Sequence (UAS), proteína fluorescente amarela aprimorada (eYFP), proteína fluorescente ciano aprimorada (eCFP). Clique aqui para ver uma versão maior desta figura.
É o uso de cruzes que fornece a natureza bipartite do sistema GAL4-UAS, que tem vantagens distintas sobre abordagens mais lineares. Por exemplo, muitas mais combinações de linhas de driver e respondente podem ser avaliadas do que seria viável se uma nova linha transgênica tivesse que ser gerada e mantida para cada combinação promotor/gene. Mais importante, permite a análise de genes que produzem fenótipos letais ou estéreis quando sua expressão é perturbada, que são difíceis de criar/manter em um sistema linear. Tais fenótipos letais podem se manifestar em todos os estágios de desenvolvimento, dependendo da função genética e expressão espacial, mas são mais frequentemente observados durante o desenvolvimento embrionário. Visualizar o desenvolvimento de embriões de mosquitos requer a limpeza da chorão opaca que reveste os ovos. Seguindo os métodos descritos em Trpiš (1970)21 e Kaiser et al. (2014)22, descrevemos os protocolos que usamos para corrigir embriões, mantendo a integridade estrutural e o branqueamento para limpar a endochorion que permite visualização e imagem microscópicas.
Protocol
Representative Results
Discussion
Entender a função genética do mosquito é vital para desenvolver novas abordagens para controlar anofelinos e impactar a transmissão da malária. O sistema GAL4-UAS descrito é um sistema versátil e poderoso para análise funcional de genes candidatos e até hoje usamos o sistema para examinar a base genética da resistência a inseticidas17 e produção de hidrocarbonetos cuticulares15,23, bem como para marcar fluorescentemente diferentes populações de células do<sup c…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Agradecemos o financiamento do LSTM e IVCC (Adriana Adolfi), BBSRC (New Investigator Award (AL), MRC (doutorado em BCP:MR/P016197/1), Wellcome (Bolsa de Pós-Doutorado Sir Henry Wellcome à LG: 215894/Z/19/Z) que incorporaram a análise da Gal4UAS nas propostas.
Materials
| 100 x 15 mm plastic Petri dish | SLS | 2175546 | Pack of 10 |
| 1000 µL Gilson Pipette | Gilson | F144059P | |
| 20/25 mL Universal Tubes | Starlab | E1412-3020 | Pack of 400 |
| 3 mL Pasteur Pipettes | SLS | G612398 | Greiner Pasteur pipette 3 mL sterile individually wrapped |
| 50 mL Falcon Tubes | Fisher Scientific | 11512303 | |
| Absolute Ethanol | Fisher Scientific | BP2818-500 | 500 mL |
| Acetic Acid | SLS | 45726-1L-F | 1 L |
| Cages | SLS | E6099 | 30x30x30 with screen port |
| Fine Paint Brushes | Amazon | UKDPB66 | KOLAMOON 9 Pieces Detail Painting Brush Set Miniture Brushes for Watercolor, Acrylic Painting, Oil Painting (Wine Red) |
| Fish food | Amazon | Tetra Min Fish Food, Complete Food for All Tropical Fish for Health, Colour and Vitality, 10 L | |
| Formaldehyde Solution | Sigma Aldrich | F8775 | |
| Mouth Aspirator | John Hock | 612 | |
| Pond Salt | Amazon | Blagdon Guardian Pond Tonic Salt, for Fish Health, Water Quality, General Tonic, pH Buffer, 9.08 kg, treats 9,092 L | |
| Pupae Pots | Cater4you | SP8OZ | 250 pots with lids |
| Small Plastic Buckets | Amazon | 2.5 L White Plastic Pail Complete with White Lid (Pack of 10) | |
| Sodium Hypochlorite | Fisher Scientific | S25552 |
References
- Brand, A. H., Perimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Duffy, J. B. GAL4 system in drosophila: A fly geneticist’s swiss army knife. Journal of Genetics and Development. 34 (1-2), 1-15 (2002).
- Dow, J. A. . ELS. , (2012).
- Edi, C. V., et al. CYP6 P450 Enzymes and ACE-1 Duplication Produce Extreme and Multiple Insecticide Resistance in the Malaria Mosquito Anopheles gambiae. PLoS Genetics. 10 (3), 1004236 (2014).
- Daborn, P. J., et al. Using Drosophila melanogaster to validate metabolism-based insecticide resistance from insect pests. Insect Biochemistry and Molecular Biology. 42 (12), 918-924 (2012).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. Genes Genomes Genetics. 7 (6), 1819-1832 (2017).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), (2014).
- Lynd, A., Lycett, G. J. Development of the Bi-Partite Gal4-UAS System in the African Malaria Mosquito, Anopheles gambiae. PLoS ONE. 7 (2), 31552 (2012).
- Lynd, A., Lycett, G. J. Optimization of the Gal4-UAS system in an Anopheles gambiae cell line. Insect Molecular Biology. 20 (5), 599-608 (2011).
- Adolfi, A., Pondeville, E., Lynd, A., Bourgouin, C., Lycett, G. J. Multi-tissue GAL4-mediated gene expression in all Anopheles gambiae life stages using an endogenous polyubiquitin promoter. Insect Biochemistry and Molecular Biology. 96, 1-9 (2018).
- Kokoza, V. A., Raikhel, A. A. Targeted gene expression in the transgenic Aedes aegypti using the binary Gal4-UAS system. Insect Biochemistry and Molecular Biology. 41, 637-644 (2011).
- O’Brochta, D. A., Pilitt, K. L., Harrell, R. A., Aluvihare, C., Alford, R. T. Gal4-based Enhancer-Trapping in the Malaria Mosquito Anopheles stephensi. Genes Genomes Genetics. 2, 21305-21315 (2012).
- Zhao, B., et al. Regulation of the Gut-Specific Carboxypeptidase: A Study Using the Binary Gal4/UAS System in the Mosquito Aedes Aegypti. Insect Biochemistry and Molecular Biology. 54, 1-10 (2014).
- Imamura, M., et al. Targeted Gene Expression Using the GAL4/UAS System in the Silkworm Bombyx mori. Génétique. 165 (3), 1329-1340 (2003).
- Lynd, A., et al. Development of a functional genetic tool for Anopheles gambiae oenocyte characterisation: application to cuticular hydrocarbon synthesis. bioRxiv. , (2019).
- Pondeville, E., et al. Hemocyte-targeted gene expression in the female malaria mosquito using the hemolectin promoter from Drosophila. Insect Biochemistry and Molecular Biology. 120, 103339 (2020).
- Adolfi, A., et al. Functional genetic validation of key genes conferring insecticide resistance in the major African malaria vector, Anopheles gambiae. Proceedings of the National Academy of Sciences of the United States of America. 116 (51), 25764-25772 (2019).
- Pondeville, E., et al. Efficient integrase-mediated site-specific germline transformation of Anopheles gambiae. Nature Protocols. 9 (7), 1698-1712 (2014).
- Horn, C., Schmid, B. G. M., Pogoda, F. S., Wimmer, E. A. Fluorescent transformation markers for insect transgenesis. Insect Biochemistry and Molecular Biology. 32, 1221-1235 (2002).
- Clements, A. . A. Biology of Mosquitoes, Volume 1: Development, Nutrition and Reproduction. 1, (1992).
- Trpiš, M. A new bleaching and decalcifying method for general use in zoology. Canadian Journal of Zoology. 48, 892-893 (1970).
- Kaiser, M. L., Duncan, F. D., Brooke, B. D. Embryonic Development and Rates of Metabolic Activity in Early and Late Hatching Eggs of the Major Malaria Vector Anopheles gambiae. PLoS ONE. 9 (12), 114381 (2014).
- Grigoraki, L., Grau-Bové, X., Yates, H. C., Lycett, G. J., Ranson, H. Isolation and transcriptomic analysis of Anopheles gambiae oenocytes enables the delineation of hydrocarbon biosynthesis. eLife. 9, 58019 (2020).
- Xiao, Y. -. H., Yin, M. -. H., Hou, L., Pei, Y. Direct amplification of intron-containing hairpin RNA construct from genomic DNA. BioTechniques. 41 (5), 548-552 (2006).
- Livak, K. J. Organization and Mapping of a Sequence on the Drosophila melanogaster X and Y Chromosomes That Is Transcribed during Spermatogenesis. Génétique. 107 (4), 611-634 (1984).
- MR4, CDC, NEI & beiResources. . The MR4 Methods in Anopheles Research Laboratory Manual. 5th Edition. , (2015).
- Sik Lee, Y., Carthew, R. W. Making a better RNAi vector for Drosophila: use of intron spacers. Methods. 30 (4), 322-329 (2003).
- Cha-aim, K., Hoshida, H., Fukunaga, T., Akada, R., Peccoud, J. . Gene Synthesis: Methods and Protocols. , 97-110 (2012).
- Cavener, D. R. Comparison of the consensus sequence flanking translational start sites in Drosophila and vertebrates. Nucleic Acids Research. 15 (4), 1353-1361 (1987).
- Wang, Y., Wang, F., Wang, R., Zhao, P., Xia, Q. 2A self-cleaving peptide-based multi-gene expression system in the silkworm Bombyx mori. Scientific Reports. 5, (2015).
- Galizi, R., et al. A synthetic sex ratio distortion system for the control of the human malaria mosquito. Nature Communications. 5, 3977 (2014).
- Kondo, S., et al. Neurochemical organisation of the Drosophila Brain Visualised by Endogenously Tagged Neurotransmitter Receptors. Cell Reports. 30 (1), 284-297 (2020).
- Lee, P. -. T., et al. A gene-specific T2A-GAL4 library for Drosophila. eLife. 7, 35574 (2018).
- Marois, E., et al. High-throughput sorting of mosquito larvae for laboratory studies and for future vector control interventions. Malaria Journal. 11, 302 (2012).
- Crawford, J. E., et al. Efficient production of male Wolbachia-infected Aedes aegypti mosquitoes enables large-scale suppression of wild populations. Nature Biotechnology. 38 (4), 482-492 (2020).
- Goltsev, Y., et al. Developmental and evolutionary basis for drought tolerance of the Anopheles gambiae embryo. Biologie du développement. 330 (2), 462-470 (2009).
- Rezende, G. L., et al. Embryonic desiccation resistance in Aedes aegypti: presumptive role of the chitinized Serosal Cuticle. BMC Developmental Biology. 8 (1), 82 (2008).
- Vargas, H. C. M., Farnesi, L. C., Martins, A. J., Valle, D., Rezende, G. L. Serosal cuticle formation and distinct degrees of desiccation resistance in embryos of the mosquito vectors Aedes aegypti, Anopheles aquasalis and Culex quinquefasciatus. Journal of Insect Physiology. 62, 54-60 (2014).
- Chang, C. -. H., et al. The non-canonical Notch signaling is essential for the control of fertility in Aedes aegypti. PLOS Neglected Tropical Diseases. 12 (3), 0006307 (2018).
- Clemons, A., Flannery, E., Kast, K., Severson, D., Duman-Scheel, M. Immunohistochemical Analysis of Protein Expression during Aedes aegypti Development. Spring Harbor Protocols. 10, 1-4 (2010).
- Juhn, J., James, A. A. Hybridization in situ of Salivary Glands, Ovaries and Embryos of Vector Mosquitoes. Journal of Visualized Experiments. , e3709 (2012).

