Una descripción cartográfica 3D de la célula mediante tomografía de rayos X cryo soft
Summary
Aquí, se presenta un protocolo que describe los pasos de preparación de la muestra y recolección de datos requeridos en la tomografía de rayos X criobla (SXT) para obtener imágenes de la ultraestructura de células crioconservadas enteras a una resolución de medio paso de 25 nm.
Abstract
Las técnicas de imagen son fundamentales para comprender la organización celular y la maquinaria en la investigación biológica y los campos relacionados. Entre estas técnicas, la tomografía criobla de rayos X (SXT) permite obtener imágenes de células crioconservadas enteras en el rango de energía de rayos X de la ventana de agua (284-543 eV), en la que las estructuras de carbono tienen una absorción intrínsecamente mayor que el agua, lo que permite la reconstrucción 3D del coeficiente de absorción lineal del material contenido en cada vóxel. La información estructural cuantitativa a nivel de celdas enteras de hasta 10 μm de espesor se puede lograr de esta manera, con un alto rendimiento y resolución espacial de hasta 25-30 nm de paso medio. Cryo-SXT ha demostrado ser relevante para la investigación biomédica actual, proporcionando información 3D sobre procesos de infección celular (virus, bacterias o parásitos), cambios morfológicos debidos a enfermedades (como enfermedades genéticas recesivas) y ayudándonos a comprender la acción de los medicamentos a nivel celular o localizando estructuras específicas en el entorno celular 3D. Además, al aprovechar la longitud de onda sintonizable en las instalaciones de sincrotrón, la espectromicroscopía o su contraparte 3D, la espectrotomografía, también se pueden utilizar para obtener imágenes y cuantificar elementos específicos en la célula, como el calcio en los procesos de biomineralización. Cryo-SXT proporciona información complementaria a otras técnicas de imagen biológica como la microscopía electrónica, la fluorescencia de rayos X o la fluorescencia de luz visible, y generalmente se usa como un método asociado para imágenes correlativas 2D o 3D en condiciones criogénicas para vincular la función, la ubicación y la morfología.
Introduction
Cryo-SXT puede desempeñar un papel central en la investigación de imágenes biológicas, ya que proporciona volúmenes de resolución media 3D (25-30 nm de medio paso) de células enteras hidratadas 1,2,3,4,5,6. En el rango de energía de la ventana de agua, entre los bordes K de absorción de carbono y oxígeno (4.4-2.3 nm), las estructuras celulares ricas en carbono absorben 10 veces más que el medio rico en oxígeno que las impregna y rodea. En este rango de energía, las células vitrificadas de hasta 10 μm de espesor se pueden obtener imágenes sin necesidad de seccionamiento o tinción, lo que lleva a proyecciones cuantitativas de alto contraste de absorción, que, combinadas con las capacidades de rotación de la muestra, permiten la reconstrucción tomográfica de la estructura celular. Cryo-SXT llena un nicho en términos de dimensiones de muestras y resolución espacial que no es fácilmente accesible por ninguna otra técnica de imagen.
En resumen, el contraste de absorción de crio-SXT es cuantitativo, ya que la atenuación de los fotones a través de la muestra de espesor t obedece a la ley de Beer-Lambert de la siguiente manera: 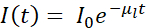 , donde I0 representa la intensidad incidente y μl el coeficiente de absorción lineal, que depende de la longitud de onda λ y la parte imaginaria β del índice de refracción de la muestra (
, donde I0 representa la intensidad incidente y μl el coeficiente de absorción lineal, que depende de la longitud de onda λ y la parte imaginaria β del índice de refracción de la muestra (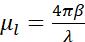 ). La atenuación es una función de la composición bioquímica y el grosor de las estructuras que se están fotografiando, y cada componente bioquímico tiene un coeficiente de absorción lineal de rayos X específico μl (LAC). Esto significa que cada valor de vóxel de tomografía depende de los elementos químicos y su concentración en ese vóxel7. Esto permite la discriminación natural de diferentes orgánulos como núcleos, nucleolos, cuerpos lipídicos o mitocondrias, o diferentes estados de compactación de la cromatina basados únicamente en sus valores inherentes de LAC reconstruidos 2,8,9.
). La atenuación es una función de la composición bioquímica y el grosor de las estructuras que se están fotografiando, y cada componente bioquímico tiene un coeficiente de absorción lineal de rayos X específico μl (LAC). Esto significa que cada valor de vóxel de tomografía depende de los elementos químicos y su concentración en ese vóxel7. Esto permite la discriminación natural de diferentes orgánulos como núcleos, nucleolos, cuerpos lipídicos o mitocondrias, o diferentes estados de compactación de la cromatina basados únicamente en sus valores inherentes de LAC reconstruidos 2,8,9.
Además, cryo-SXT es una técnica de alto rendimiento con tomogramas que se recolectan en pocos minutos. Esto permite específicamente la obtención de imágenes a mesoescala de poblaciones celulares que se pueden capturar en puntos de tiempo clave como la división, la diferenciación y la apoptosis, pero también en diferentes estados de respuesta, como los inducidos por la exposición química a terapias farmacológicas específicas o a infecciones patógenas. Los datos recopilados en esos puntos clave entregarán una descripción en 3D del sistema con un registro fiel de la organización espacial de los diferentes orgánulos celulares en esos momentos específicos.
Por lo general, el crio-SXT se utiliza en combinación con otras técnicas siguiendo enfoques correlativos que permiten localizar características, eventos o macromoléculas específicas dentro del entorno celular 3D 4,10,11,12,13,14,15,16, o datos de fluorescencia de rayos X duros17,18 . Los enfoques correlativos en condiciones criogénicas son de suma importancia para obtener la imagen más completa y valiosa del sistema de interés. En la Figura 1 se esboza un resumen sucinto del flujo de trabajo típico en las líneas de haz crio-SXT Mistral (Alba) y B24 (Diamond).
Además, aprovechando la capacidad de ajuste de longitud de onda en las instalaciones de sincrotrón, se puede obtener información espectroscópica además de la estructural utilizando la absorción diferencial específica de elementos particulares contenidos en la muestra. Un ejemplo de ello sería la localización del calcio en el estudio de los procesos de biomineralización en las células 19,20,21. Al tomar imágenes 2D a diferentes energías de fotones (espectros) o tomogramas debajo y en el borde de absorción de rayos X de interés, se pueden identificar los píxeles o vóxeles que contienen el elemento seleccionado. Spectra también permite diferenciar estados químicos (es decir, la evolución del calcio amorfo a hidroxiapatita como en el ejemplo de biomineralización anterior20). La cuantificación de diferentes elementos es posible en 2D y 3D. Las imágenes espectroscópicas de células vitrificadas se realizan típicamente en la ventana de agua, pero también son posibles en otros rangos de energía si el contenido de agua es lo suficientemente bajo o si se utilizan otros protocolos de preparación de muestras, incluida la deshidratación,22. Un protocolo detallado de espectroscopia paso a paso está más allá del enfoque del protocolo en este documento.
En lo que sigue, el protocolo se centra en resumir brevemente los principales pasos de preparación de la muestra, aunque cada sistema puede necesitar un refinamiento individual, seguido de un procedimiento detallado de recopilación de datos paso a paso para la tomografía de rayos X crioblables.
Protocol
Representative Results
Discussion
La preparación de muestras es un paso crítico para obtener tomogramas de rayos X blandos de alta calidad, ya que su calidad depende directamente de la calidad de la vitrificación de la muestra y del espesor de la capa de hielo en la que está incrustada la célula. Las proyecciones con alta relación señal-ruido se recogerán en regiones con capa de hielo delgada, lo que permitirá minimizar la dosis de radiación requerida para lograr la mayor resolución posible. Además, la confluencia celular también afectará la calidad del tomograma final, ya que se debe evitar que las células vecinas entren en el FoV al rotar. Finalmente, la dispersión correcta de los marcadores Au fiduciales determinará la precisión de la alineación de proyección y luego determinará en última instancia la calidad del volumen final reconstruido en 3D. Tenga en cuenta que una distribución adecuada de Au fiducials en la cuadrícula permite la automatización del paso de alineación de proyección, sin el cual se necesita una gran experiencia para un paso tan crítico.
El protocolo aquí presente solo representa una posible estrategia de preparación de muestras, que tiene similitudes con las utilizadas en la criotomografía electrónica (crio-ET). En ambos casos, los protocolos que mejoren la exigente preparación de la muestra para una mejor reproducibilidad serán fundamentales para el éxito de estas técnicas, y se están realizando esfuerzos hacia este objetivo29. Vale la pena mencionar que, además de obtener imágenes de células aisladas, también se pueden visualizar secciones de tejido siempre que la señal de transmisión a través de la sección sea suficiente en ángulos de inclinación altos. Por lo general, esto implicará secciones de pocas micras (por debajo de 10 μm).
Para obtener una imagen de una estructura o evento específico dentro de una celda, uno debe asegurarse de que esta característica en particular esté dentro del FoV de la serie de inclinación. Como el FoV en crio-SXT está limitado a 10 x 10 μm2 a 15 x 15 μm2 dependiendo de la lente y teniendo en cuenta un sobremuestreo de píxeles de la resolución a al menos un factor de 2, a menudo es más pequeño que la extensión completa de la celda (ver los cuadrados rojos indicados en la Figura 5). Por lo tanto, el ROI debe encontrarse y etiquetarse adecuadamente. Esto generalmente se hace por medio de etiquetas fluorescentes y enfoques correlativos de luz visible. Las estrategias 2D que combinan la epifluorescencia son sencillas, ya que el microscopio de transmisión de rayos X blando tiene un microscopio de fluorescencia de luz visible en línea integrado, pero otros enfoques para la señal de fluorescencia 2D o 3D de alta resolución también están disponibles 4,12,13,15,16 . En esos casos, la cuadrícula debe ser fotografiada primero en instrumentos específicos, como microscopios de súper resolución. Tenga en cuenta que los enfoques correlativos más eficientes son aquellos que involucran la recopilación de datos en condiciones criogénicas. Esto se debe a que el lapso de tiempo entre la temperatura ambiente (RT), las imágenes de fluorescencia de luz visible y la vitrificación de muestras, por ejemplo, dificultará la captura del evento celular correcto a tiempo; además, el procedimiento de vitrificación podría separar la celda de interés que se ha fotografiado en RT de la cuadrícula. Incluso si la mayoría de los enfoques de imágenes correlativas podrían implicar que las rejillas de muestra tienen que ser manipuladas y transportadas de un instrumento a otro, y a pesar del mayor riesgo de contaminación o daño de la red que esto plantea, la recompensa es clara: poder identificar eventos o moléculas específicas dentro del paisaje celular.
Cuando se requieren imágenes de células enteras, es posible coser diferentes tomogramas siempre que la dosis total aplicada no exceda el límite de daño por radiación. Por lo general, la dosis depositada para recolectar pocos tomogramas en la misma célula está muy por debajo del límite a la resolución alcanzable (109 Gy) y, por lo tanto, no se requiere una estrategia específica para reducir la dosis, aunque esto depende de la muestra y del experimento. En el caso de la recopilación intensiva de datos, como la espectrotomografía, sería necesario reducir al mínimo la dosis y sería necesario aplicar estrategias convenientes de recopilación de datos y procesamiento específicos.
Cryo-SXT tiene varias limitaciones, que deben mencionarse aquí. La primera es la conocida cuña faltante, que es intrínseca al uso de soportes de muestra plana. Los soportes de muestra capilar que permiten una rotación de 180 grados se han utilizado en el pasado y todavía se utilizan en algunas instalaciones, pero también presentan inconvenientes como un contraste empobrecido debido a la absorción de vidrio y la restricción del uso de células en suspensión. Una forma de disminuir el efecto de la cuña faltante es mediante la realización de una tomografía de doble inclinación. Esto es posible en la línea de haz Mistral hoy en día. La segunda limitación es establecida por la lente de placa de la zona de Fresnel utilizada en tales microscopios. Esta lente establece la resolución final alcanzable y la profundidad de campo (DoF), ambas estrechamente relacionadas. Esto implica que aumentar la resolución disminuirá el DoF, mientras que el grosor de la célula a menudo será mayor. Por ejemplo, una lente de 40 nm tendrá en teoría un DoF de 3 μm y una resolución de 24,4 nm de medio paso. El compromiso entre resolución y DoF es, por lo tanto, estratégico y la elección de la lente dependerá del tipo de celda30,31. Finalmente, los TXM operativos en todo el mundo están lejos de ser microscopios ideales y se están haciendo esfuerzos para mejorar los sistemas ópticos para alcanzar las expectativas teóricas. Finalmente, la visualización y segmentación de los volúmenes reconstruidos se puede llevar a cabo con herramientas de software específicas 25,32,33,34.
En resumen, crio-SXT permite obtener imágenes de células cuantitativamente a resolución media (25-30 nm de medio paso) y en números estadísticos (pocas decenas de tomogramas por día). Esto permite obtener la organización, distribución y dimensión de los orgánulos en condiciones específicas, por ejemplo, durante infecciones o enfermedades patógenas, en puntos de tiempo precisos o después de tratamientos particulares. Es, por lo tanto, una técnica de imagen biológica complementaria útil para las microscopías de electrones y luz visible más comunes, cada una de ellas abordando un rango específico de dimensiones y resolución de la muestra. Cryo-SXT se utiliza con frecuencia en enfoques correlativos que involucran fluorescencia de luz visible, pero también son posibles otras estrategias criocorrectivas.
Divulgations
The authors have nothing to disclose.
Acknowledgements
Este proyecto ha recibido financiación del proyecto iNEXT-Discovery Horizonte 2020 de la Comisión Europea y del programa de investigación e innovación Horizonte 2020 de la Unión Europea en virtud del acuerdo de subvención Marie Skłodowska-Curie n.º 75439.
Materials
| Amira | Thermo Fisher | Software for segmentation | |
| Au Holey Carbon Films finder grids | (Quantifoil Micro Tools Gmb | R 2/2 Au G200F1 | Au Holey Carbon Films finder grids |
| Au nanoparticles | BBI Group, Cardiff, UK | Au nanoparticles 100nm | 100 nm Au nanoparticles (NPs) at Mistral (Alba) |
| Au nanoparticles | BBI Group, Cardiff, UK | Au nanoparticles 250nm | 250 nm Au nanoparticles (NPs) at B24 (Diamond) |
| Axio Scope A1 | Zeiss | 430035 9060 | Fluorescence microscope |
| Blotting No.1 filter paper | Whatman | WHA10010155 | Blotting filter |
| Bsoft | Software for projection alignment, reconstruction and visualization (Heymann et al., 2008) | ||
| Chimera | Software for segmentation (Pettersen et al. 2004) | ||
| Cryo-EM Glow Discharge Set | PELCO easiGlow | 91000S | Glow Discharge Cleaning System |
| Cu Holey Carbon Films finder grids | (Quantifoil Micro Tools Gmb | R 2/2Cu G200F1 | Cu Holey Carbon Films finder grids |
| Fetal calf serum | Sigma | F9665 | Heat Inactivated, sterile-filtered, suitable for cell culture |
| ImageJ | Software for image processing and analysis in Java (NIH & LOCI University of Wisconsin) | ||
| IMOD | Software for projection alignment, reconstruction and visualization (Kremer et al., 1996) | ||
| Leica EM GP Grid Plunger | Leica | 16706401 | Automatic Plunge Freezer EM |
| LINKAM cryo-stage | Linkam Scientific Instruments | CMS 196 | Cryo-Correlative Microscopy Stage |
| MIB | Software for segmentation (Belevich et al. 2016) | ||
| P100 Petri dish | Sigma | P6106 | Treated for cell culture and sterile |
| P60 Petri dish | Sigma | D8054 | Treated for cell culture and sterile |
| Polylysin | Sigma | P4707 | Poly-L-lysine solution 0.01%, sterile-filtered |
| Soft X-Ray microscope 0.25-1.2keV | Xradia | NCT-SB | Transmission soft X-Ray microscope |
| SURVOS | Software for segmentation (Luengo et al. 2017) | ||
| Tomo3d | Software for reconstruction (SIRT, WBP) (Agulleiro et al. 2011) | ||
| TomoJ | Software for reconstruction (ART) (Messaoudi et al., 2007) | ||
| XM Data Explorer | Zeiss | TXM software |
References
- Carrascosa, J. L., et al. Cryo X-ray tomography of vaccinia virus membranes and inner compartments. Journal of Structural Biology. 168, 234-239 (2009).
- Uchida, M., et al. Soft X-ray tomography of phenotypic switching and the cellular response to antifungal peptoids in Candida albicans. PNAS. 106, 19375-19380 (2009).
- Schneider, G., et al. Three-dimensional cellular ultrastructure resolved by X-ray microscopy. Nature Methods. 10, 1-3 (2010).
- Chichón, F. J., et al. Cryo nano-tomography of vaccinia virus infected cells. Journal of Structural Biology. 177, 202-211 (2012).
- Groen, J., Conesa, J. J., Valcárcel, R., Pereiro, E. The cellular landscape by soft X-ray tomography. Biophysical Reviews. 11, 611-619 (2019).
- Kepsutlu, B., et al. Cells undergo major changes in the quantity of cytoplasmic organelles after uptake of gold nanoparticles with biologically relevant surface coatings. ACS Nano. 14, 2248-2264 (2020).
- Natterer, F. . The mathematics of computerized tomography. , (1986).
- Weiss, D., et al. Computed tomography of cryogenic biological specimens based on X-ray microscopic images. Ultramicroscopy. 84, 185-197 (2000).
- Clowney, E. J., et al. Nuclear aggregation of olfactory receptor genes governs their monogenic expression. Cell. 151, 724-737 (2012).
- Duke, E. M. H., et al. Imaging endosomes and autophagosomes in whole mammalian cells using correlative cryo-fluorescence and cryo-soft X-ray microscopy (cryo-CLXM). Ultramicroscopy. 143, 77-87 (2014).
- Cinquin, B., et al. Putting molecules in their place. Journal of Cellular Biochemistry. 115, 209-216 (2014).
- Hagen, C., et al. Multimodal nanoparticles as alignment and correlation markers in fluorescence/soft X-ray cryo-microscopy/tomography of nucleoplasmic reticulum and apoptosis in mammalian cells. Ultramicroscopy. 146, 46-54 (2014).
- Pérez-Berná, A. J., et al. Structural changes in cells images by soft X-ray cryo-tomography during Hepatitis C virus infection. ACS Nano. 10, 6597-6611 (2016).
- Chiappi, M., et al. Cryo-soft X-ray tomography as a quantitative three-dimensional tool to model nanoparticle:cell interaction. Journal of Nanobiotechnology. 14, 15 (2016).
- Varsano, N., et al. Development of correlative cryo-soft X-ray tomography and stochastic reconstruction microscopy. A study of cholesterol crystal early formation in cells. Journal of the American Chemical Society. 138, 14931-14940 (2016).
- Kounatidis, I., et al. Correlative cryo-structured illumination fluorescence microscopy and soft X-ray tomography elucidates reovirus intracellular release pathway. Cell. 182, 1-16 (2020).
- Kapishnikov, S., et al. Mode of action of quinoline antimalarial drugs in red blood cells infected by Plasmodium falciparum revealed in vivo. PNAS. 116 (46), 22946-22952 (2019).
- Conesa, J. J., et al. Unambiguous intracellular localization and quantification of a potent iridium anticancer compound by correlative 3D cryo X-ray imaging. Angewandte Chemie International Edition. 59, 1270-1278 (2020).
- Gal, A., et al. Native-state imaging of calcifying and noncalcifying microalgae reveals similarities in their calcium storage organelles. PNAS. 115 (43), 11000-11005 (2018).
- Procopio, A., et al. Chemical fingerprint of Zn-hydroxyapatite in the early stages of osteogenic differentiation. ACS Central Science. 5, 1449-1460 (2019).
- Kahil, K., et al. Cellular pathways of calcium transport and concentration toward mineral formation in sea urchin larvae. PNAS. 117 (49), 30957-30965 (2020).
- Conesa, J. J., et al. Intracellular nanoparticles mass quantification by near-edge absorption soft X-ray nanotomography. Scientific Reports. 6, 22354 (2016).
- Otón, J., Sorzano, C. O. S., Maribini, R., Pereiro, E., Carazo, J. M. Measurement of the modulation transfer function of an X-ray microscope based on multiple Fourier orders analysis of a Siemens star. Optics Express. 23 (8), 9567-9572 (2015).
- Otón, J., et al. Characterization of transfer function, resolution and depth of field of a soft X-ray microscope applied to tomography enhancement by Wiener deconvolution. Biomedical Optics Express. 7, 5092-5103 (2016).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Heymann, J. B., Cardone, G., Winkler, D. C., Steven, A. C. Computational resources for cryo-electron tomography in Bsoft. Journal of Structural Biology. 161, 232-242 (2008).
- Messaoudi, C., et al. Three-dimensional chemical mapping by EFTEM-TomoJ including improvement of SNR by PCA and ART reconstruction of volume by noise supression. Microscopy Microanalysis. 19, 1669-1677 (2013).
- Agulleiro, J. I., Fernández, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27, 582-583 (2011).
- Toro-Nahuelpan, M., et al. Tailoring cryo-electron microscopy grids by photo-micropatterning for in-cell structural studies. Nature Methods. 17, 50-54 (2020).
- Attwood, D. . Soft X-rays and Extreme Ultraviolet Radiation. Principles and Applications. , (2000).
- Howells, M., Jacobsen, C., Warwick, T. . Principles and Applications of Zone Plate X-ray. Microscopes. Science of Microscopy. , (2007).
- Pettersen, E. F., et al. UCSF Chimera–a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25, 1605-1612 (2004).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy image browser: a platform for segmentation and analysis of multidimensional datasets. PLOS Biology. 14 (1), 1002340 (2016).
- Luengo, I., et al. SuRVoS: Super-region volume segmentation workbench. Journal of Structural Biology. 198, 43-53 (2017).

