颗粒模板化乳化可实现无微流体液滴检测
Summary
油包水液滴测定可用于分析化学、酶进化和单细胞分析,但通常需要微流体才能形成液滴。在这里,我们描述了颗粒模板化乳化,这是一种进行液滴测定的无微流体方法。
Abstract
与散装液滴相比,在单分散液滴中进行的反应具有更高的准确性和灵敏度。然而,微流体形成受控液滴的要求对非专家施加了障碍,限制了它们的使用。在这里,我们描述了颗粒模板化乳化,这是一种在没有微流体的情况下产生单分散液滴的方法。使用模板化水凝胶球,我们通过简单的涡旋将样品封装在单分散的液滴中。我们通过使用它来执行无微流体数字PCR来演示该方法。
Introduction
与批量反应相比,液滴微流体利用皮升液滴中的区室化来提高测定的灵敏度和准确性,并且在化学筛选、蛋白质工程和下一代测序中具有许多应用1,2,3。例如,与本体定量聚合酶链反应 (qPCR) 相比,数字液滴聚合酶链反应 (ddPCR) 具有更高的准确性,可用于癌症的遗传变异、致病突变检测和产前诊断4,5,6。然而,液滴微流体的挑战是需要微流体装置来分割样品;虽然微流体对液滴特性具有出色的控制能力,但它们需要专业知识来构建和操作7,8。因此,基于液滴的方法主要限于专家实验室,或者在极少数情况下,可用于商业仪器的应用9,10。为了扩大液滴检测的使用范围,对专用微流体仪器的要求是必须克服的障碍。
在本文中,我们描述了颗粒模板化乳化(PTE),这是一种无微流体的方法,用于在单分散液滴中进行反应。在PTE中,模板颗粒通过简单的涡旋将样品吞没到载体油中的液滴中(图1)。随着系统的混合,水部分碎片成尺寸减小的液滴,直到液滴包含单个颗粒,此时不可能进一步破碎,因为它需要破坏颗粒。被吞没的样品将颗粒作为液滴中的壳包围,从而封装任何分散的细胞,试剂或功能部分(图1D)。因此,PTE不需要任何设备或专业知识来执行普通涡旋器以外的液滴反应。此外,与微流体的几分钟或几小时相比,液滴的产生需要几秒钟,并且产生的量与容器体积成正比,而不是设备操作时间,使其具有极高的可扩展性。这些优点使PTE非常适合在微流体不切实际的各种情况下进行液滴测定。在这里,我们演示了PTE并使用它来进行ddPCR。
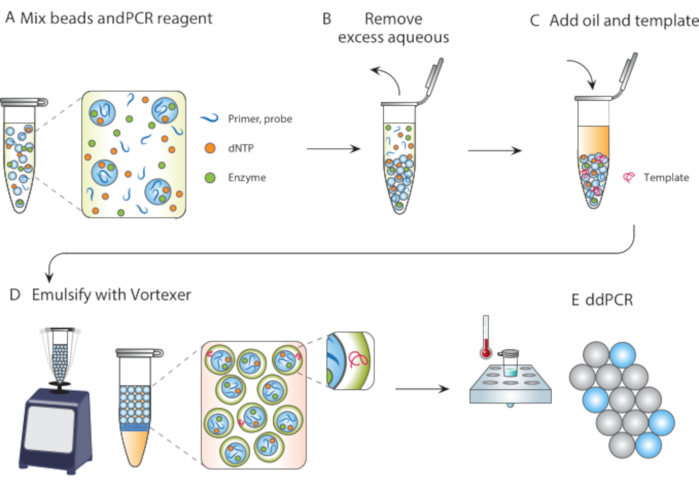
图 1.颗粒模板化乳化工艺概述。 (A)模板颗粒与试剂混合。(B)离心后除去多余的试剂。(C)模板分子的添加发生在添加油之前。(D)涡旋产生含有单个模板分子的液滴。(E)随后的热循环和成像允许对目标模板进行数字液滴分析。 请点击此处查看此图的放大版本。
Protocol
Representative Results
Discussion
PTE使用颗粒通过涡旋将样品封装在单分散的液滴中。除了简单性和可访问性之外,PTE还具有其他几个优点,包括允许立即产生大量液滴。此外,该过程可以在隔离管中进行,无需将样品转移到微流体设备,简化了整体工作流程并限制了样品污染或丢失的机会。模板颗粒还提供了一种设计所得液滴反应内容物的方法。例如,可以设计用于靶向生物分子或细胞捕获的粒径,化学和润湿性,而功能部分(如酶,活性物质或核酸)可以显示在颗粒上以促进反应,例如用于单细胞测序或功能表征。虽然这种方法是灵活的,但其使用仍然存在重要的限制。例如,目前不可能像通常使用微流体一样进行液滴添加,要求在封装之前引入所有反应组分;这要求试剂具有相容性和稳定性,直到可以产生液滴,并且在麻烦的组合的情况下,通常可以通过在冰上快速混合和乳化样品来解决。或者,可以使用可在外部用光或热触发的反应元件13。因此,PTE为进行非专家可访问的液滴测定提供了一种灵活且可扩展的方法。这一点,再加上其与生俱来的简单性和灵活性,使PTE成为执行和开发众多液滴应用的理想选择。
Divulgations
The authors have nothing to disclose.
Acknowledgements
开发该协议的工作得到了美国国立卫生研究院(R01-EB019453-02),国家情报总监办公室的支持,通过雷神BBN Technologies Corp(N66001-18-C-4507)进行的情报高级研究项目活动,Chan-Zuckerberg Biohub研究员计划,德克萨斯A&M大学的国防高级研究计划局(W911NF1920013)以及约翰霍普金斯大学应用疾病控制和预防中心的支持 物理实验室 (75D30-11-9C-06818 (CDC3)).此处包含的观点和结论是作者的观点和结论,不应被解释为必然代表上述组织或美国政府的明示或暗示的官方政策。美国政府有权出于政府目的复制和分发重印本,尽管其中有任何版权说明。
Materials
| 0.22 um syringe filter | Milipore Sigma | SLGP033RS | |
| 0.5M EDTA, pH 8.0 | Thermo-Fisher | 15575020 | |
| 0.75 mm biopsy punch | World Precision Instruments | 504529 | |
| 1 mL syringes | BD | 309628 | |
| 1H,1H,2H-Perfluoro-1-Octanol (PFO) | Sigma-Aldrich | 370533 | |
| 1M Tris-HCI, pH 8.0 | Thermo-Fisher | 15568025 | |
| 27 gauge needles | BD | 305109 | |
| 3" silicon wafers, P type, virgin test grade | University Wafers | 447 | |
| 3D-printed centrifuge syringe holder | (custom) | (custom) | |
| Acrylamide solution,40%, for electrophoresis, sterile-filtered | Sigma-Aldrich | A4058-100ML | |
| Ammonium persulfate | Sigma-Aldrich | A3678-25G | |
| Aquapel (fluorinated surface treatment) | Pittsburgh Glass Works | 47100 | |
| Hexane | Sigma-Aldrich | 139386 | |
| FC-40 fluorinated oil | Sigma-Aldrich | F9755 | |
| Isopropanol | Sigma-Aldrich | 109827 | |
| N,N′-Methylenebis(acrylamide) | Sigma-Aldrich | 146072-100G | |
| NaCl | Sigma-Aldrich | S9888 | |
| Novec-7500 Engineering Fluid (HFE oil) | 3M | 98-0212-2928-5 | |
| polyethylene tubing | Scientific Commodities | B31695-PE/2 | |
| fluorosurfactant | Ran Biotechnologies | 008-FluoroSurfactant | |
| PGMEA developer | Sigma-Aldrich | 484431 | |
| Photomasks | CadArt Servcies | (custom) | |
| Platinum Multiplex PCR Master Mix (Taq Master Mix) | Applied Biosystems | 4464263 | |
| Spin coater | Specialty Coating Systems | G3P-8 | |
| Span 80 (sorbitane monooleate) | Sigma-Aldrich | s6760 | |
| SU-8 3025 photoresist | Kayaku | 17030192 | |
| Triton X-100 (octylphenol ethoxylate) | Sigma-Aldrich | t8787 | |
| Tween 20 (polysorbate 20) | Sigma-Aldrich | p2287 | |
| Platinum Multiplex PCR Master Mix (Taq Master Mix) | Applied Biosystems | 4464263 | |
| Yeast FWD | IDT | 5′-GCAGACCAGACCAGAACAAA-3′ | |
| Yeast REV | IDT | 5′-ACACGTATGTATCTAGCCGAATA AC-3 |
|
| Yeast Probe | IDT | 5′-/56-FAM/ATATGTTGT/ZEN/TCACTCGCGCCTGGG/3IABk FQ/-3′ |
|
| EVOS FL AUTO | Life Technologies | ||
| EVOS LED Cube, GFP | Life Technologies | AMEP4651 | |
| SYLGARD 184 KIT 1.1 LB (PDMS base and curing reagents) | Dow Corning | DC4019862 | |
| TEMED | Thermo Fisher | 17919 | |
| Saccharomyces cerevisiae genomic DNA | Milipore | 69240-3 | |
| Expanded plasma cleaner (plasma bonder) | Harrick Plasma | PDC-002 (230V) | |
References
- Mashaghi, S., Abbaspourrad, A., Weitz, D. A., van Oijen, A. M. Droplet microfluidics: A tool for biology, chemistry, and nanotechnology. Trends in Analytical Chemistry. 82, 118-125 (2016).
- Gielen, F., et al. Ultrahigh-throughput-directed enzyme evolution by absorbance-activated droplet sorting (AADS). Proceedings of the National Academy of Sciences of USA. 113 (47), 7383-7389 (2016).
- Mai, S., Murphy, T. W., Lu, C. Microfluidics for genome-wide studies involving next generation sequencing. Biomicrofluidics. 11 (2), 021501 (2017).
- Olmedillas-López, S., García-Arranz, M., García-Olmo, D. Current and emerging Applications of Droplet Digital PCR in Oncology. Molecular Diagnosis and Therapy. 21 (5), 493-510 (2017).
- Tong, Y., Shen, S., Jiang, H., Chen, Z. Application of Digital PCR in Detecting Human Diseases Associated Gene Mutation. Cellular Physiology and Biochemistry. 43 (3), 1718-1730 (2017).
- Yan, Y., et al. Evaluation of droplet digital PCR for non-invasive prenatal diagnosis of phenylketonuria. Analytical and Bioanalytical Chemistry. 411 (27), 7115-7126 (2019).
- Shang, L., Cheng, Y., Zhao, Y. Emerging Droplet Microfluidics. Chemical Reviews. 117 (12), 7964-8040 (2017).
- The, S., Lin, R., Hung, L., Lee, A. P. Droplet Microfluidics. Lab Chip. 8 (2), 198-220 (2008).
- Baker, M. Digital PCR hits its stride. Nature Methods. 9, 541-544 (2012).
- Kolodziejczyk, A. A., Kim, J. K., Svensson, V., Marioni, J. C., Teichmann, S. A. The Technology and Biology of Single-Cell RNA Sequencing. Molecular Cell. 58 (4), 610-620 (2015).
- Hatori, M. N., Kim, S. C., Abate, A. R. Particle-Templated Emulsification for Microfluidics-Free Digital Biology. Analytical Chemistry. 90 (16), 9813-9820 (2018).
- Panda, P., et al. Stop-flow lithography to generate cell-laden microgel particles. Lab Chip. 8 (7), 1056-1061 (2008).
- Yozwiak, C. E., Hirschhorn, T., Stockwell, B. R. Towards a microparticle-based system for pooled assays of small molecules in cellular contexts. ACS Chemical Biology. 13 (3), 761-771 (2018).

